Teknik PCR- - Fakultas Biologi
advertisement

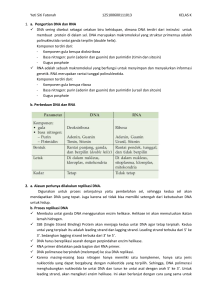
TEKMK PCR
oleh
Drs. Agus Hery Susanto, M.S.
staf pengajar Fakultas Biologi Unsoed
Pendahuluan
Teknik PCR (polymerase chain reaction) digunakan untuk
fragmen DNA (urutan basa nukleotida) tertentu secara
menggandakan
invitro melalui reaksi polimerisasi
DNA secara berulang-ulang selama beberapa siklus reaksi. Teknik yang ditemukan oleh
Kary Mullis pada tahun 1987 ini telah mengalami berbagai macam modifikasi dan
penggunaannya meliputi ruang lingkup yang begitu luas.
Komponen PCR
Tiap reaksi polimerisasi membutuhkan komponen-komponen sintesis DNA
seperti untai DNA yang akan digunakan sebagai cetakan (templat), molekul
oligonukleotida untai tunggal dengan ujung 3'-OH bebas yang berfungsi sebagai
prekursor (primer), sumber basa nukleotida berupa empat macam dNTP (dATP, dGTP,
dCTP, dTTP), dan enzim DNA polimerase.
DNA templat adalah DNA untai ganda yang membawa urutan basa fragmen atau
gen yang akan digandakan. Urutan basa
ini disebut juga urutan target (target
sequence).
Penggandaan urutan target pada dasarnya merupakan akumulasi hasil polimerisasi
molekul primer.
Primer adalatr molekul oligonukleotida untai tunggal yang terdiri atas sekitar 30
basa. Polimerisasi primer dapat berlangsung karena adanya penambahan basa demi basa
dari dNTP ypng dikatalisasi oleh enzim DNA polimerase. Namun, pada PCR enzim DNA
polimerase yang digunakan harus termostabil karena salah satu tahap reaksinya adalah
denaturasi untai ganda DNA yang membutuhkan suhu sangat tinggi (sekitar 95'C). Salah
satu enzim
DNA polimerase yang umum digunakan adalah Zaq DNA polimerase, yang
berasal dari bakteri termofilik Thermus aquaticus.
Fakultas Biologi Unsoed
Tahapan PCR
Tiap putaran reaksi PCR terdiri atas tiga tahap, yaitu denaturasi templat,
penempelan primer, dan polimerisasi primer, yong masing-masing berlangsung pada suhu
lebih kurang 95oC, 50oC, dan 70"C. Pada tahap denaturasi, pasangan untai DNA templat
dipisahkan satu sama lain sehingga menjadi untai tunggal. Pada tahap selanjutnya,
masing-masing untai tunggal akan ditempeli oleh primer. Jadi, ada dua buah primer yang
masing-masing menempel pada untai tunggal DNA templat. Biasanya, kedua primer
tersebut dinamakan primer maju (forword primer) dan primer mundur (reverse primer).
Setelah menempel pada untai
DNA templat, primer mengalami polimerisasi mulai dari
tempat penempelannya hingga ujung
5' DNA
templat (ingat polimerisasi DNA selalu
berjalan dari ujung 5' ke 3' etau berqrti dori ujung 3' ke 5' untai templatnya). Dengan
demikian, pada akhir putaran reaksi pertama akan diperoleh dua pasang untai DNA jika
DNA templat awalnya berupa sepasang untai DNA.
Pasangan-pasangan untai
DNA yang diperoleh pada suatu akhir putaran reaksi
akan menjadi templat pada putaran reaksi berikutnya. Begitu seterusnya hingga pada
putaran yang ke n diharapkan akan diperoleh fragmen DNA pendek sebanyak
2-
2n.
Fragmen DNA pendek yang dimaksudkan adalah fragmen yang ukurannya s:rma dengan
jarak antara kedua tempat penempelan primer. Fragmen pendek inilah yang merupakan
urutan target yang memang dikehendaki untuk digandakan (diamplifftasi).
Bisa dibayangkan seandainya PCR dilakukan dalam 20 putaran saj4 maka pada
akhir reaksi akan diperoleh fragmen urutan target sebanyak
224
-2.20:
1.048576
- 40:
1.048536 ! Jumlah ini masih dengan asumsi bahwa DNA templat awalnya hanya satu
untai ganda. Padahal kenyataanny4 hampir tidak mungkin DNA templat awal hanya
berupa satu untai ganda. Jika DNA templat awal terdiri atzs 20 untai ganda saj4 maka
jumlah tadi tinggal dikalikan 20 menjadi 20.970.720, suatu jumlah yang sangat cukup
bila akan digunakan sebagai fragmen pelacak.
Fakultas Biologi Unsoed
5',
DNA templat awal berupa DNA untai ganda
l***^i
(-ss"c)
primer
$rn"*o"tan
primer maju 5'
-'
at
-'t
ft
s
oil
3' +# primer mundur
I
poU.rrir^i primer (!70"C)
ti.
at
J.
J
-
.
5
Gambar 1. Putaran pertama PCR
Perancangan Primer
Tahapan PCR yang paling menentukan adalah penempelan primer. Sepasang
primer oligonukleotida (primer maju dan primer mundur) yang akan dipolimerisasi
masing-masing harus menempel pada sekuens target, tepatnya pada kedua ujung fragmen
yang akan diamplifikasi. Untuk itu urutan basanya harus komplementer atau setidak-
{
tidaknya memiliki homologi cukup tinggi dengan urutan basa kedua daerah ujung
fragmen yang akan diamplifikasi itu. Padahal, kita belum mengetahui dengan pasti urutan
basa sekuens target. Oleh karena
basa kedua
itu, diperlukan cara tertenfu untuk merancang urutan
primer yang akan digunakan.
Dasar yang digunakan adalah urutan basa yang diduga mempunyai kemiripan
ini adalahUnsoed
Fakultas Biologi
urutan serupa dari
dengan urutan basa sekuens target. Urutan
sejumlah
spesies/strain organisme lainnya yang telah diketahuildipublikasikan. Sebagai contoh,
untuk merancang sepasang primer yang diharapkan dapat mengamplifikasi sebagian gen
lipase pada isolat Bacillus termofilik tertentu dapat digunakan informasi urutan
basa gen
lipase dari strain-strain Pseudomonas fluorescens, P. mendocina
sebelumnya telah diketahui.
, dan sebagainya,
ymg
Urutan-urutan basa fragmen tertentu dari berbagai strain tersebut kemudian
dijajarkan dan dicari satu daerah atau lebih yang memperlihatkan homologi tinggi antara
satu strain dan lainnya. Daerah
ini
dinamakan daerah lestari (conserved area).
Sebagian/seluruh urutan basa pada daerah lestari inilah yang akan menjadi urutan
basa
primer.
Sebenarnya, daerah lestari juga dapat ditentukan melalui penjajaran urutan asam
amino pada tingkat protein. Urutan asam amino ini kemudian diturunkan ke urutan basa
DNA. Dari satu urutan asam amino sangat mungkin akan diperoleh lebih dari satu urutan
basa
DNA karena setiap
asam amino dapat disandi oleh lebih dari satu triplet kodon.
Dengan demikian, urutan basa primer yang disusun dapat merupakan kombinasi beberapa
kemungkinan- Primer dengan urutan basa semacam
ini dinamakan primer
degenerate.
selain itu, primer yang disusun melalui penjajaran urutan basa DNA pun
dapat
merupakan primer degenerate karena urutan basa pada daerah lestari di tingkat DNA pun
tidak selamanya memperlihatkan homologi sempurna
(
1
00%).
Urutan basa pasangan primer yang telah disusun kemudian dianalisis
menggunakan program komputer untuk mengetahui kemungkinan terjadinya primerdimer akibat homologi sendiri (setf-homalog,,) atau homologi silang (cross-homotogt).
Selain itu, juga perlu dilihat kemungkinan terjadinya salah tempel (mispriming), yaitu
penempelan primer di luar sekuens target. Analisis juga dilakukan untuk
mengetahui
titik
leleh (7L) qrasing-masing primer dan kandungan GC-nya, sepasang primer yang
baik
harus mempunyai TnIangrelatif sama dengan kandungan GC yang
cukup tinggi.
Fakultas Biologi Unsoed
5',
3'
5'
-t
5'
5'
3
Fgmen pendek
5',
yang diinginkan
3',
5'
.5',
3'
5'
5'
3' .
5'#
a)
J
5o Bg*"n pendek
y'angdiinginkan
a)
J
5',-
3'
Gambar 2. Hasil PCR putaran kedua dan ketiga
Penutup
Melalui teknik PCR dapat diperoleh sejumlah besar fragmen DNA tertentu dalam
<
waktu yang relatif singkat. Namun, keberhasilan amplifftasi dengan teknik PCR
ditentukan oleh berbagai faktor terutama kompatibilitas primer oligonukleotida yang
digunakan.
Daftar Pustaks
Alberts, 8., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P.2007. Molecular
Biology of the Cell 5tr Ed. Garland Science, New York.
Elliott, W.H. and Elliott, D.C.2001. Biochemistry and Molecular Biology 2d Ed. Oxford
University Press, Oxford.
Fakultas Biologi Unsoed
Lodish, H., Berk, A., Kaiser, c.A., Krieger, M., Scott, M,p., Bretscher, A., ploegh, H.,
and Matsudain.20a7. Molecular cell Biology 6th Ed. w.H. Freeman, New york.
watson, J.D., Baker, T.A., Bell, s.P., Gann, A.A.F., Levine, M., and Losick, R.M. 2007.
Molecular Biology ofthe Gene 6ft Ed. Benjamin cummings, New york.
Fakultas Biologi Unsoed