molecular identification using pcr (polymerase chain reaction)
advertisement

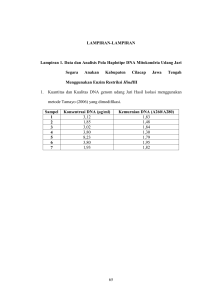
MOLECULAR IDENTIFICATION USING PCR (POLYMERASE CHAIN REACTION) AND DNA SEQUENCING OF STRAIN DETERMINATION PATHOGENIC BACTERIA CAUSE ACUTE RESPIRATORY INFECTION (Klebsiella pneumoniae) IDENTIFIKASI MOLEKULAR DENGAN TEKNIK PCR (POLYMERASE CHAIN REACTION) DAN SEKUENSING DNA TERHADAP PENENTUAN STRAIN BAKTERI PATOGEN PENYEBAB INFEKSI SALURAN PERNAPASAN AKUT (Klebsiella pneumoniae) Suraya Chairunisa, Priyo Wahyudi, Supandi Fakultas Farmasi dan Sains Universitas Muhammadiyah Prof. Dr. Hamka Abstract Klebsiella pneumoniae is a patogenic bacteria causing acute respiratory infection disease especially for pneumoniae. This research aims at determining the strain of the bacterium Klebsiella pneumoniae. Accurate identification in a short time for the bacteria Klebsiella pneumoniae by Polymerase Chain Reaction (PCR) technique. PCR is an enzymatic process that can double a particular nucleotide sequence in vitro. The process begining by isolating Klebsiella pneumoniae bacteria as a pathogenic bacteria by Gene Purification JET KIT methods. DNA isolates was amplified with primers 16S rRNA and processed through the stages of DNA sequencing. Sequencing process was evidenced by uploading results to Genbank sequences using the BLAST program. The results stated that the sequence DNA of Klebsiella pneumoniae isolated from Siloam Hospital Karawaci Tangerang-Banten have a percent similarity of 97% of the bacteria Klebsiella pneumoniae strain DSM 30104. Keyword : Klebsiella pneumoniae, DNA, PCR, DNA Sequencing Klebsiella pneumoniae adalah bakteri patogen penyebab penyakit ISPA khususnya pneumonia. Penelitian ini bertujuan untuk mengidentifikasi dan menentukan strain dari bakteri Klebsiella pneumoniae .Identifikasi yang akurat dengan waktu singkat untuk bakteri Klebsiella pneumoniae yaitu dengan teknik Polymerase Chain Reaction (PCR). PCR merupakan suatu proses enzimatik yang dapat melipatgandakan suatu sekuen nukleotida tertentu secara in vitro. Proses diawali dengan mengisolasi DNA genom bakteri Klebsiella pneumoniae sebagai bakteri patogenik dengan metode Gene JET Purification KIT. Isolat DNA diamplifikasi dengan primer 16S rRNA dan diproses melalui tahap sekuensing DNA. Proses sekuensing dibuktikan dengan mengupload hasil sekuens ke Genbank menggunakan program BLAST. Hasil menyatakan bahwa sekuens DNA Klebsiella pneumoniae dari isolat Rumah Sakit Siloam Karawaci Tangerang-Banten memiliki persen kemiripan sebesar 97% terhadap bakteri Klebsiella pneumoniae strain DSM 30104. Kata Kunci : Klebsiella pneumoniae, DNA, PCR, Sekuensing DNA 1 PENDAHULUAN Infeksi Saluran Pernapasan Akut (ISPA) merupakan infeksi yang terdapat pada saluran nafas atas maupun saluran nafas bagian bawah yang dapat berlanjut menjadi penyakit pneumonia. Klebsiella pneumoniae merupakan bakteri Gram negatif penyebab utama pneumonia. Di Indonesia, Klebsiella pneumoniae salah satu penyebab dari pneumonia komunitas dan pneumonia nosokomial. Tercatat sebesar 45.18% sebagai penyebab pneumonia komunitas pada tahun 1998-2003 dengan jumlah terbanyak. Sedangkan penyebab pneumonia nosokomial tercatat sekitar 19,51% pada tahun 2002 (PDPI 2003). Karena jumlah kasus yang telah tercatat cukup mengkhawatirkan, identifikasi yang akurat untuk bakteri Klebsiella pneumoniae menjadi peranan yang sangat penting. Metode identifikasi bakteri yang umum dilakukan adalah metode dengan teknik mikrobiologi. Seiring dengan perkembangan teknologi, proses pengidentifikasian bakteri dapat diupayakan lebih cepat dan lebih akurat dengan menggunakan teknik Polymerase Chain Reaction (PCR). PCR merupakan suatu proses sintesis enzimatik yang dapat melipatgandakan secara eksponensial suatu sekuen nukleotida tertentu secara in vitro (Yuwono 2006) dengan menggunakan sepasang primer oligonukleotida yang berperan sebagai inisiasi amplifikasi molekul DNA dalam waktu singkat. Gen penanda yang akan digunakan adalah Gen 16S rRNA. Penggunaan Gen 16S rRNA untuk mengidentifikasi suatu jenis isolat bakteri yang belum dikenali (Clarridge 2004). Identifikasi bakteri uji (Klebsiella pneumoniae) dilakukan dengan menggunakan sekuen Gen 16S rRNA untuk mempelajari hubungan filogenetik dari suatu spesies bakteri berdasarkan daerah conserved-nya (Willson et al. 2011). Penentukan analisis sekuen dilakukan dengan proses sekuensing DNA. Proses sekuensing DNA adalah proses penentuan urutan basa suatu DNA dengan prinsip reaksi polimerisasi DNA secara enzimatis. Hasil analisa sekuen gen tersebut menemukan persamaan secara jelas tentang kemungkinan bakteri uji tersebut bersifat spesifik terhadap spesies dari galur Klebsiella pneumoniae atau tidak. Penelitian ini bertujuan untuk mengidentifikasi bakteri penyebab ISPA berdasarkan hasil sekuen Gen 16S rRNA dari suatu isolat yang diperoleh dari Rumah Sakit Siloam Karawaci Tangerang-Banten. Penelitian ini diharapkan dapat menentukan strain bakteri Klebsiella pneumoniae. METODOLOGI Alat Penelitian Alat-alat yang digunakan adalah tabung reaksi, gelas ukur, erlenmeyer, mortar, stemper, batang pengaduk, spatel, timbangan analitik, vortex, micropipet, tube sentrifus 2 ml, jarum ose, inkubator, autoklaf, laminar air flow, water bath, microcentrifuge refrigerator (Bio-Lion XC-HR 20), elektroforesis (Mupid EXU), Peltier Thermo Cycler (Tanach RAY MG48), UV-transiluminator (Extra Gene). 2 Bahan Penelitian Isolat bakteri dari Rumah Sakit Siloam Karawaci Tangerang-Banten, Aquabides, Ethanol 50% dan 96%, ddH2O, dan GeneJET Genomic DNA Purification Kit (Thermo Scientific) yang terdiri dari lysis solution, digestion solution, proteinase K, RNAse solution, wash buffer I, wash buffer II dan elution buffer, Agarosa, Bufer TBE (Tris Bifosfat EDTA) 1X, Etidium Bromida, loading dye (Thermo Scientific), Gene Ruler 1 kb DNA ladder (Fermentas), Maxima Hot Star Green PCR Master Mix 2x (Thermo Scientific), berupa forward primer 63f (5’-CAG GCC TAA CAC ATG CAA GTC-3’) dan reverse primer 1387r (5’-GGG CGG WGT GTA CAA GGC-3’) Isolasi DNA Genom dengan GENEJet Kit Isolat bakteri terlebih dahulu dikultur dalam medium nutrient broth dan diinkubasi selama 24 jam pada suhu 37oC. Sebanyak 1,5-2,0 ml kultur bakteri dalam tabung mikrosentrifus steril disentrifus pada kecepatan 5000 x g selama 10 menit. Supernatan yang terbentuk dalam tabung dibuang lalu endapan sel bakteri ditambahkan dengan 20 μl larutan Proteinase K dan 180 μl digestion solution, lalu diinkubasi selama 30 menit pada suhu 56oC sambil sesekali dihomogenkan. Kemudian 20 μl RNAse solution ditambahkan ke dalam tabung, dihomogenkan dan diinkubasi suhu ruang selama 10 menit. Lalu 200 μl lysis solution ditambahkan dan dihomogenkan selama 15 menit. Selanjutnya ditambahkan 400 μl ethanol 50% sampai tercampur. Kemudian campuran tersebut dipindahkan ke kolom GeneJET Genomic DNA Purification yang dilengkapi dengan collection tube dan disentrifus pada kecepatan 6000 x g selama 1 menit. Kemudian larutan dalam collection tube dibuang, diganti dengan collection tube 2 ml yang baru untuk ditambahkan 500 μl wash buffer I yang telah dilarutkan dengan etanol 96% dan disentrifus pada kecepatan 8000 x g selama 1 menit. Larutan dalam collection tube dibuang, diganti dengan collection tube yang baru. Kemudian di tambahkan 500 μl wash buffer II yang sudah dilarutkan dengan etanol 96%, disentrifus dengan kecepatan 12000 x g selama 3 menit. Larutan dipindahkan kedalam kolom GeneJET Genomic DNA Purification ke tube mikrosentrifus steril 1,5 ml dan ditambahkan 200 μl elution buffer. Larutan diinkubasi selama 2 menit pada suhu ruang, dan disentrifus pada kecepatan 8000 x g selama 1 menit. Kolom purifikasi di buang, DNA disimpan dalam suhu 20oC (Thermo Scientific 2011). Analisis DNA Genom dengan Elektroforesis Gel agarosa 1,4% digunakan untuk elektroforesis DNA genom dilakukan dengan voltase 100 volt selama 30 menit. Agarosa 1,4% dibuat dengan cara mendidihkannya sebanyak 0,7 g dalam 50 ml aquabides steril sampai larut. Setelah larut, agarosa didiamkan sampai agak dingin. Setelah itu larutan dituang dalam cetakan gel, sisir dipasangkan dan didiamkan hingga membeku. Kemudian sisir di angkat dan nampan dipindahkan ke wadah elektroforesis yang terlebih dahulu diberikan larutan bufer TBE 1x hingga menggenangi permukaan agarosa. Hasil isolasi DNA sampel sebanyak 2 μl dicampurkan dengan 10 μl loading dye 6x. Larutan dicampurkan secara pipetting kemudian dimasukkan ke dalam sumur gel sebanyak 10 μl. Sebanyak campuran 2 μl DNA ladder, 2 μl loading dye 6x, dan 8 μl 3 ddH2O dimasukkan ke sumur gel yang berbeda. Gel kemudian direndam dalam larutan etidium bromida selama 15 menit dalam ruangan yang gelap. DNA kemudian divisualisasikan di UV-transluminator. Amplifikasi DNA Target 16S rRNA dengan PCR Sejumlah 25 μl Maxima Hot Star Green PCR Master Mix 2x (Thermo Scientific), 3 μl hasil isolasi DNA, 2,5 μl primer forward dan reverse dan 17 μl ddH2O ditambahkan dalam mikrotube 0,2 ml. Larutan kemudian divortex dan di spin down. Sepasang primer sebanyak 2 μl ditambahkan ke dalam larutan lalu dihomogenkan. Larutan dipipet sebanyak 22 μl dan dimasukkan ke dalam microtube. Kemudian ditambahkan 3 μl template DNA, dihomogenkan dan spin down kembali lalu dimasukkan ke dalam PCR. Proses PCR diawali dengan proses denaturasi awal pada suhu 94 oC selama 3 menit untuk memisahkan untai DNA secara sempurna. Selanjutnya diikuti dengan 30 siklus yang terdiri atas denaturasi 94oC selama 1 menit, annealing 55oC selama 1 menit, ekstensi 72oC selama 10 menit dan ekstensi akhir dilakukan pada suhu 72oC selama 10 menit. Amplikon dilakukan elektroforesis selama 30 menit pada gel agarosa 1,4 % dengan tegangan 100 volt. Pada sumur yang berbeda, dimasukkan DNA ladder 1 kb dalam aquabides lalu divisualisasikan di UV-transluminator. (Thermo Scientific 2011). Sekuensing Gen 16S rRNA Amplikon dimasukkan ke dalam microtube 0,5 ml kering dan steril. Untuk mencegah kebocoran dan perembesan, microtube diberi label dan disegel dengan parafilm. Sampel dikirim ke First BASE Laboratories, Malaysia untuk selanjutnya dilakukan purifikasi dan sekuensing DNA. Sekuen DNA yang diperoleh kemudian dibandingkan dan dikarakterisasi dengan sekuen database National Centre of Biotecnologi Information (NCBI) pada situs http://www.ncbi.nlm.nih.gov dengan program BLAST. Kemudian dipilih sekuen Gen 16S rRNA dari bakteri yang muncul pada database setelah diperoleh kemiripannya dengan bakteri lain. Analisa Data Data hasil deteksi PCR dengan elektroforesis dianalisis berdasarkan ada tidaknya potongan pita DNA yang terbentuk, dan data yang ditampilkan dalam bentuk deskriptif berisi tabel dan gambar. Hasil dan Pembahasan Proses isolasi DNA dilakukan sesuai dengan protokol yang terdapat pada GeneJET Genomic DNA Purification Kit untuk bakteri Gram negatif karena bakteri patogen hasil isolasi merupakan bakteri patogen jenis Gram negatif. Tahap penting dalam isolasi DNA adalah proses perusakan atau penghancuran membran dan dinding sel. Pemecahan sel (lisis) bertujuan untuk mengeluarkan isi sel (Holme et al. 1998). Sentrifus dilakukan untuk memisahkan medium dengan bakteri dan menjamin agar medium tidak ikut terbawa. Penambahan digestion solution dan proteinase K akan membuat sel lisis, sehingga memungkinkan hancurnya struktur protein dan pelepasan 4 asam nukleat dari inti sel. Komponen RNA juga harus dihilangkan dengan penambahan RNAase solution, sehingga DNA dapat diisolasi secara utuh. Pemurnian atau purifikasi DNA dapat dilakukan dengan penambahan lysis solution. Larutan wash buffer I dan wash buffer II digunakan untuk menjaga struktur DNA selama proses penghancuran dan purifikasi sehingga memudahkan dalam menghilangkan protein dan RNA serta mencegah perubahan pada molekul DNA. Elution buffer digunakan untuk menghasilkan DNA dengan tingkat kemurnian yang tinggi dan menjadikan DNA terelusi dengan sempurna. Setelah didapat DNA murni hasil isolasi, DNA dipisahkan menggunakan elektroforesis gel agarose 1,4% dalam bufer TBE 1X dengan voltase 100 volt selama 30 menit. TBE 1x berfungsi sebagai media penghantaran listrik, karena mengandung elektrolit berupa ion-ion yang dapat meningkatkan konduktifitas sehingga laju migrasi DNA akan lebih mudah. Loading dye mengandung xylene cyanol dan bromophenol blue berfungsi sebagai pewarna DNA sehingga mempermudah visualisasi pada saat elektroforesis. Pemisahan ditandai dengan DNA ladder 1 kb yang memiliki rentang pemisahan dari 250 bp hingga 10000 bp. Hasil elektroforesis (elektroforegram) direndam dalam etidium bromida dan dibilas dalam aquabides. Proses pembilasan dilakukan dalam waktu 5 menit untuk menghindari kerusakan DNA akibat pengaruh lingkungan. Elektroforegram divisualisasikan di atas UV transluminator untuk melihat fragmen DNA. X Gambar 1. Elektroforegram isolat DNA Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten. M: DNA ladder 1 kb, K6: Isolat DNA, X: Pita DNA Klebsiella pneumoniae K6 Gambar 1 memperlihatkan visualisasi dengan UV transiluminator yang menunjukan pita DNA dengan ukuran sekitar 250 pb. Ukuran pita DNA sangat kecil dan terlihat smear, karena pengaruh mekanis saat proses isolasi yang kurang hati hati dan teliti dan penyimpanan yang terlalu lama sehingga kemungkinan berkurangnya jumlah komponen murni DNA bisa terjadi. Proses selanjutnya adalah amplifikasi isolat DNA menggunakan metode Polymerase Chain Reaction (PCR) dengan menggunakan forward primer 63f (5’CAG GCC TAA CAC ATG CAA GTC-3’) dan reverse primer 1387r (5’-GGG CGG WGT GTA CAA GGC-3’). Primer ini banyak digunakan untuk mengamplifikasi gen 5 16S rRNA dari bakteri secara umum (Marchesi et al. 1998). Proses PCR dalam penelitian ini menggunakan PCR master mix 2x yang mengandung taq polymerase DNA, bufer, MgCl2, dan dNTP. PCR master mix 2x adalah larutan dengan dua kali jumlah konsentrasi dari semua komponen yang dibutuhkan dalam PCR, kecuali primer dan DNA template. PCR master mix 2x memiliki keuntungan yaitu dapat mengurangi kontaminasi karena meminimalisir proses pippeting dan menghemat waktu karena dapat langsung digunakan tanpa harus melakukan proses pencampuran. Proses amplifikasi dilakukan menggunakan PCR dengan denaturasi awal pada suhu 94°C selama 3 menit untuk mempersiapkan untai DNA yang akan didenaturasi. Denaturasi pada suhu 94°C selama 1 menit bertujuan untuk memisahkan untai ganda DNA. Annealing pada suhu 55°C selama 1 menit untuk memberi waktu pada primer menempel pada daerah target tertentu dari target DNA. Ekstensi pada suhu 72oC selama 5 menit dilakukan dengan tujuan untuk memperpanjang primer. Ekstensi akhir pada suhu 72 oC yang bertujuan untuk menyempurnakan proses penggabungan untai DNA. Proses denaturasi, annealing, dan ekstensi dilakukan sebanyak 30 siklus bertujuan untuk melipatgandakan amplikon. Amplifikasi DNA dilakukan menggunakan Maxima Hot Start Green PCR Master mix (2x) dari Thermo Scientific untuk mempermudah pengerjaan dan ketepatan jumlah komponen PCR yang ditambahkan. Semua proses penambahan komponen PCR dilakukan pada suhu dingin karena enzim DNA polimerase pada master mix akan memulai aktivitasnya pada suhu dingin. Analisis amplikon hasil PCR dipisahkan menggunakan elektroforesis gel agarosa 1,4% pada tegangan 100 volt selama 30 menit dan divisualisasikan di atas UV transluminator. X Gambar 2. Elektroforegram Amplikon Gen 16S rRNA bakteri Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten. M: DNA ladder 1 kb, K6: Hasil amplifikasi isolat DNA, X: Pita amplikon DNA Klebsiella pneumoniae K6 Gambar 2 memperlihatkan visualisasi dengan UV translluminator menunjukan pita DNA dengan ukuran sekitar 4000 pb. Meskipun pita-pita DNA yang dihasilkan kurang baik, tetapi DNA hasil isolasi dapat teramplifikasi dengan baik, 6 sehingga didapatkan produk DNA target yang cukup untuk proses selanjutnya. Hal ini dapat disimpulkan bahwa pada proses amplifikasi dengan PCR tidak harus dibutuhkan DNA dengan kuantitas yang benar-benar murni, karena keuntungan PCR salah satunya adalah dapat mengamplifikasi DNA target hanya dalam jumlah yang relatif kecil dan PCR dapat dikatakan memiliki tingkat sensifitas yang tinggi terhadap DNA genom. Proses selanjutnya adalah proses Sekuensing DNA. Analisa hasil sekuensing dilakukan dengan meng-input data hasil sekuens (query) pada program resmi GenBank. Sebelum meng-input data ke GenBank, data hasil sekuen dilakukan analisa terlebih dahulu untuk melihat bentuk elektroferogram yang didapat. Gambar 3. Elektroforegram Sekuen Gen 16S rRNA dari bakteri Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten dengan Primer Reverse 1387r Gambar 4. Elektroforegram Sekuen Gen 16S rRNA bakteri Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten dengan Primer Forward 63f Gambar 3 dan Gambar 4 menunjukkan hasil analisa sekuens yang terdiri dari puncak dan urutan basa nitrogen. Hasil puncak menunjukan data yang kurang baik, karena puncak yang dihasilkan saling berhimpitan satu sama lain. Puncak yang saling berhimpitan ini bisa terjadi akibat penempelan primer yang kurang sempurna pada saat proses amplifikasi, sehingga akan muncul puncak yang saling bertumpuk saat dianalisa dengan mesin sekuenser. Sekuen yang telah diperoleh dianalisis dengan data yang serupa dengan data yang telah dipublikasikan sebelumnya di GenBank menggunakan program situs online yaitu situs http://www.ncbi.nlm.gov. Program yang digunakan untuk menentukan similaritas antar sekuens DNA adalah BLAST dan ClustalW. Di dalam 7 kedua program tersebut terdapat algoritme yang dapat berfungsi mengukur kemiripan antar sekuens DNA yang diperbandingkan. Perbedaannya, ClustalW mengukur kemiripan di antara beberapa input sekuens, sedangkan BLAST membandingkan satu input sekuens dengan semua sekuens yang ditemukan dalam database. Pengukuran kemiripan menggunakan ClustalW dapat digunakan untuk membuat pohon filogenetik yang menggambarkan hubungan evolusi antar spesies, sedangkan BLAST mengukur semua kemiripan tanpa melihat hubungan evolusi. Gambar 5. Hasil Uji Spesifitas Sekuen Gen 16S rRNA bakteri Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten terhadap Primer 63f dan 1387r Dengan BLASTN Dari gambar 5 menunjukan hasil BLAST dengan NCBI didapatkan data berupa garis-garis bewarna merah. Hasil ini menunjukkan tingkat kemiripan berdasarkan suatu skala pada sekuens-sekuens yang telah disejajarkan. Warna merah menunjukkan bahwa data hasil BLAST valid, karena jika datanya kurang bagus akan ditunjukkan dengan warna biru sampai hitam. Hasil yang didapat menunjukkan garisgaris bewarna merah yang berarti kedua sekuens memiliki tingkat kemiripan yang sangat mirip yaitu lebih dari 200 nukleotida 8 Gambar 6. Hasil Uji Spesifitas Sekuen Gen 16S rRNA bakteri Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten dengan BLASTN Gambar 6 merupakan kelanjutan dari hasil BLASTN, dari data tersebut didapatkan hasil bahwa sampel isolat memiliki persen kemiripan 97% pada jenis bakteri Klebsiella pneumoniae strain DSM 30104. Nilai nol pada E-value menunjukkan sekuens homolog dengan sekuens yang terdapat di GenBank. Dari hasil identifikasi yang dilakukan secara molekular, didapatkan hasil yang sama dengan identifikasi secara mikrobiologi. Namun penelitian secara molekular memiliki hasil yang lebih luas karena bisa terlihat sampai pada tingkatan strain bakteri tersebut sedangkan untuk identifikasi secara mikrobiologi hanya sampai pada pembuktian suatu spesies bakterinya saja. 9 Gambar 7. Sekuens Gen 16S rRNA bakteri Klebsiella pneumoniae strain DSM 30104 dengan BLASTN Dari gambar 7 menunjukan bahwa terdapat garis-garis penghubung antara sekuens yang berada di atas dengan sekuens yang berada di bawah. Garis ini menunjukkan adanya kesesuaian antara kedua sekuens dengan tingkat kesesuaian sebesar 97%. Sedangkan bagian-bagian yang tak terhubung pada garis (gaps) menunjukkan adanya perbedaan dari kedua sekuens tersebut pada saat proses penyejajaran. Data sekuen Gen 16S rRNA dari sampel isolat kemudian dibandingkan dengan beberapa data sekuen Gen 16S rRNA dari beberapa spesies lainnya. Hasil perbandingan sekuen ini kemudian akan divisualisasikan dalam pohon filogenetik yang dapat menunjukkan hubungan kekerabatan antara satu galur dengan galur yang lainnya. 10 Gambar 8. Pohon Filogenetik Sekuen Gen 16S rRNA bakteri Klebsiella pneumoniae K6 dari pasien ISPA Rumah Sakit Siloam Karawaci Tangerang-Banten dengan Blast Tree View Pohon filogenetik selain menunjukkan kekerabatan antar spesies yang diperbandingkan, juga menggambarkan perubahan yang terjadi pada gen penanda untuk masing-masing spesies. Gambar 8 merupakan hasil representasi dari sampel. Dari pohon filogenetik diketahui bahwa sampel yang memiliki persentase kemiripan 97% dengan Klebsiella pneumoniae strain DSM 30104 memiliki hubungan yang dekat dengan Klebsiella variicola strain At-22. SIMPULAN Berdasarkan penelitian yang telah dilakukan dengan penyejajaran Gen 16S rRNA menggunakan program NCBI, didapatkan hasil akhir yang menunjukkan bahwa sekuens DNA Klebsiella pneumoniae K6 dari isolat bakeri Rumah Sakit Siloam Karawaci Tangerang-Banten memiliki persen kemiripan sebesar 97% terhadap Klebsiella pneumoniae strain DSM 30104. DAFTAR PUSTAKA Clarridge JE. 2004. Impact of 16S rRNA Gene Sequence Analysis for Identification of Bacteria on Clinical Microbiology and Infectious Diseases. Journal Clinical Microbiology Reviews. Vol 17 Hlm. 840-862. Edvotek. 2003. Principles and Practice of Agarose Gel Electrophoresis. The Biotechnology Education Company. http://www.edvotek.com. diakses 19 feb 2014. 11 PDPI (Perhimpunan Dokter Paru Indonesia). 2003. Pneumonia Komuniti: Pedoman Diagnosis & Penatalaksanaan di Indonesia. PDPI. Jakarta. Hlm. 8. Promega corporation. 2010. Wizard Genomic DNA Purification Kit. USA. Thermo Scientific. 2011. Genomic DNA Purification Protocols. Thermo Fisher Scientific Inc. Thermo Scientific. 2011. Protocol Maxima Hot Start Green PCR Master Mix (2x). Thermo Fisher Scientific Inc. Wilson BA, Salyers AA, Whitt DD, Winkler ME. 2011. Bacterial Pathogenesis Third Edition. ASM Press, Washington DC. Hlm. 12, 80-83. Yuwono T. 2006. Teori Dan Aplikasi Polymerase Chain Reaction. Andy Publisher, Yogyakarta. Hlm. 1-23, 23-278, 217-218. 12