hasil - IPB Repository
advertisement
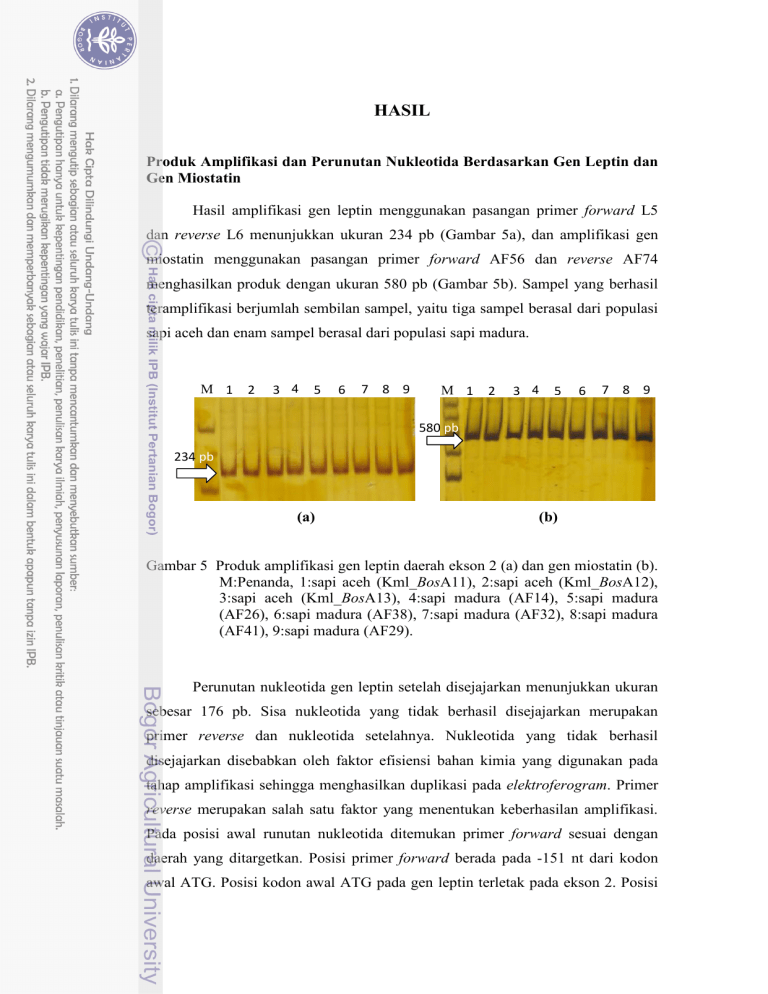
HASIL Produk Amplifikasi dan Perunutan Nukleotida Berdasarkan Gen Leptin dan Gen Miostatin Hasil amplifikasi gen leptin menggunakan pasangan primer forward L5 dan reverse L6 menunjukkan ukuran 234 pb (Gambar 5a), dan amplifikasi gen miostatin menggunakan pasangan primer forward AF56 dan reverse AF74 menghasilkan produk dengan ukuran 580 pb (Gambar 5b). Sampel yang berhasil teramplifikasi berjumlah sembilan sampel, yaitu tiga sampel berasal dari populasi sapi aceh dan enam sampel berasal dari populasi sapi madura. M 1 2 3 4 5 6 7 8 9 M 1 2 3 4 5 6 7 8 9 580 pb 234 pb (a) (b) Gambar 5 Produk amplifikasi gen leptin daerah ekson 2 (a) dan gen miostatin (b). M:Penanda, 1:sapi aceh (Kml_BosA11), 2:sapi aceh (Kml_BosA12), 3:sapi aceh (Kml_BosA13), 4:sapi madura (AF14), 5:sapi madura (AF26), 6:sapi madura (AF38), 7:sapi madura (AF32), 8:sapi madura (AF41), 9:sapi madura (AF29). Perunutan nukleotida gen leptin setelah disejajarkan menunjukkan ukuran sebesar 176 pb. Sisa nukleotida yang tidak berhasil disejajarkan merupakan primer reverse dan nukleotida setelahnya. Nukleotida yang tidak berhasil disejajarkan disebabkan oleh faktor efisiensi bahan kimia yang digunakan pada tahap amplifikasi sehingga menghasilkan duplikasi pada elektroferogram. Primer reverse merupakan salah satu faktor yang menentukan keberhasilan amplifikasi. Pada posisi awal runutan nukleotida ditemukan primer forward sesuai dengan daerah yang ditargetkan. Posisi primer forward berada pada -151 nt dari kodon awal ATG. Posisi kodon awal ATG pada gen leptin terletak pada ekson 2. Posisi kodon awal pada penelitian ini ditemukan sesuai dengan posisi pada urutan referensi dari data GenBank, yaitu B. indicus haplotipe GCATC dengan no asesi FJ626855.1 (Gambar 6). Perunutan nukleotida gen miostatin setelah disejajarkan dengan urutan referensi dari GenBank menunjukkan ukuran sebesar 523 pb. Sisa nukleotida dari hasil amplifikasi merupakan primer forward sebesar 23 pb dan nukleotida setelahnya sebesar 38 pb. Gambar 6 Posisi kodon awal (ATG) pada daerah ekson 2 gen leptin berdasarkan B. indicus (No. akses FJ626855.1). Haplotipe Gen Leptin pada Sapi Aceh dan Sapi Madura Urutan nukleotida gen leptin sepanjang 176 pb yang disejajarkan dengan urutan referensi dari data GenBank menunjukkan polimorfisme. Polimorfisme tersebut membedakan sapi aceh dan sapi madura dengan B. indicus haplotipe ATGCT, B. indicus haplotipe GCATC, B. taurus, B. frontalis, dan sapi hibridisasi B. indicus x B. taurus dari data GenBank. Secara umum gen leptin terdiri atas dua haplotipe, yaitu haplotipe ATGCT dan haplotipe GCATC. Perbedaan haplotipe pada gen leptin didasarkan pada lima titik mutasi. Pada penelitian ini perbedaan haplotipe hanya ditemukan pada tiga titik mutasi, yaitu pada posisi 32 nt, 87 nt, dan 89 nt (Lampiran 1). Perbandingan urutan nukleotida antara sapi aceh dengan B. indicus haplotipe ATGCT menunjukkan empat perbedaan, tiga perbedaan di antaranya membedakan antara sapi aceh dengan B. indicus haplotipe ATGCT. Sapi aceh mengikuti pola haplotipe GCATC (Gambar 7a). Pada hasil perunutan nukleotida pada sapi madura menunjukkan perbedaan haplotipe antar sampel. Sapi madura mempunyai dua haplotipe, yaitu haplotipe ATGCT dan haplotipe GCATC. Urutan nukleotida sampel sapi madura (AF14) mengikuti pola haplotipe ATGCT, sedangkan runutan nukleotida sampel sapi madura lainnya mengikuti pola haplotipe GCATC (Gambar 7b). (a) (b) Gambar 7 Posisi tiga titik mutasi pada penelitian ini menunjukkan perbedaan haplotipe: (a) Haplotipe GCATC pada sapi aceh, (b) Haplotipe ATGCT dan haplotipe GCATC pada sapi madura. Keragaman Gen Leptin dan Perubahan Asam Amino Pada gen leptin daerah ekson 2 antara sapi aceh dan sapi madura dengan B. indicus haplotipe ATGCT, B. indicus haplotipe GCATC, B. taurus, dan sapi hibridisasi B. indicus x B. taurus dari data GenBank ditemukan 12 varian. Tiga varian membedakan antara sapi aceh, sapi madura, dan runutan referensi dari data GenBank berdasarkan haplotipe, delapan polimorfisme hanya dimiliki oleh sapi hibridisasi antara B. indicus dengan B. taurus, dan satu polimorfisme ditemukan pada dua sampel sapi aceh dan satu sampel sapi madura (Gambar 8). Perubahan satu basa nukleotida pada dua sampel sapi aceh dan satu sampel sapi madura ditemukan pada posisi - 91 nt dari kodon awal ATG. Pada posisi tersebut dua sampel sapi aceh dan satu sampel sapi madura mempunyai basa T, sedangkan satu sampel sapi aceh, lima sampel sapi madura, dan populasi lainnya dari data GenBank mempunyai basa C (Gambar 8). Tiga basa nukleotida yang membedakan antara haplotipe ATGCT dengan haplotipe GCATC ditemukan pada posisi -63 nt, -65 nt, dan -120 nt. Pada posisi -63 nt mengalami perubahan basa A menjadi basa G. Pada posisi -65 nt mengalami perubahan basa T menjadi basa C. Pada posisi -120 nt ditemukan perubahan basa G menjadi basa A. Mutasi yang terjadi pada posisi sebelum kodon awal ATG tidak ditranskripsikan ke dalam asam amino. Gambar 8 Keragaman gen leptin daerah ekson 2 pada sapi aceh dan sapi madura Keragaman Gen Miostatin pada Sapi Aceh dan Sapi Madura Keragaman gen miostatin pada ruas promotor setelah diurutkan antara sapi aceh dan sapi madura dengan B. taurus dari data GenBank menunjukkan 10 perbedaan. Tiga perbedaan ditemukan pada sapi aceh dan sapi madura, yaitu perubahan basa T→C pada posisi 494 nt, C→T pada posisi 508 nt, dan A→G pada posisi 575 nt. Tujuh perbedaan ditemukan pada sapi madura, sedangkan urutan nukleotida pada sapi aceh mempunyai kesamaan dengan B. taurus. Mutasi yang membedakan antara sapi madura dengan sapi aceh dan B. taurus adalah perubahan basa T menjadi basa C pada posisi 499 nt. Selain itu, enam mutasi lainnya tidak hanya membedakan antara sapi madura dengan sapi aceh tetapi juga membedakan antar sampel sapi madura, yaitu perubahan basa C→T pada posisi 350 nt, perubahan basa G→A pada posisi 423 nt, perubahan basa A→G pada posisi 456 nt, perubahan basa C→G pada posisi 459 nt, perubahan basa A→G pada posisi 490 nt, dan satu delesi basa T ditemukan pada sampel sapi madura pada posisi 560 nt (Gambar 9). Gambar 9 Keragaman gen miostatin daerah promotor pada sapi aceh dan sapi madura Analisis Filogeni Berdasarkan Gen Leptin Rekonstruksi pohon filogeni gen leptin berdasarkan persamaan dan perbedaan haplotipe. Gen leptin mempunyai dua kelompok haplotipe secara umum, yaitu haplotipe ATGCT dan haplotipe GCATC. Haplotipe dibedakan berdasarkan lima titik mutasi. Pada penelitian ini perbedaan haplotipe sepanjang nukleotida yang telah diurutkan hanya ditemukan tiga titik mutasi. Pada pohon filogeni sapi aceh berada pada percabangan haplotipe GCATC, sedangkan sapi madura mempunyai dua kelompok percabangan, yaitu percabangan haplotipe AGCAT dan haplotipe ATGCT (Gambar 10). Urutan haplotipe sapi aceh mengikuti pola GCATC sehingga pada pohon filogeni sapi aceh berada pada satu kelompok dengan B. indicus haplotipe GCATC dari data GenBank (No. Asesi FJ626855.1). Sapi madura mempunyai pola haplotipe ATGCT berada pada satu kelompok dengan B. indicus haplotipe ATGCT (No. Asesi FJ626856.1), sedangkan sapi madura mempunyai pola haplotipe GCATC berada pada kelompok B. indicus haplotipe GCATC (No. Asesi FJ626855.1). Percabangan antara sapi madura dengan B. indicus haplotipe ATGCT menunjukkan nilai bootstrap sebesar 89% berdasarkan nukleotida. Nilai bootstrap di atas (≥50%) menunjukkan kekuatan percabangan (Robust) yang mendukung pengelompokan sapi madura dengan B. indicus haplotipe ATGCT (No. Asesi FJ626856.1). Selain sapi madura, jenis sapi lainnya yang mempunyai kemiripan urutan nukleotida dengan B. indicus haplotipe ATGCT adalah B. frontalis (EU642566.1), tetapi titik mutasi posisi -64 nt pada B. frontalis berbeda dengan B. indicus haplotipe ATGCT, sehingga pada percabangan filogeni B. frontalis menunjukkan sistergroup dari kelompok sapi madura dan B. indicus haplotipe ATGCT. Gambar 10 Rekonstruksi pohon filogeni gen leptin berdasarkan nukleotida menggunakan metode Neighbour-Joining (NJ), model Kimura-2Parameter, dan butsrap pengulangan 1000x. Topologi pohon filogeni berdasarkan gen leptin tidak menunjukkan percabangan yang berbeda antara kelompok sapi aceh dan sapi madura dengan kelompok Bos taurus. Pola haplotipe B. taurus GCATC berada pada percabangan yang sama dengan sapi aceh dan sapi madura haplotipe GCATC. Sampel sapi aceh (Kml_BosA11 dan Kml_BosA13) dan sapi madura (AF38) menunjukkan percabangan khusus di dalam kelompok B. indicus haplotipe GCATC pada pohon filogeni. Percabangan tersebut menunjukkan nilai bootstrap sebesar 63%. Pengelompokan tersebut membentuk percabangan tersendiri mempunyai perbedaan pada satu nukleotida. Pengelompokan sapi aceh dan sapi madura dengan sapi hibridisasi antara B. indicus dengan B. taurus tidak pada satu percabangan yang sama. Sapi B. indicus X B. taurus mempunyai kesamaan haplotipe dengan B. indicus haplotipe GCATC (No. Asesi FJ626855.1), tetapi sapi hibridisasi tersebut mengalami mutasi di delapan posisi nukleotida. Perbedaan posisi nukleotida antara sapi hibrid dengan sapi aceh dan sapi madura haplotipe GCATC menyebabkan percabangan pada pohon filogeni terpisah antara kelompok sapi aceh dan sapi madura haplotipe GCATC dengan kelompok sapi B. indicus X B. taurus. Analisis Filogeni Berdasarkan Gen Miostatin Topologi pohon filogeni berdasarkan gen miostatin memisahkan sapi aceh dengan sapi madura (Gambar 11). Pemisahan kelompok sapi aceh dengan sapi madura disebabkan oleh tujuh variasi nukleotida. Kelompok sapi aceh berada pada satu percabangan dengan B. taurus dari data GenBank (No. akses AF348479.1). Pada pohon filogeni percabangan sapi madura antar sampel yang digunakan menunjukkan perbedaan pengelompokan. Sampel sapi madura (AF38, AF14, AF26, dan AF41) mengelompok pada percabangan yang sama. Cabang pohon filogeni pada kelompok sapi madura (AF38, AF14, AF26, dan AF41) didukung dengan nilai bootstrap sebesar 94%. Sapi madura (AF29) dan sapi madura (AF32) terpisah dari percabangan pertama. Perbedaan cabang antara sapi madura (AF38, AF14, AF26, dan AF41) dengan sapi madura (AF29) dan sapi madura (AF32) disebabkan tiga variasi nukleotida. Percabangan sapi madura (AF32) terpisah dari kelompok sapi madura lainnya. Perbedaan cabang antara sapi madura (AF32) terpisah dengan sapi madura lainnya disebabkan adanya dua variasi nukleotida. Pengelompokan sapi madura tersebut didukung dengan nilai bootstrap sebesar 72%. Pada penelitian ini Ovis aries (DQ530260.1) digunakan sebagai outgroup untuk membedakan cabang pengelompokan sapi aceh dengan sapi madura. Ovis aries mempunyai karakter nukleotida gen Leptin yang sangat berbeda dengan sapi aceh dengan sapi madura, sehingga percabangan O. aries pada pohon filogeni tidak masuk ke dalam ingrup. Percabangan O. aries yang berbeda dari ingroup (Sapi aceh dan sapi madura) disebabkan adanya 40 variasi nukleotida. Gambar 11 Rekonstruksi pohon filogeni gen miostatin menggunakan metode Neighbour-Joining (NJ) berdasarkan model Kimura-2-Parameter dengan butsrap pengulangan 1000x.










