Desain Primer Multiplex Polymerase Chain Reaction (PCR) Gen E6
advertisement

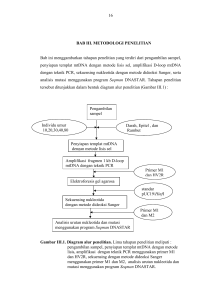
P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 Desain Primer Multiplex Polymerase Chain Reaction (PCR) Gen E6 HPV Tipe 45 dan HPV Tipe 52 (Primer Design of Multiplex Polymerase Chain Reaction (PCR) Gen E6 HPV Tipe 45 and HPV Tipe 52) Marlina1; Prima Ramadhani1; Andani Eka Putra2; Rustini1; Yufri Aldi1 1Faculty of Pharmacy, Andalas University, Post Code 25163, Padang, Indonesia 2 Faculty of Medicine, Andalas University, Post Code 25163, Padang, Indonesia. *Corresponding email: [email protected] ABSTRAK Human Papillomavirus (HPV) merupakan penyebab utama kanker serviks. Terdapat lebih dari 200 tipe HPV. Penentuan tipe HPV dilakukan menggunakan primer spesifik untuk masing-masing tipe. Saat ini primer untuk HPV tipe 45 dan HPV tipe 52 masih jarang dipublikasikan. Penelitian ini desain sebuah primer multiplex untuk mendeteksi HPV tipe 45 dan HPV tipe 52. Primer didesain menggunakan software AmplifX 1.5.4, Oligo Analyzer serta beberapa situs resmi seperti NCBI dan primer3plus. Keseluruhan hasil yang diperoleh dibandingkan dengan kriteria desain primer sehingga diperoleh primer yang terbaik. Primer multiplex gen E6 yang diperoleh memiliki panjang amplikon 129 bp untuk HPV tipe 45 dan 402 bp untuk HPV tipe 52. Kata Kunci: Kanker serviks, desain primer, HPV tipe 45, HPV tipe 52, Multiplex PCR PENDAHULUAN Polymerase chain reaction (PCR) adalah Bioinformatika teknik suatu teknik sintesis dan amplifikasi DNA yang komputasi untuk mengelola dan menganalisis dapat menjawab tantangan ini. Primer adalah informasi metode salah satu unsur penting, merupakan deretan fisika, protein yang akan melipatgandakan DNA target. biologi, dan ilmu kedokteran yang saling Primer berfungsi sebagai pembatas fragmen DNA menunjang satu sama lain (Utama, 2003). Seiring target yang akan diamplifikasi dan sekaligus berkembangnya bioteknologi molekuler saat ini, menyediakan gugus hidroksi (-OH) pada ujung 3’ bioinformatika menjadi karena yang diperlukan untuk proses eksistensi DNA perkembangan teknologi dan (Handoyo & Rudiretna, 2001; Haswan, dkk., biologis, matematika, merupakan mencakup statistika, informatika, penting informasi peningkatan ilmu komputer membuka sudut 2012). pandang baru dalam menyeleaikan berbagai Kanker serviks merupakan suatu tumor persoalan biologi molekuler, terutama dengan ganas yang diawali dengan pertumbuhan sel menggunakan sequence DNA dan asam amino yang tidak normal yang terjadi pada permukaan (Baxevanis & Ouelette, 2005). epitel leher rahim atau mulut rahim (Sarwono, 2008; Manuaba, 2009). Kanker serviks 158 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 menduduki peringkat ketiga diseluruh dunia dan kedua daerah ujung yang diamplifikasikan (Aris, peringkat kedua di Indonesia sebagai penyakit dkk., 2013). yang menyebabkan kematian pada perempuan (Bruni, et al., 2014). Penyebab utama kanker METODE PENELITIAN serviks adalah Human papillomavirus (HPV) Pencarian isolat primer (WHO, 2010). Virus ini dapat ditularkan melalui hubungan seksual. Berdasarkan Langkah awal dalam pembuatan primer tingkat adalah mencari isolat primer yang akan di desain. keparahannya dalam menyebabkan infeksi, virus Isolat primer HPV tipe 45 dan HPV tipe 52 ini dapat diklasifikasikan menjadi dua tipe yaitu didapat HPV tipe high risk dan low risk. HPV tipe high risk (http://www.ncbi.nlm.nih.gov), antara lain tipe 16, 18, 31, 33, 34, 35, 39, 45, 51, untuk isolat reference HPV tipe 45 dan HPV tipe 52, 56, 58, 59, 66, 68, dan 82. HPV tipe inilah yang 52 seringkali menyebabkan terjadinya kanker, (http://www.hpvcenter.se). dari diambil database dari situs NCBI sedangkan HPV center terutama kanker serviks (WHO, 2010). HPV tipe 16, 18, 31 dan 33 merupakan Analisis penjajaran primer HPV peringkat teratas sebagai penyebab kanker Analisis penjajaran primer dilakukan serviks. Studi di Indonesia menunjukan 76.9% terhadap isolat primer HPV tipe 45 dan HPV tipe kanker serviks invasif dikaitkan dengan HPV tipe 52 terhadap masing-masing isolat reference 16 dan 18 (Bruni, et al., 2014). Pada penelitian menggunakan program BLAST yang dapat Marlina (2014) telah didesain primer HPV tipe diakses 16, 18, 31 dan 33 untuk mendeteksi kanker (http://www.ncbi.nlm.nih.gov/BLAST). Analisis serviks HPV tipe 16, 18, 31 dan 33. Tidak hanya penjajaran ini dilakukan untuk mengetahui HPV tipe 16, 18, 31 dan 33, namun HPV tipe 45 posisi basa yang tidak mengalami mutasi. Hal ini dan 52 juga menjadi penyebab terjadinya kanker dapat diamati dengan tidak adanya kekosongan serviks. yang garis vertikal pada urutan basanya, selain itu disebabkan oleh HPV tipe 45 adalah 7.4% untuk mempermudah penentuan pemilihan sedangkan HPV tipe 52 adalah 8.3 % (ICO HPV posisi information centre, 2014). Aldi, Y., et al (2015) sequence dalam rentang 50 basa. Berdasarkan menyatakan bahwa tingkat prevalensi HPV tipe pemisahan ini dapat diketahui daerah tanpa 45 untuk daerah sumatera barat dan Riau adalah mutasi (conversed) yang dapat dijadikan acuan 60,47%. dalam pemilihan posisi kandidat primer. Prevalensi Pada kanker penelitian serviks ini dengan memanfaatkan bioinformatika dilakukan desain pada primer website dapat dilakukan NCBI pemisahan Pemilihan kandidat primer Pemilihan kandidat primer dapat primer multiplex PCR gen E6 HPV tipe 45 dan dilakukan dengan memasukan sequence gen E6 HPV tipe 52. Desain primer dilakukan untuk HPV tipe 45 dan HPV tipe 52 pada AmplifiX 1.5.4 memperoleh primer yang spesifik dengan gen dan situs Primer3plus (www.primer3plus.com). target, harus Hal ini bertujuan agar diperoleh kandidat primer komplementer atau kurang lebih memiliki dengan sifat yang bervariasi. Pada kedua homologi yang cukup tinggi dengan urutan perangkat yang digunakan tersedia sifat-sifat sehingga urutan basanya primer seperti ukuran primer, posisi primer, 159 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 komposisi GC, dan amplikon. Pemilihan kandidat primer pada kedua perangkat didasarkan pada Tabel 1. Kriteria Desain Primer Kriteria Desain Primer Nilai Tm (ºC) 50 ºC -65 ºC sebelumnya. Komposisi GC 40 % - 60 % Analisis struktur sekunder dan sifat lainnya Panjang Primer 18 bp – 20 bp GC Clamp < 3 basa lainnya dilakukan pada kandidat primer yang Hairpin (kcal/mol) < -3 kcal/mol terpilih menggunakan perangkat Oligo Analyzer. Self dimer (kcal/mol) < -6 kcal/mol Perangkat ini menampilkan sifat-sifat seperti Cross dimer (kcal/mol) < -6 kcal/mol nilai haipin, self dimer dan cross dimer. Selain itu Runs < 4 basa dilakukan penghitungan nilai Tm primer secara Repeats < 4 basa manual dengan rumus : Tm mismatch (ºC) < 5 ºC daerah conversed yang telah di ketahui Analisis struktur sekunder dan sifat HASIL DAN DISKUSI Penelitian tentang desain primer untuk Penentuan jumlah GC clamp dilakukan mendeteksi gen E6 pada HPV tipe 45 dan HPV dengan cara menghitung jumlah basa G dan C tipe 52 pada pasien kanker serviks telah pada 5 basa terakhir ujung 3’ primer dengan dilakukan. Primer yang diperoleh bertutut-turut ketentuan jumlah optimal G dan C tidak lebih dari adalah forward 5’-TGC GGT GCC AGA AAC CAT 3 basa. Penentuan run dilakukan dengan TGA-3’; reverse 3’-TTT CTT GCC GTG CCT GGT menghitung jumlah pengulangan basa sejenis CA-5’ untuk HPV tipe 45 dengan panjang aplikon yang terjadi pada setiap sequence dengan 129 dan forward 5’-CAC GAA TTG TGT GAG GTG ketentuan pengulangan tidak boleh lebih dari 5 CTG-3’; reverse 3’-GGT CAC AGG TCG GGG TCT-5’ basa untuk HPV tipe 52 dengan panjang amplikon 402 (contoh: GGGGG). Penentuan repeat dilakukan dengan menghitung pengulangan 2 basa yang terjadi tidak boleh lebih dari 4 kali pengulangan (contoh: CTCTCTCT). Pemilihan primer bp. Penelitian rancangan dan pengujian primer HPV tipe 45 dan 52 telah dilakukan. Rancangan ditentukan primer dilakukan untuk pencarian primer terbaik berdasarkan analisis sifat-sifat kandidat primer yang akan digunakan untuk mengidentifikasi yang paling sesuai dengan kriteria desain primer HPV tipe 45 dan 52 menggunakan Multiplex seperti tabel berikut : Polymerase Chain Reaction (MPCR). Primer ini didesain dengan menggunakan situs resmi dan beberapa software seperti NCBI, primer3plus, AmplifX 1.5.4 dan Oligo Analyzer. Pencarian primer dilakukan pada situs resmi NCBI (www.ncbi.nlm.nih.gov) dengan kata kunci isolat gen E6 HPV tipe 45 dan isolat gen E6 HPV tipe 52. Isolat gen E6 HPV tipe 45 dan HPV tipe 52 telah diperoleh dari gene bank NCBI masing- 160 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 masing sebanyak 4 isolat. Isolat ini dianalisis secara molekuler untuk Setiap primer dianalisis hingga mempermudah didapatkan primer dengan kriteria terbaik yang pemilihan kandidat primer. Hal pertama yang dianalisis berdasarkan karakteristik primer yang dilakukan adalah penjajaran isolat HPV tipe 45 meliputi dan HPV tipe 52 dengan isolat reference. Isolat komposisi primer (% GC) dan panjang amplikon reference telah yang diperoleh dari primer3plus, sedangkan sifat dipublikasikan dan dijadikan acuan dalam sekunder primer yang meliputi haipin, self dimer, mendesain primer yang diperoleh dari situs dan cross dimer diproleh dari oligo analyzer. Nilai resmi Tm (time melting), run, repeat dilakukan secara merupakan international isolat HPV yang reference center (www.hpvcenter.se). posisi primer, panjang primer, manual. Penjajaran (alignment) dilakukan pada Primer yang didesain memiliki posisi program BLAST nukleotida (BLASTn) dalam conserved 100% sehingga dapat menghindari laman web NCBI. Berdasarkan penjajaran ini terjadinya mutasi pada saat amplifikasi. Panjang diperoleh daerah conserved, yaitu daerah yang primer yang diharapkan masuk dalam rentang memiliki homologi tinggi pada suatu isolat. 18-30 basa. Jika panjang primer kurang dari 18 Daerah daerah basa dapat mengakibatkan spesifisitas primer terjadinya mutasi, sehingga untuk mendesain rendah dan dapat memungkinkan terjadinya sebuah primer daerah non-conserved harus mispriming (penempelan primer ditempat yang dihindari. Sequence nukleotida dianalisis lebih tidak diinginkan), sedangkan untuk primer yang lanjut dengan membagi sequence dalam rentang lebih dari 30 basa tidak akan meningkatkan 50 nukleotida untuk mempermudah pencarian spesifisitas primer secara bermakna dan ini akan daerah conserved yang akan dijadikan sebagai menyebabkan biaya produksi yang lebih besar. posisi target desain primer. Tahap selanjutnya Panjang primer HPV tipe 45 forward adalah 21 dilakukan analisis molekuler, dimana dari hasil basa dan reverse adalah 20 basa, sedangkan HPV pemilahan daerah conserved, dipilih sequence tipe 52 forward adalah 21 basa dan reverse 18 dengan daerah conserved 100% (Widowati, basa (Handoyo & Rudiretna, 2001; Sasmito, dkk., 2013). 2014) non-conserved merupakan Pencarian kandidat primer dilakukan Perancangan sebuah primer perlu setelah menemukan daerah conserved untuk memperhatikan nilai Tm (Time melting) primer kedua tipe HPV. Pencarian kandidat primer yang merupakan temperatur dimana 50% untai dilakukan ganda terpisah sehingga primer dapat masuk dan pada (www.primer3plus.com) situs primer3plus dan AmplifX 1.5.4 beramplifikasi. Pemilihan Tm primer sangat dengan kriteria posisi primer termasuk dalam penting karna berpengaruh terhadap pemilihan daerah conserved. Berdasarkan daerah conserved suhu annealing pada proses PCR. Penentuan Tm dari masing-masing sequence didapatkan 3 primer dihitung secara manual menggunakan primer HPV tipe 45 yaitu 2 dari primer3plus dan rumus 1 dari AmplifX 1.5.4, sedangkan untuk HPV tipe Sebaiknya Tm primer berkisar antara 50 – 65 oC 52 berjumlah 8 primer dari primer3plus dan (Handoyo & Rudiretna, 2001). Primer HPV tipe tidak ada primer yang memenuhi syarat dari 45 dan HPV tipe 52 memiliki Tm yang sesuai AmplifX 1.5.4. dengan karakteristik Tm optimal, yaitu Tm HPV [2(A+T) + 4(C+G)] (lampiran 2). 161 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 tipe 45 forward 62 oC dan reverse 62 oC terjadi pada primer sejenis maupun dengan sedangkan HPV tipe 52 forward 62 oC dan reverse primer lainnya. Interaksi primer-primer harus 60 oC. Nilai Tm untuk primer HPV tipe 45 dan dihindari, demikian juga dengan mispriming HPV tipe 52 memenuhi kriteria rentang yang pada daerah lain yang tidak dikehendaki. Hal ini ditentukan yaitu 50-65 oC, sedangkan Tm diatas dapat menyebabkan spesifisitas primer menjadi 65 oC akan mengurangi efektivitas annealing rendah sehingga proses amplifikasi DNA kurang berjalan berpengaruh pada efisiensi proses PCR. baik (Widowati, 2013). dan terjadinya Analisis hairpin mispriming dilakukan dapat untuk Komposisi primer juga merupakan salah mengetahui ada tidaknya struktur sekunder satu pertimbangan penting. Deretan nukleotida yang terbentuk. Terbentuknya loop/hairpin yang sama pada setiap primer perlu dihindari sebaiknya dihindari agar tidak mengurangi karna dapat menurunkan spesifisitas primer dan spesifisitas primer. Batas nilai hairpin yang memungkinkan di diizinkan adalah kurang dari -3 kcal/mol tempat lain. Kandungan GC sebaiknya pada (Widowati, 2013). HPV tipe 45 hanya memiliki 1 rentang 40% - 60%. Primer dengan GC rendah hairpin yaitu pada primer forward dengan nilai tidak dapat berkompetisi untuk menempel ∆G= -1.08 kcal/mol, sedangkan HPV tipe 52 secara efektif pada tempat tujuan, sedangkan memiliki 3 hairpin pada primer forward yaitu primer dengan komposisi GC terlalu tinggi dapat dengan nilai ∆G = 0.04 kcal/mol, 0.04 kcal/mol, menyebabkan hasil menjadi tidak spesifik. 0.04 kcal/mol. terajadinya mispriming Komposisi GC primer HPV tipe 52 reverse Self dimer merupakan ikatan yang termasuk tinggi yaitu 66.7 % dibandingkan terbentuk antar primer sejenis, batas nilai self dengan forward 52.4%, HPV tipe 45 forward 55% dimer yaitu ∆G= -6 kcal/mol (Widowati, 2013). dan reverse 52%, namun primer HPV tipe 52 Pada HPV tipe 45 terbentuk 1 self dimer pada reverse memiliki struktur sekunder yang lebih primer forward dengan nilai ∆G= -1.20 kcal/mol baik dibandingkan dengan kandidat primer yang sedangkan pada HPV tipe 52 terbentuk 4 self lain. Selain itu nilai GC yang tinggi dapat diatasi dimer pada primer forward dengan nilai ∆G= - dengan pemilihan supermix yang tepat. Terdapat 2.16 kcal/mol, -0.08 kcal/mol, -0.08 kcal/mol, - berbagai macam supermix yang tersedia untuk 0.08 kcal/mol. membantu hasil amplifikasi PCR menjadi lebih Cross dimer merupakan ikatan yang speifik, salah satunya adalah Supermix KAPA2G terbentuk antar primer, yaitu antara primer Fast Multiplex PCR kit. Supermix ini khusus forward dan reverse HPV tipe 45 maupun HPV digunakan untuk multiplex PCR dan mengizinkan tipe 52. Batas nilai cross dimer yaitu ∆G= -6 nilai GC yang digunakan lebih dari 70% dengan kcal/mol (Widowati, 2013). Pada HPV tipe 45 ketentuan penambahan DMSO 5-10% pada primer nomor 3 terbentuk 5 cross dimer (∆G= - komposisi PCR (Handoyo & Rudiretna, 2001; 3.86, Sasmito, dkk., 2014; KAPA biosystem, 2014). sedangkan pada HPV tipe 52 primer nomor 8 Analisis struktur sekunder dilakukan dengan terbentuk 4 cross dimer (∆G= -3.61, -2.04, -0.34, - menggunakan Oligo Analyzer, diperoleh nilai 0.08 kcal/mol). -3.47, -3.41, -3.16, -0.31 kcal/mol) hairpin, self dimer dan cross dimer. Analisis ini Analisis cross dimer selanjutnya dilakukan sangat penting untuk mengetahui reaksi yang antar primer HPV tipe 45 dan HPV tipe 52. Ini 162 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 merupakan tahapan lanjutan untuk menemukan AAC CAT TGA-3’ dan reverse 3’-TTT CTT GCC GTG primer Multipex Polymerase Chain Reaction CCT GGT CA-5’ dan primer nomor 8 dengan (MPCR). Analisis ini bertujuan untuk mengetahui urutan forward 5’-CAC GAA TTG TGT GAG GTG ikatan yang terjadi diantara kedua primer CTG-3’ dan reverse 3’-GGT CAC AGG TCG GGG tersebut. Berdasarkan kandidat primer HPV tipe TCT-5’. Primer nomor 3 dan primer nomor 8 ini 45 (primer nomor 2 dan 3) dengan HPV tipe 52 memiliki ikatan yang paling sedikit dan nilai (Primer nomor 4, 7, 8), diperoleh primer nomor 3 cross dimer yang memenuhi syarat yaitu ∆G <-6 urutan nukleotida forward 5’-TGC GGT GCC AGA kcal/mol. Tabel 2. Accession number isolat gen E6 HPV 45 yang diakses pada NCBI Nomor Isolat 1 2 3 4 Isolat Accession Number B3587 B3730 B8463 B2408 KC662573.1 KC662572.1 KC662571.1 KC662570.1 Tabel 3. Accession number isolat gen E6 HPV tipe 52 yang diakses pada NCBI Nomor Isolat 1 2 3 4 Isolat Accession Number CZ52E429 CZ52A1023 CZ52A336 CZ52A277 JN874452.1 JN874451.1 JN874450.1 JN874449.1 Tabel 4. Posisi urutan nukleotida yang conserved dari gen E6 HPV tipe 45 No Urutan Nukleotida Jumlah isolat Persentase (%) 1 1-50 4 100 2 51-100 4 100 3 101-150 3 75 4 151-200 1 25 5 201-250 4 100 6 251-300 4 100 7 301-350 4 100 8 351-400 0 0 9 401-450 4 100 10 451-477 4 100 Keterangan : - Posisi adalah daerah conserved sequence isolat. - Jumlah isolat adalah banyak isolat yang memiliki posisi conserved - Persentase (%) adalah persentase jumlah isolat yang memiliki posisi conserved dari jumlah seluruh isolat yang digunakan 163 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 Tabel 5. Posisi urutan nukleotida yang conserved dari gen E6 HPV tipe 52 No Urutan Nukleotida Jumlah isolat Persentase (%) 1 1-50 4 100 2 51-100 4 100 3 101-150 4 100 4 151-200 4 100 5 201-250 0 0 6 251-300 0 0 7 301-350 4 100 8 351-400 3 75 9 401-450 4 100 10 451-477 4 100 Keterangan : - Posisi adalah daerah conserved sequence isolat. - Jumlah isolat adalah banyak isolat yang memiliki posisi conserved - Persentase (%) adalah persentase jumlah isolat yang memiliki posisi conserved dari jumlah seluruh isolat yang digunakan Tabel 6. Karakteristik Primer HPV Tipe 45 No Primer Panjang Posisi 1 F gacgatccaaagcaacgacc 20 13 R cgaagtctttcttgccgtgc 20 452 2 F acgaccctacaagctaccag 20 27 R tccctacgtctgcgaagtct 20 464 3 F tgcggtgccagaaaccattga 21 317 R tttcttgccgtgcctggtca 20 426 Keterangan : - Posisi adalah letak primer pada sequence target Tm (ºC) 62 62 62 62 63 62 GC (%) 55 55 55 55 52 55 Amplikon (bp) 440 Primer3plus 438 Primer3plus 129 AmplifX Metoda Tabel 7. Karakteristik Primer HPV Tipe 52 No 1 2 3 4 5 6 7 8 Primer Panjang Posisi F tgaggatccagcaacacgac 20 6 R ttacacttgggtcacaggcc 20 447 F gatccagcaacacgacccc 19 10 R tccaacactctgaacagcgc 20 421 F tgtttgaggatccagcaacac 21 2 R ccctgtccaacgacccataa 20 402 F cggaccctgcacgaattgt 19 28 R cggggtctccaacactctga 20 428 F cggggtctccaacactctga 21 37 R gtcacaggccggggtctc 18 437 F tgaggatccagcaacacgac 20 6 R tacacttgggtcacaggtcg 20 446 F cggaccctgcacgaattgt 19 28 R tcggggtctccaacactct 19 429 F cacgaattgtgtgaggtgctg 21 37 R ggtcacaggtcggggtct 18 438 Keterangan : - Posisi adalah letak primer pada sequence target Tm (ºC) 62 62 62 62 62 62 60 64 64 62 62 62 60 60 62 60 GC (%) 55 55 63.2 55 47.6 55 57.9 60 52.4 72.2 55 55 57.9 57.9 52.4 66.7 Amplikon (bp) Metoda 442 Primer3plus 412 Primer3plus 401 Primer3plus 401 Primer3plus 401 Primer3plus 441 Primer3plus 402 Primer3plus 402 Primer3plus 164 P ro sid ing Sem ina r Na siona l & Wo rkshop “Pe rkemba ngan Te rki ni Sa in s Fa rma si & K l in i k 5” | Padang , 6 -7 No vembe r 2015 KESIMPULAN CCT GGT CA-5’ sedangkan primer HPV tipe 52 Berdasarkan kriteria desain primer mempunyai panjang amplikon 402 bp terhadap kandidat primer HPV tipe 45 dan HPV mempunyai urutan basa nukleotida forward 5’- tipe 52 diperoleh primer HPV tipe 45 dengan CAC GAA TTG TGT GAG GTG CTG-3’ dan reverse panjang amplikon 129 bp mempunyai urutan 3’-GGT CAC AGG TCG GGG TCT-5’. basa nukleotida forward 5’-TGC GGT GCC AGA AAC CAT TGA-3’ dan reverse 3’-TTT CTT GCC GTG DAFTAR PUSTAKA Aldi, Y., Trisnawati, A.N., Putra, A.E., Djamaan, K., Marlina. (2015). Detection of hpv type 45 L2 gene in cervical cancer patients by polymerase chain reaction method. Int. J. Phram and Pharmaceutical Sci., 7(11). Aris, M., Sukenda, Enang Haris., M.Fatuchri., Munti Yuhana. (2013). Identifikasi molekular bakteri patogen dan desain primer PCR. Bioteknologi 1(3):43-50. Baxevanis, A.D., & Ouelette, B.F.F. (2005). Bioinformatics A Practical Guide to The Analysisof Genes and Protein, 3rd edition. Wiley Interscience. Bruni, L., Barrionuevo-Rosas L, Serrano B, Brotons M, Albero G, Cosano R, Muñoz J, Bosch FX, de Sanjosé S, & Castellsagué X. (2014). Human Papillomavirus and Related Diseases Report in Indonesia. Barcelona: ICO Information Centre on HPV and Cancer (HPV Information Centre). Handoyo, D., & Ari Rudiretna, (2001). Prinsip Umum Dan Pelaksanaan Polymerase Chain Reaction (PCR) [General Principles and Implementation of Polymerase Chain Reaction]. Unitas, 9 (1): 17-29. Haswan, N., Syahrul R., & Mardiah T. (2012). Infeksi Human Papilloma Virus dengan Perubahan Sitologi Serviks Pada Ibu Hamil. [cited 21 November 2014] tersedia dari URL : http://pasca.unhas.ac.id/jurnal. Kapa biosystem. (2014). KAPA2G Fast Multiplex PCR Kit. Technical Data Sheet. United States: Boston. Manuaba. (2009). Understanding the Female Reproductive Health. Jakarta: EGC. Marlina, Putra, A. E., & Burlis, T. (2014). Desain Primer Human Papillomavirus (HPV) Tipe 16 dan HPV Tipe 18 Penyebab Kanker Serviks secara Multiplex Polymerase Chain Reaction. Skripsi . Padang : Fakultas Farmasi Universitas Andalas (Unpublished). Marlina, Putra, A. E., & Yusal, C. M. (2014). Desain Primer Human Papillomavirus (HPV) Tipe 31 dan HPV Tipe 33 Penyebab Kanker Serviks untuk Metode Multiplex Polymerase Chain Reaction (PCR). Skripsi. Padang: Fakultas Farmasi Universitas Andalas (Unpublished). Sarwono, P. (2008). Ilmu Kandungan. Jakarta : Yayasan Bina Pustaka. Sasmito, D.E.K., Rahardian K., & Izzati M., Karakteristik Primer pada Polymerase Chain Reaction (PCR) untuk Sequencesing DNA : Mini Review di Seminar Nasional Infoematika Medis (SNIMed) V, 6 Desember 2014 : 90-102. Utama, A. (2003). Peranan Bioinformatika Dalam Dunia Kedokteran. [cited 20 September 2014] tersedia dalri URL: www.ilmukomputer.com. WHO/ICO Information Centre on HPV and Cervical Cancer (HPV Information Centre). (2010). Human papillomavirus and related cancers. Summary Report Update. 3rd edition. Available from URL : http://www.who.int/hpvcentre. Widowati, E.W. (2013). Desain Primer Sitokrom B (cyt b) sebagai salah satu komponen PCR (Polymerase Chain Reaction) untuk Deteksi DNA Babi. Yogyakarta : Universitas Islam Negeri Sunan Kalijaga. 165