TEKNIK PENGGUNAAN MARKA RAPD DENGAN PCR RINGKASAN
advertisement

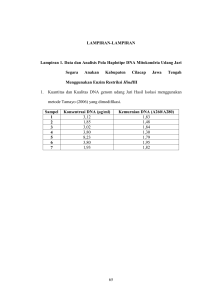
Temu Teknis Fungsional Non Peneliti 2001 TEKNIK PENGGUNAAN MARKA RAPD DENGAN PCR ZULQOYAH LAYLA Balai Penelitian Ternak,Po . Box 221 Bogor 16002 RINGKASAN PCR (Polymerase Chain Reaction) atau reaksi polimerase berantai adalah teknik amplifikasi DNA yang spesifik dengan cara melakukan proses pemanjangan nukleotida dari primer yang merupakan pasangan komplemen dari utas DNA secara simultan . Untuk mengatahui hasil amplifikasi, perlu dilakukan migrasi produk PCR di dalam gel (elektro-foresis) dan diamati dengan UVtransiluminator . Hasil visualisasi akan menunjukkan adanya perbedaan sifat diantara sampel yang diuji berdasarkan pada ukuran DNA yang terseparasi . Salah satu kegunaan proses PCR adalah untuk kajian keragaman molekuler seperti RAPD. Teknik RAPD (Random Amplified Polimorphic DNA) sederhana dalam pengerjaannya, memerlukan sedikit sampel, dan paling cepat dalam penggunaannya, namun relatif cukup mahal terutama pada enzym Taq polimerase. Dari masing-masing 5 sampel darah domba bangsa Garut, Sumatera, StCroix, Merino, dan Ekor Gemuk, telah berhasil diisolasi sebanyak 25 nomor DNA dengan kemumian berkisar antara 1,440-2,330, serta memiliki konsentrasi antara 87,5ng/ul - 1912,5 ng/ul . Dari hasil separasi produk PCR dengan primer OPH-03, muncul pita-pita dengan jumlah berfariasi antara 2-4 . Hal ini menunjukkan bahwa bangsa domba yang diperiksa memiliki sifat polimorfis . PENDAHULi1AN PCR (Polymerase Chain Reaction) atau reaksi polimerase berantai adalah teknik amplifikasi DNA yang spesifik dengan cara melakukan proses pemanjangan nukleotida dari primer yang merupakan pasangan komplemen dari utas DNA secara simultan . Proses pemanjangan nukleotida merupakan proses polimerase yang dilakukan oleh DNA karena adanya "primer" yang berkomplemen dengan DNA utas tunggal .(SUHARSONO, 2000). Pengulangan dari siklus berdasarkan pada perubahan suhu yang terdiri dari tahap "denaturasi", "annealing " dan "extension" . Tahap "denaturasi" merupakan tahap awal dimana pada tahap ini DNA yang merupakan rantai utas panda akan terlepas sehingga membentuk rantai utas tunggal . Tahap "annealing" merupakan tahap dimana terjadi perlekatan primer pada ikatan DNA Was tunggal_ Primer yang memiliki sekuens basa tertentu akan mengenali sekuens basa DNA utas tunggal . Tahap selanjutnya adalah tahap "extension" atau pemanjangan , dimana pada tahap ini terjadi pemanjangan primer dengan 147 Temu Teknis Fungsional Non Peneliti 200/ bantuan enzym Taq DNA polimerase. Hasil produk ini berfungsi sebagai template (cetakan) pada siklus berikutnya. PCR sangat sensitive, dapat mengamplifikasi sampai lebih dari sejuta kali, sehingga dapat menghasilkan DNA dalam jumlah yang sangat besar. Karena dapat mengamplifikasi demikian banyak, maka DNA cetakan (template) dibutuhkan dalam jumlah yang sedikit.(SUHARSONO, 2000) . Untuk mengetahui hasil amplifikasi maka perlu dilakukan migrasi produk PCR di dalam gel (elektroforesis) dan diamati dengan UV transiluminator . Hasil visualisasi akan menunjukkan adanya perbedaan sifat diantara sampel yang diuji , berdasarkan pada ukuran DNA yang terseparasi. Proses PCR dapat dipergunakan untuk berbagai keperluan diantaranya untuk identifikasi suatu gen atau DNA yang spesifik ; untuk identifikasi adanya suatu patogen penyebab suatu penyakit, seperti HepatitisB, TBC, AIDS, atau kelainan lainnya ;perbanyakan gen untuk berbagai keperluan ; untuk kajian keragaman molekuler seperti RAPD(Random Amplified Polimorphic DNA) ; untuk pengurutan DNA, dll . RAPD merupakan salah satu teknik yang paling luas dipergunakan karena kesederhanaannya .. Primer yang digunakan adalah primer oligonukleotida dimana urutan basanya dibuat secara random (acak) . Dalam RAPD diperlukan sedikit sampel DNA untuk analisa . Teknik ini merupakan teknik yang paling cepat dalam penggunaannya, namun relatif cukup mahal, tenitama untuk enzym Taq DNA polimerase .(WILLIAM at al, 1990). Tujuan penulisan ini adalah untuk mengemukakan salah satu metode isolasi DNA, pelaksanaan metode marka RAPD yang digunakan di laboratorium Genetika Balitnak Bogor terhadap sampel darah domba . BAHAN DAN METODA Alat dan bahan yang digunakan Mikropipet, tips, tabung eppendorf 1500 ul, tabung eppendorf 200 ul, mikrosentrifus, spektrofotometer double beam UV150-02 Shimadzu, tangki elektroforesis, cetakan gel + sisir mesin PCR MJ Research, kamera Polaroid., UV transiluminator, sarung tangan latex. Larutan yang digunakan Penyangga TE, penyangga TBE, NP-40, SDS, alkohol 70 %, alkohol absolut, NaOAc 3M, Proteinase-K , buffer PCR(Promega), dATP(Promega), dCTI'(Promega), dGTP(Promega), dTTP(Promega), MgCl z(Promega), DNA Taq polimerase(Promega), primer OPH-03(Operon), dd HZO. Penanda ukuran molekuler 1 Kb dan 100 bp. Temu Teknis Fungsional Non Peneliti 100/ Metode Isolasi DNA DNA cetakan diisolasi dari darah domba berdasarkan metode at al, (1989) yang telah dimodifikasi . Sebanyak 5 ekor dari masing masing domba bangsa Ekor gemuk, Merino, Garut, StCroix dan Sumatra diambil darahnya secara aseptis melalui vena jugularis . Pengambilan darah sebanyak 5 ml dimasukkan ke dalam tabung vacutainer yang telah berisi anti koagulant EDTA. Sebanyak 350 ul plasma darah dimasukkan ke dalam tabung eppendorf 1500ul, ditambah sebanyak 70 ul larutan NP40, 350 ul larutan SDS dan 10 ul Proteinase K . Tabung divortex, kemudian diinkubasi pada suhu 50°C semalam . Keesokan harinya ditambah 350ul larutan fenol, disentrifugasi pada 13000 rpm selama 5 menit . Fase atas diambil untuk dipindahkan ke dalam tabung eppendorf baru, ditambah 1/10 bagian larutan NaOAc 3M dan 2 bagian larutan alkohol absolut dingin . Tabung digoyang perlahan ( dasar tabung di jentik jentik ) hingga terbentuk endapan putih (DNA) . Tabung disimpan pada suhu --20°C semalam . Keesokan harinya tabung disentrifugasi pada kecepatan 13000 rpm selama 5 menit . Cairan yang ada di dalam tabung di buang dengan hati hati hingga yang tersisa berupa endapan (DNA) berwarna putih . Ke dalam tabung yang berisi DNA tersebut ditambahkan sebanyak 1 ml larutan alkohol 70 %, campur perlahan, sentrifugasi pada kecepatan 13000 rpm selama 5 menit. Cairan alkohol dibuang, tabung diinkubasi pada suhu 50°C selama 1 jam , atau dikering anginkan hingga terbentuk pelet DNA didasar tabung . Ke dalam tabung yang berisi pelet DNA diisikan sebanyak 200 ul larutan TE untuk melarutkan kembali DNA, diinkubasi pada suhu 65°C sampai DNA larut. Ke dalam DNA ditambahkan 5 ul larutan RNAse, diinkubasi selama 2 jam pada suhu 37°C, kemudian direbus selama 5 menit untuk menghentikan reaksi. DNA yang dihasilkan selanjutnya diukur kualitas dan kuantitasnya . Kuantitas DNA dapat dilihat dengan UVspektrofotometer pada panjang gelombang 260 nm dan 280 nm. Adapun prosedurnya yaitu memasukkan sebanyak 5 ul DNA ke dalam tabung cuvet ditambah 2495 ul TE sebagai pengencer, dilihat (dibaca) nilai optikal densitinya pada UVspektrofotometer dengan panjang gelombang 260nm dan 280 nm. Sebagai larutan standar dipakai larutan penyangga TE. Dari hasil pengecekkan tersebut didapat data kemurnian DNA yang dapat dihitung dengan melihat perbandingan bacaan pada spektrofotometer dengan panjang gelombang 260 nm dan 280 nm. Selanjutnya dari hasil pengukuran tersebut dapat ditentukan jumlah DNA sampel yang diperoleh dengan rumus : Jumlah DNA = [ 260 x 2500/5 x 50 ]/1000. Informasi kuantitas ini dapat digunakan untuk menentukan perhitungan dalam pengenceran DNA agar dapat diketahui secara tepat konsentrasi DNA diinginkan. Uji kualitas DNA dapat dilakukan dengan elektroforesis . DNA basil isolasi diambil sebanyak 3 ul dan dicampur dengan larutan blue juice (pemberat/penanda migrasi), lalu diisikan ke dalam sumuran gel yang SAMBROOK 149 Temu Teknis Fungsionnl Non Peneliti 2001 mengandung 1 % agarose . Pada sumuran lain, diisi penanda DNA berukuran 1Kb . Gel dielektroforesis dengan tegangan 90 volt selama 1,5 jam, kemudian direndam dalam larutan Ethidium Bromida selama 10 menit, dibilas dengan merendam gel di dalam air suling, diamati dengan bantuan UV transiluminator, bila perlu selanjutnya difoto dengan kamera Polaroid . Metode RAPD dengan PCR 1. 2. Sampel DNA yang akan diuji masing-masing diencerkan dengan konsentrasi 5 ng/ul .dalam larutan penyangga TE. Untuk setiap bangsa dibuat larutan gabungan (bulk) 10 ul DNA 5ng/ul setiap individu dijadikan satu. Membuat larutan campuran(cocktail) untuk tiap sampel bulk DNA ke dalam tabung eppendorf isi 200 ul dengan komposisi : Buffer PCR [ Promega] dATP [Gibco] dCTP [ Gibco] dGTP [ Gibco] dTTP [ Gibco] MgC1 2 [ Promega] Primer [Operon] H-03 Taq DNA Polimerase [Promega] ddH Z0 Templat DNA 5ng/ul Total volume 2,5 ul 0,5 ul 0,5 ul 0,5 ul 0,5 ul 1,5 ul 1,25 ul 1,2 ul 7,5 ul 10 ul 25 ul "Cocktail "yang sudah dibuat, kemudian disentrifugasi dengan kecepatan 6000 rpm agar bahan- bahan dalam "cocktail" dapat tercampur sempurna. Untuk menghindari terjadinya penguapan pada saat proses amplifikasi berlangsung, diatas permukaan larutan "cocktail "dilapisi minyak mineral sebanyak 25 ul . "Cocktail" dibuat dengan tujuan untuk memudahkan cara kerja. Buffer PCR berfungsi sebagai larutan penyangga pada proses amplifikasi . Sedangkan dATP dCTP dGTP, dTTP, digunakan untuk pemanjangan primer dalam membentuk utas DNA baru. Primer yang digunakan adalah OPH-03 buatan Operon yang mempunyai urutan basa 5'-AGACGTCCAC-3' . Enzim Taq DNA polimerase berfungsi untuk memanjangkan primer pada tahap "elongasi/extension (BUDIARTI.P,1993). Amplifikasi DNA dengan PCR Setelah "cocktail" selesai dibuat maka tahap selanjutnya adalah mengamplifikasi DNA sample dengan mesin PCR (MJ Research Inc ). 150 Temu Teknis Fungsional Non Peneliti 200/ Amplifikasi dilakukan sebanyak 45 siklus. Setiap siklus terdiri dari tiga tahap yaitu tahap "denaturasi" dengan suhu 94°C selama 1 menit, tahap "annealing" dengan suhu 36°C selama 1 menit dan tahap "elongasi" dengan suhu 72°C selama 2 menit . Setelah siklus selesai, selanjutnya PCR akan melakukan proses inkubasi pada suhu 72°C selama 5 menit. Waktu inkubasi ini dimaksudkan untuk memastikan bahwa DNA yang diamplifikasi telah mengalami renaturasi . Setelah proses amplifikasi selesai selanjutnya tabung eppendorf disimpan pada suhu 0°C sampai saatnya untuk melakukan separasi dalam gel agarose . Mengetahui hasil amplifkasi [separasil Hasil amplifikasi dapat dilihat dalam gel agarose yang diamati dengan UVtransiluminator. Tahapannya adalah sebagai berikut : 1 . Membuat larutan agarose 1,2% dalam larutan penyangga 0,5x TBE dengan cara memanaskannya hingga larut, menuangkannya ke dalam cetakan husus dengan sisir terpasang, biarkan sampai beku. Sisir selanjutnya dilepas . 2 . Cetakan yang berisi gel beku dimasukkan ke dalam tangki elektroforesis yang sudah diisi larutan penyangga 0,5xTBE hingga terendam. 3 . Sebanyak 3ul sampel DNA hasil amplifikasi dicampur dengan 2ul larutan pemberat , diaduk hingga homogen . Tujuan pemberian larutan pemberat adalah sebagai indikator warna dan juga sebagai pemberat sehingga ketika sampel DNA dimasukkan ke dalam sumuran gel, dapat turun kedasar sumuran. Sumuran paling kanan diisi penanda ukuran molekuler 100 by buatan Promega dengan isi yang sama. . 4. Proses separasi (elektroforesis) dilakukan pada tegangan 90 volt selama sekitar 2jam. 5 . Separasi dihentikan apabila sampel DNA telah bergerak hampir mendekati akliir gel . Visualisasi fragmen DNA dalam gel Setelah proses separasi selesai, selanjutnya gel direndam dalam larutan Ethidium Bromida dengan konsentrasi 0,5 ul /ml selama 10 menit. Tujuan perendaman ini diharapkan Ethidium Bromida dapat menyisip pada DNA, sehingga pita- pita DNA yang terseparasi dapat terlihat dengan bantuan sinar UV dart UV transiluminator . Selanjutnya gel direndam dalam dH,O selama 10 menit, untuk menghilangkan senyawa Ethidium Bromida yang terikat secara lion spesifik pada bagian gel yang tidak ada DNAnya . Bila tidak dilakukan perendaman, maka pita-pita DNA akan tampak pucat karena pendaran latar belakang gel yang mengikat Ethidium Bromida. Gel dikeluarkan dart rendaman air suling dan diletakkan diatas pennukaan UVtransiluminator untuk diarnati ada tidaknya pita-pita DNA, dan bisa dilanjutkan pemotretan dengan menggunakan kamera Polaroid. Temu Teknis Fungsional Non Peneliti 2001 HASIL DAN PEMBAHASAN LIsolasi DNA Dari sebanyak masing-masing lima nomor darah yang berasal dari domba Garut, St.Croix, Merino, Sumatera, dan Ekor Gemuk, dengan menggunakan metode Sambrook yang telah disempurnakan, telah diisolasi sebanyak 25 nomor DNA . 2Xualifikasi DNA Kualitas DNA yang dihasilkan dapat diamati pada gambar 1 . Gambar l . Isolasi DNA Keterangan I So'a= 108 2 .Sumatera I I I 3. Sumtera 119 4. Sumatera 120 5. Sumatera 128 6 Ekor Gemuk M1 7. Ekor Gemuk 111 8. Ekor gemuk Sus 4 9. Ekor Gemuk Sus2 10 Fkor Gemuk Isa 2 11 . 12 . 13 . 14 . 15 . Merino 1003 Merino 1023 Merino 1249 Merino1036 Merino 1109 16 . 17 . 18 . 19 . 20. Garut Garut Garut Garut Garut 3579 3613 9062 1094 9105 21 . 22 . 23 . 2425 . St Croix 40015 St Croix32257 St .Croix 20113 St . Croix 519 St.Croix 603 Kemurnian DNA berdasarkan basil bagi nilai absorbensi pada panjang gelombang 260 terhadap nilai absorbensi pada panjang gelombang 280 dari sampel DNA bangsa Ekor Gemuk, Sumatera, Garut, St.Croix dan Merino, berada antara 1,440-2,330 . Menurut standard yang ditetapka kemurnian DNA sebaiknya berada pada kisaran 1,8 (SAMBROO at al, 1989). Karena untuk pengerjaan RAPD sifatnya adalah random (acak), maka DNA dengan kisaran trilai 1,44-2,33 masih dapat digunakan . Temu Teknis Fungsional Non Peneliti 2001 3. Kuantifikasi DNA Dari hasil kuantifikasi DNA dengan spektrofotometer, didapat konsentrasi DNA seperti terdapat pada tabel 1 . Konsentrasi DNA total yang dihasilkan berkisar antara 87,5 ng/ul - 1912,5 ng/ul . Tabel 1 . Kuantifikasi DNA Domba pada panjang gelombang 260 nm No. Spesimen X11 C7 1 2 3 4 EG Ml H1 Sus4 SUS2 5 6 7 8 9 10 11 12 13 14 15 16 17 ISA2 Mer 1003 1023 1249 1036 1109 Smt 108 120 119 111 128 Grt 3579 3613 9062 1094 9105 StCr40015 32257 20113 519 603 0,018 0,020 0,034 0,036 0,013 0,013 0,040 0,008 0,007 0,014 0,040 0,062 0,113 0,026 0,067 0,031 0,013 0,043 0,020 0,016 0,047 0,036 0,015 0,053 0,055 18 19 20 21 22 23 24 25 XE 0 0,011 0,012 0,019 0,019 0,007 0,006 0,022 0,004 0,003 0,006 0,025 0,0036 0,063 0,018 0,038 0,017 0,007 0,025 0,012 0,008 0,029 0,022 0,008 0,027 0,029 Konsentrasi DNA ng/ul 225 250 430 450 163 162,5 500 100 87,5 175 500 775 1912,5 325 837,5 387,5 162,5 537,5 250 200 587,5 450 187,5 662,5 687,5 Kemumian 1,640 1,670 1,800 1,890 1,860 2,170 1,820 2,000 2,330 2,330 1,600 1,720 1,800 1,440 1,760 1,820 1,860 1,720 1,670 2,000 1,620 1,640 1,880 1,960 1,900 4. Visualisasi produk PCR Hasil visualisasi yang dapat dilihat pada gambar 2, menunjukkan bahwa pita-pita DNA terlihat jelas untuk setiap bangsa domba dengan jumlah 153 Temu Teknis Fungsional Non Peneliti 2001 pita yang muncul berkisar antara 2-4 pita. Jumlah pita yang muncul bervariasi, yaitu 4 dari domba bangsa StCroix, Sumatera, dan Garut dan 2 buah pada domba bangsa Merino dan Ekor Gemuk. Dengan terbentuknya pita DNA dengan primer OPH-03, maka primer ini dapat digunakan untuk analisis keragaman bangsa-bangsa domba. Pada hasil visualisasi menunjukkan sifat polimorfis, artinya antar bangsa memiliki ukuran DNA yang berbeda berdasarkan ukuran pasangan basa . Hal ini menunjukkan adanya perbedaan sifat diantara bangsa domba yang diamati yang dapat dilihat dari ukuran DNA yang terseparasi. Sifat polimorfis inilah yang diharapkan muncul dari analisa RAPD ini. Tabel 2.. Larutan untuk proses persiapan sampel Nama larutan 1 M TrisCl . pH 8,5 1 M TrisCl . pH 7,5 5M Nacl TBE buffer 5x TE buffer NaOAc 3M Penyangga NP-40 SDS Blue juice 70 % alkohol Proteinase K Komposisi 6,06 gr(40 ml air bebas ion + 1ml HCI pekat) 6,06 gr(40 ml air bebas ion + 2ml HCI pekat 14,61 gr Nacl tris base 54 gr boric acid 27,5 gr 0,5 M EDTA pH 8,0 20 ml 1M Tris Hcl pH 8,0 10 ml 0,5 M EDTA pH 8,2 ml 62,52 gr NaOAc anhydrous 102,02 gr NaOAc 3H20 pH 5,2 (acetat glasial) 5M Nacl 2,80 ml 1M MgC12 0,15 ml lM Tris Hcl pH 8,5 10 ml 10% SDS 6ml 0,5M EDTA 1,2m1 5M Nacl 1,2 ml 0,125 gr blue juice + 20 gr sucrose 70 ml alkohol absolut 1M TrisCI pH 7,5 50u1 Proteinase K 100mg(18 unit/mg) glyserol 100% 5 ml Pelarut/volume akhir air bebas ion/50 ml air bebas ion/50 ml air bebas ion/50 ml air bebas ion/1000 ml air bebas ion/ 1000 ml air bebas ion/ 150 ml air bebas ion/ 100 ml air bebas ion/50 ml air bebas ion/50 ml air bebas ion/100 ml air bebas ion/ 10 ml Temu Teknis Fungsional Non Peneliti 1001 Ekor Gemul Garut Merino Surnatera St. Croix 100 by Gambar 2. Hasil separasi amplifikasi dengan primer 01H-03 KESIMPi1LAN Dari hasil amplifikasi DNA yang dihasilkan dengan teknik RAPD menggunakan mesin PCR memperlihatkar_ bahwa munculnya pita-pita dengan jutnlah yang bervariasi yaitu antara 2-4 . Ini menunjukkan bahwa primer OPH 03 dapat mengenali DNA domba, sehingga primer ini dapat melakukan pembentukan komplemen sekuen DNA. Perbedaan ukuran pasangan basa antar bangsa domba yang terlihat pada gambar, menunjukkan sifat polimorfis dari bangsa domba yang diperiksa . DAFTAR BACAAN BUDIARTI S . 1993 . Tehnik PCR dan aplikasinya, Kursus Singkat Biologi Molekuler PAU . Bioteknologi IPB. SAMBROOK J.E .F, FRITISCH AND T.MANIATIS . 1989. Molecular Cloning . A 2nd Laboratory manual Ed. Cold Spring Harbor Laboratory Press . Analysis and Cloning of Eukaryotic Genomic DNA,9.19 . SUHARSONO S. 2000. Prinsip Amplifikasi DNA dengan PCR . Pelatihan Peningkatan Pengetahuan dan Ketrampilan Teknisi Tentang Teknik Laboratonum Biologi Molekuler .PAU . IPB . WILLIAM J.G.K, A.R .KUBELIC, K.J. LIVAK J.A. RAFALSKI AND S.V. TINGEY, 1990. DNA Polymorphis Amplified by Arbitary Primer are usefull as Genetic Marker, Nucleic Acid-Res. 18 :6531-6535 .