BAB IV Hasil dan Pembahasan Hasil yang diperoleh dari tahapan
advertisement

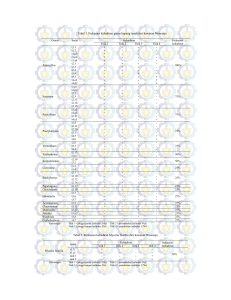
BAB IV Hasil dan Pembahasan Hasil yang diperoleh dari tahapan penelitian akan dijelaskan pada bab ini. Dimulai dengan amplifikasi gen katG, penentuan urutan nukleotida (sequencing), dan diakhiri dengan analisis in silico. IV.1 Amplifikasi gen katG M. tuberculosis galur alami H37Rv dan isolat L5 MDR-TB Hasil amplifikasi gen katG M. tuberculosis H37Rv dan isolat L5 MDR-TB dapat dilihat dari visualisasi hasil elektroforesis gel agarosa pada gambar IV.1, M. tuberculosis H37Rv ditunjukkan sebagai kontrol wild type. 1 2 3 4 1417 pb 517 pb 397 pb 0,43 kb 214 pb 75 pb Gambar IV.1 Elektroforesis agarosa M.tuberculosis H37RV dan isolat L5 MDRTB. Tampak pada gambar lajur 1 adalah penanda (marker) DNA puC19/HinfI, lajur 2 M.tuberculosis galur alami H37Rv, lajur 3 isolat L5 MDR-TB, dan lajur 4 adalah kontrol negatif. Pada gambar tampak kedua pita sampel berukuran 0,43 kb. Elektroforesis gel agarosa M. tuberculosis galur alami H37Rv dan isolat L5 MDR-TB memperlihatkan perkiraan hasil pita DNA berukuran 0,43 kb. Letak pita tersebut berada pada posisi antara pita kedua dan ketiga pita penanda (marker) pUC19/HinfI dengan ukuran 0,512 kb dan 0,397 kb. Ukuran fragmen DNA yang diperoleh sesuai dengan perkiraan panjang fragmen yang menggunakan primer KF dan KR pada program Editseq, DNA*STAR. Posisi fragmen DNA kedua sampel dalam gen katG diperlihatkan pada gambar IV.2. 738 5’ 1172 3’ KF 2,359 kb KR 3’ 5’ 5’ 3’ 3’ 5’ 0,43 kb Gambar IV.2 Posisi fragmen PCR pada gen katG M. tuberculosis. Pada gambar terlihat posisi fragmen DNA dimulai dari urutan nukleotida ke 738 hingga 1172 pada gen katG. Jumlah basa dalam fragmen DNA adalah 430 pb. IV.2 Hasil penentuan urutan nukleotida Data urutan nukleotida yang diperoleh berupa elektroforegram dalam bentuk file abi. Masing-masing basa nukleotida gen katG kedua sampel ditunjukkan oleh warna yang berbeda, yaitu warna hijau untuk basa adenin (A), warna biru untuk sitosin (C), warna hitam untuk guanin (G) dan warna merah untuk timin (T). Elektroforegram dapat dilihat pada gambar IV.3. Gambar IV.3 Elektroforegram hasil sequencing isolat L5 MDR-TB. Elektroforegram isolat L5 MDR-TB memperlihatkan puncak basa penyusun fragmen DNA. Basa timin berwarna merah, adenin berwarna hijau, sitosin berwarna biru, dan guanin berwarna hitam. Selain dalam bentuk elektroforegram, didapatkan pula hasil sequencing dalam bentuk teks. Data teks urutan nukleotida fragmen DNA isolat L5 MDR-TB ditunjukkan pada gambar IV.4. AGGCGAGGGA CCGGAATCCA TGGCCGCGGC GGTCGACATT 40 CGCGAGACGT TTCGGCGCAT GGCCATGAAC GACGTCGAAA 80 CAGCGGCGCT GATCGTCGGC GGTCACACTT TCGGTAAGAC 120 TACATGGCGC CGGCCCGGCC GATCTGGTCG GCCCCGAACC 160 CGAGGCTGCT CCGCTGGAGC AGATGGGCTT GGGCTGGAAG 200 AGCTCGTATG GCACCGGAAC CGGTAAGGAC GCGATCACCA 240 GCGGCATCGA GGTCGTATGG ACGAACACCC CGACGAAATG 280 GGACAACAGT TTCCTCGAGA TCCTGTACGG CTACGAGTGG 320 GAGCTGACGA AGAGCCCTGC TGGCGCTTGG CAATACACCG 360 CCAAGGACGG CGCCGGTGCC GGCACCATCC CGGACCCGTT 400 ATGAACTTGG GC 412 Gambar IV.4. Data teks urutan nukleotida isolat L5 MDR-TB. Data teks ini memperlihatkan urutan nukleotida isolat L5 MDR-TB dalam bentuk teks, nukleotida yang dapat dibaca berjumlah 412 pb. Hasil sequencing isolat L5 MDR-TB dalam bentuk teks menunjukkan nukleotida yang dapat di baca berjumlah 412 pasang basa (pb) dari 430 pb dan nukleotida yang dapat di baca pada M. tuberculosis H37Rv berjumlah 356 pb dari 430 pb. Urutan nukleotida M. tuberculosis H37Rv dapat dilihat pada lampiran. Pada penelitian sebelumnya telah dilaporkan bahwa mutasi gen katG G944T yang merubah residu asam amino Ser315Thr merupakan mutasi yang paling sering terjadi (Mokrousov et al., 2002), namun pada penelitian ini mutasi tersebut tidak ditemukan. 944 M. tuberculosis H37Rv 944 Isolat L5 MDR-TB Gambar IV.5 Eletroforegram nukleotida posisi 944. Grafik ini menunjukkan tidak ada mutasi pada posisi basa 944, penelitian sebelumnya melaporkan posisi ini merupakan posisi yang paling banyak mengalami mutasi. IV.3 Analisis homologi Analisis homologi gen katG antara M. tuberculosis galur alami H37Rv dan isolat L5 MDR-TB dimulai dengan memasukkan urutan nukleotida kedua sampel dalam bentuk abi file ke dalam program SeqmanTM versi 4.0.0. Setelah disejajarkan program akan secara otomatis memberi tanda adanya mutasi, pada pensejajaran antara dua sampel diatas dapat terlihat adanya perbedaan basa pada posisi yang sama, yaitu C pada M. tuberculosis galur alami H37Rv dan T pada isolat L5 MDR-TB. Hal ini menandakan adanya mutasi subsitusi C menjadi T. Posisi nukleotida yang mengalami mutasi dapat diketahui dengan memasukkan urutan nukleotida lengkap gen katG M. tuberculosis galur alami H37Rv yang berasal dari GenBank, pensejajaran ini berhasil menunjukkan posisi nukleotida yang mengalami mutasi, yaitu posisi 825. M. tuberculosis H37Rv Isolat L5 MDR-TB Gambar IV.6 Elektroforegram fragmen DNA gen katG M. tuberculosis H37Rv dan Isolat L5 MDR-TB. Pada grafik terlihat adanya mutasi C825T pada isolat L5 MDR-TB, analisis menggunakan program Seqman, DNA*STAR. 825 Gen katG H37Rv L5 Gambar IV.7 Posisi nukleotida 825 isolat L5 MDR-TB yang mengalami mutasi C825T. Pada gambar ini terlihat posisi nukleotida yang mengalami mutasi berada pada urutan ke 825. selain itu, ditunjukkan pula perbedaan warna pada nukleotida yang mengalami mutasi. Untuk melihat adanya perubahan asam amino akibat mutasi C825T, maka dilakukan pensejajaran antara nukleotida M. tuberculosis H37Rv, isolat L5 MDRTB dan open reading frame (ORF) gen katG yang diperoleh dari GeneBank. Hasil pensejajaran dapat dilihat pada gambar IV.6. Gen katG 796 266 267 268 269 270 271 272 273 274 275 276 277 278 279 280 281 atc gtc ggc ggt cac act ttc ggt aag acc aca tgg cgc cgg ccc ggc Ile Val Gly Gly His Thr Phe Gly Lys Thr Thr Trp Arg Arg Pro Gly I V G G H T F G K T T W R R P G M. tuberculosis H37Rv 796 266 267 268 269 270 271 272 273 274 275 276 277 278 279 280 281 atc gtc ggc ggt cac act ttc ggt aag acc aca tgg cgc cgg ccc ggc Ile Val Gly Gly His Thr Phe Gly Lys Thr Thr Trp Arg Arg Pro Gly I V G G H T F G K T T W R R P G Isolat L5 MDR-TB : 796 266 267 268 269 270 271 272 273 274 275 276 277 278 279 280 281 atc gtc ggc ggt cac act ttc ggt aag act aca tgg cgc cgg ccc ggc Ile Val Gly Gly His Thr Phe Gly Lys Thr Thr Trp Arg Arg Pro Gly I V G G H T F G K T T W R R P G Gambar IV.8 Posisi dan jenis asam amino yang dikode oleh kodon 275. Terlihat pada gambar asam amino yang mengalami mutasi adalah Thr275. Gambar diatas menunjukkan bahwa mutasi pada nukleotida 825 terletak pada kodon 275 basa ketiga, ACC menjadi ACT. Perubahan basa ini tidak merubah asam amino 275, treonin. Tidak adanya perubahan residu asam amino 275 maka dapat dipastikan mutasi tersebut bukan merupakan penyebab sifat resisten terhadap INH. Pada penelitian sebelumnya isolat L5 MDR-TB telah diamplifikasi menggunakan metode PCR multipleks katG315, primer yang digunakan adalah dua primer luar KF dan KR serta satu primer dalam K315, daerah gen katG yang diamplifikasi berada pada urutan 738 hingga 1172. Hasil amplifikasi isolat tersebut menghasilkan dua pita berukuran 0,29 dan 0,43 kb, yang berarti isolat L5 tidak mengalami mutasi S315T, namun dari hasil PCR multipleks tidak ditentukan urutan nukleotidanya, sehingga mutasi pada isolat L5 belum diketahui. Pada penelitian ini, untuk mengamplifikasi gen katG digunakan dua primer luar yang dipakai pada PCR multipleks. Berdasarkan penentuan urutan nukleotida dan setelah dilakukan analisis homologi, ditemukan mutasi C825T pada isolat L5 MDR-TB, tetapi tidak merubah residu asam amino T275. Sehingga dapat dikatakan mutasi yang menyebabkan sifat resisten antibiotik diduga berada diluar fragmen yang dianalisa, mengingat primer KF dan KR mengamplifikasi daerah gen katG sepanjang 0,43 kb sedangkan jumlah urutan nukleotida lengkap gen katG 2,3 kb, maka diperlukan penelitian lanjutan yang mengamplifikasi daerah diluar fragmen yang telah diamplifikasi. 1 kb 2,3 kb T275 S315 0,43 kb Gambar IV.9 Fragmen amplifikasi DNA pada gen katG M. tuberculosis Gambar ini menujukkan letak fragmen DNA yang diamplifikasi, letak residu asam amino yang mengalami mutasi (merah) dan letak Ser315 yang tidak mengalami mutasi (hijau). IV.4 Model struktur protein katalase peroksidase Protein katalase peroksidase M. tuberculosis telah berhasil dikristalkan dan ditentukan struktur tiga dimensinya (Bertrand et al., 2004). Struktur tiga dimensi dari enzim ini meperlihatkan bahwa enzim tersebut adalah homodimer dengan masing-masing monomer berukuran 80 kDa. Data struktur protein dapat dilihat pada www.pdb.org dengan nama ISJ2. Posisi residu asam amino T275 berada dekat dengan sisi pengikatan substrat INH, sisi pengikatan substrat tersebut berada di domain N. Gambar IV.10 Struktur tiga dimensi enzim KatG M. tuberculosis (pdb kode ISJ2). Gambar diatas menunjukkan bahwa enzim KatG merupakan homodimer. Perbedaan warna menunjukkan masing-masing monomer. Mutasi C825T pada isolat L5 MDR-TB tidak menyebabkan perubahan struktur pada posisi 275 enzim KatG, tidak berubahnya struktur maka fungsi enzim tersebut tidak mengalami perubahan. Informasi tersebut menjelaskan bahwa penyebab resistensi berada di daerah lain pada struktur KatG. Selain itu, mutasi diduga berada pada protein target INH yaitu InhA. Gambar IV.11 Posisi asam amino Thr275 pada model tiga dimensi enzim KatG M. tuberculosis. Warna merah tua menunjukkan atom O, warna merah muda menunjukkan atom C dan biru atom H. Interaksi polar dengan residu lain, air dan ligan heme ditunjukkan sebagai garis putus-putus beserta jarak antar keduanya.