(Cucumis sativus L.) menggunakan Metode RAPD
advertisement

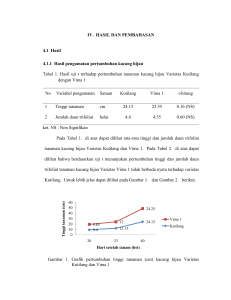
BIODIVERSITAS Volume 9, Nomor 2 Halaman: 99-102 ISSN: 1412-033X April 2008 DOI: 10.13057/biodiv/d090205 Analisis Kekerabatan Mentimun (Cucumis sativus L.) menggunakan Metode RAPD-PCR dan Isozim Cucumber (Cucumis sativus L.) relationship analysis using RAPD-PCR and isozyme methods NUR INDAH JULISANIAH1,♥, LILIEK SULISTYOWATI2, ARIFIN NOOR SUGIHARTO2 1 Jurusan Biologi FMIPA Universitas Mataram (UNRAM), Mataram 83125. 2 Fakultas Pertanian Universitas Brawijaya (UNIBRAW), Malang 65145. Diterima: 19 Desember 2007. Disetujui: 24 Maret 2008. ABSTRACT RAPD-PCR method and isozyme analysis were used to obtain information of genetic relationship among cucumber varieties. Such information is urgently utilized to support plant breeding program of cucumber. Research was done at the Biotechnology Laboratory of Plant Pest and Diseases, Faculty of Agriculture of Brawijaya University, Malang and Molecular Biology Laboratory, Faculty of Matemathic and Natural Sciences of Brawijaya University, Malang. DNA isolation was done using CTAB method by additional NaCl modification. Sixteen primers from operon were employed to amplify DNA genome by RAPD-PCR. Two enzymes, Esterase and AAT were chosen for isozyme analysis. Clad 97 Program was used for analyzing the data and results in data grouping based on proximity value. Cluster analysis based on isozyme data indicated that there was an adequate lower genetic variation in cucumber, where seven of nine tested varieties showed proximity value of 1.00. Eleven of sixteen primers in RAPD-PCR analysis produced DNA bands. Relativity analysis by using RAPD-PCR method showed high enough of genetic variation. Relativity analysis by using both methods showed that variety 07 was the furthermost. The proximity value between varieties 01 and 02 was 0.916667, these varieties have the higest proximity value among all varieties. © 2008 Jurusan Biologi FMIPA UNS Surakarta Key words: Cucumis sativus L., RAPD-PCR, isozyme. PENDAHULUAN Mentimun (Cucumis sativus L.) merupakan salah satu jenis sayur yang cukup populer di hampir semua negara. Mentimun berasal dari dataran tinggi Himalaya dan pada saat ini budidayanya sudah meluas ke seluruh wilayah tropis dan subtropis. Di Indonesia mentimun banyak ditanam di Jawa dan Sumatra (Elsya, 2003). Buah mentimun mengandung mineral seperti kalsium, fosfor, kalium, dan besi, serta vitamin A, B, dan C. Kemajuan di bidang teknologi kecantikan mengungkap bahwa mentimun dapat dimanfaatkan sebagai bahan kosmetika untuk perawatan kecantikan dengan diolah menggunakan teknologi modern. Dari sudut pandang ekonomi, mentimun memiliki prospek yang cukup baik, karena diminati di banyak negara. Mentimun di Indonesia memiliki cukup banyak varietas. Beberapa macam penanda yang dapat digunakan untuk membedakan varietas antara lain: morfologi tanaman, pola pita isozim, dan pola pita DNA. Karakter morfologi seringkali dipengaruhi oleh perubahan lingkungan. Pengamatan morfologi juga harus memperhatikan umur tanaman, karena perubahan umur mempengaruhi perubahan morfologi. Sifat genetik cenderung stabil terhadap perubahan lingkungan, dan tidak dipengaruhi oleh umur, sehingga penanda genetik dapat memberikan informasi yang relatif lebih akurat ♥ Alamat korespondensi: Jl. Majapahit 62, Mataram 83125. Tel. +62-370-631166, 633007. Fax.: +62-370-636041 e-mail: [email protected] (Pratamaningtyas, 1997; Sukartini, 2001). Metode RAPD-PCR merupakan kombinasi teknik PCR menggunakan primer-primer dengan sekuen acak untuk keperluan amplifikasi lokus acak dari genom (Rafalski et al., 1991). Metode ini mempunyai keunggulan pada kesederhanaan tekniknya dan cepat pengerjaannya (Hu dan Quiros, 1991), sehingga RAPD layak digunakan dalam suatu analisis yang menggunakan jumlah sampel cukup besar dan dimanfaatkan dalam upaya pemuliaan tanaman, genetika populasi, dan studi biodiversitas (Rafalski et al., 1991; Waugh dan Powell,1992; Yang dan Quiros, 1993). Penggunaan penanda RAPD akan menguntungkan industri benih karena dapat meningkatkan efisiensi identifikasi kultivar dan menurunkan biaya (Horejsi dan Staub, 1998). Penanda isozim juga dapat digunakan dalam analisis keragaman genetik karena dikendalikan oleh gen tunggal dan bersifat kodominan dalam pewarisannya. Kelebihannya adalah mudah dilakukan dan membutuhkan bahan dalam jumlah sedikit. Metode isozim telah banyak dimanfaatkan oleh pemulia tanaman untuk mengidentifikasi varietas. Wardani (1999) melakukan analisis isozim esterase dan peroksidase untuk memilih tetua tanaman tebu yang berpotensi produksi tinggi. Di Indonesia, Rahayu dan Hartana (2002) meneliti kekerabatan marga Cucurbitaceae di Jawa menggunakan pewarna isozim aspartat amino transferase (AAT), endopeptidase (ENP), asam fosfatase (ACP), dan peroksidase (PRX). Informasi hubungan genetik antara individu di dalam dan di antara spesies mempunyai kegunaan penting bagi perbaikan tanaman. Dalam program pemuliaan tanaman, 100 B I O D I V E R S I T A S Vol. 9, No. 2, April 2008, hal. 99-102 pendugaan hubungan genetik sangat berguna untuk mengelola plasma nutfah, identifiksi kultifar, membantu seleksi tetua untuk persilangan, serta mengurangi jumlah individu yang dibutuhkan untuk pengambilan sampel dengan kisaran keragaman genetik yang luas (Thorman et al., 1994). Staub dan Ivandic (2000) telah melakukan analisis genetik terhadap koleksi mentimun di Amerika Serikat mengguna-kan metode RAPD dan isozim untuk mendeterminasi struktur populasi dari koleksi yang ada. Analisis hubungan kekerabatan secara molekuler dapat memberikan informasi genetik tetua yang akan dipilih dalam persilangan, sehingga bermanfaat untuk budidaya mentimun, antara lain untuk perakitan varietas unggul. BAHAN DAN METODE Tempat dan waktu penelitian Penelitian ini dilaksanakan di Laboratorium Bioteknologi, Jurusan Hama dan Penyakit Tumbuhan, Fakultas Pertanian, Universitas Brawijaya (Unibraw) dan Laboratorium Biologi Molekuler Fakultas MIPA Unibraw Malang. Penelitian dilaksanakan dari bulan Juli 2003 s.d. September 2004. Alat dan bahan Sampel yang digunakan dalam penelitian ini adalah daun tanaman mentimun (Cucumis sativus L.) yang benihnya diperoleh dari sebuah perusahaan benih di Jawa Timur. Sampel yang diteliti sebanyak sembilan varietas, dengan kode: 01, 02, 03, 04, 05, 06, 07, 08, 09. Bahanbahan yang dibutuhkan dalam analisis DNA total dengan metode RAPD-PCR adalah: CTAB, EDTA, Tris-HCL, PVP, akuades steril, mercaptoethanol, NaCl, nitrogen cair, khloroform, isoamil alcohol, etanol absolut, alkohol 70%, PCR buffer reaction, dNTP mix, Taq DNA polymerase, MgCl2, dan primer. Primer yang digunakan sebanyak 16 macam. Sebanyak 1 kb Ladder Gibco, BRL digunakan sebagai penanda, dengan buffer TAE (tris acetic acid EDTA) dan TE. Bahan-bahan yang dibutuhkan dalam analisis isozim adalah: akuades, asam borak, borak, sukrosa, asam askorbit, sistein, Tris-HCl, TEMED (N,N,N’,N’ tetramethylethylenediamine), APS (ammonium persulphate), akrilamid, bisakrilamid, tris-base, gliserol, aseton, α-naphthyl asetat, β-naphthyl asetat, Na-Phospate, sodium malate, 0,2 M buffer phospat pH 6,5, garam Fast Blue RR salt. Selain itu digunakan juga α-ketoglutaric acid, L-aspartic acid, PVP-40, EDTA, sodium phosphate dibasic, dan glisin. Analisis RAPD-PCR Isolasi DNA total. Sampel diambil dari daun segar, diekstrak dengan menambahkan bufer ekstraksi (100 mM Tris-HCl, 20 mM EDTA, 1,4 M NaCl, 2% CTAB, 1% PVP, 1% mercapto-ethanol). Selanjutnya diinkubasi pada suhu 65oC. Sampel disentrifugasi pada kecepatan 7600 rpm. Supernatan ditambahkan larutan CI, selanjutnya disentrifugasi pada 10.000 rpm. Supernatan ditambahkan NaCl hangat, diinversi selama 5 menit, dan ditambahkan lagi CI. Supernatan dipisahkan dengan sentrifugasi pada 10.000 rpm kemudian ditambah dengan etanol absolut dingin, dan diinkubasi pada suhu ruang. Larutan disentrifugasi pada 13.000 rpm. Pelet dicuci dua kali dengan alkohol 70%. Pelet dikeringanginkan, lalu ditambah buffer TE pH 8. Amplifikasi DNA. Amplifikasi dilakukan pada kondisi: 3 menit pada 95oC untuk denaturasi awal, denaturasi selama 45 detik pada 94oC, annealing primer selama 45 detik pada o o 35 C, 90 detik pada 72 C untuk elongasi, ketiga tahap terakhir dilakukan 39 kali siklus. Dilanjutkan pada tahap o elongasi akhir selama 10 menit pada 72 C. tahap terakhir o adalah perendaman pada 4 C selama 60 menit. Reaksi amplifikasi dilakukan pada volume sampel 25 µL, dengan komposisi sebagai berikut: 8 mM dNTP mix, PCR buffer 10x, MgCl2, Taq DNA polymerase, DNA template, primer, ditambah akuabides hingga volume mencapai 25 µL. Hasil amplifikasi divisualisasi menggunakan elektroforesis horisontal dengan gel agarosa 1%. Gel agarosa diberi EtBr yang mampu melakukan khelat dengan DNA, sehingga dapat dilihat di bawah sinar ultraviolet (Toha, 2001). Analisis isozim Satu bagian sampel (g) dilumatkan dan dilarutkan dalam empat bagian buffer sampel (mL). Sampel disentrifugasi dengan kecepatan 8500 rpm selama 20 menit pada suhu 4oC untuk mendapatkan supernatan. Supernatan dielektroforesis selama 180 menit. Running buffer yang digunakan adalah larutan tris-glisin pH 8,3. Gel yang telah dielektroforesis, selanjutnya direndam dalam pewarna isozim esterase yang terdiri atas α-napthyl asetat, β-napthyl asetat aseton, buffer Na-phosphate pH 6,0, dan fast blue RR salt (Brewer dan Sing, 1970). Larutan pewarna untuk mendeteksi adanya enzim AAT terdiri atas α-ketoglutaric acid, L-aspartic acid, PVP 40, EDTA Na salt, sodium phosphate dibasic, fast blue RR salt, dan H2O. Gel yang telah dielektroforesis, diinkubasi dalam larutan pewarna. Setelah terbentuk pita, gel dicuci menggunakan akuades steril. Seluruh gel yang telah diwarnai selanjutnya difiksasi. Analisis data Pola pita DNA hasil amplifikasi diterjemahkan dalam data biner, demikian pula pada analisis isozim, data biner ditentukan berdasarkan kemunculan pita. Selanjutnya data ini digunakan untuk menghitung koefisien similaritas (persamaan) dan menyusun dendogram. Koefisien persamaan antar aksesi dihitung menggunakan rumus Nei dan Li (1979). Analisis data menggunakan program Clad 97. Koefisien persamaan menggunakan skala nilai 0,00 s.d. 1,00. Nilai 1,00 menunjukkan nilai persamaan 100% yang berarti juga nilai jarak genetiknya 0,00. Pengelompokan kluster ditampilkan dalam suatu dendogram. Hubungan kekerabatan antar varietas C. sativus ditentukan oleh nilai persamaan yang dihubungkan dalam diagram fenetik. HASIL DAN PEMBAHASAN RAPD-PCR Isolasi DNA pada penelitian ini dilakukan menggunakan metode buffer CTAB yang dimodifikasi dengan penambahan NaCl, metode ini menghasilkan fragmen tunggal genom yang relatif sama. Dari 16 primer yang digunakan, ditemukan 11 primer yang menghasilkan polimorfisme cukup jelas untuk analisis RAPD pada komoditas C. sativus, yaitu primer: OPO-01, OPO-04, OPO-05, OPO-10, OPO-11, OPO-13, OPO-14, OPO-15, OPO-16, OPO-18, OPO-20. Menurut Waugh dan Powel (1992) primer yang banyak digunakan dalam analisis molekuler tanaman adalah yang terdiri 9-10 nukleutida. Primer-primer yang digunakan dalam penelitian ini memiliki proporsi GC% antara 60-70%. Kondisi ini sesuai dengan pernyataan Newton dan Graham (1994) bahwa jika mungkin menggunakan primer dengan susunan keempat basa yang jumlahnya seimbang, sedangkan yang sesuai untuk eukariot yaitu proporsi GC sebesar 60%. JULISANIAH dkk. – Kekerabatan Cucumis sativus dengan RAPD-PCR dan isozim 101 ini sangat rendah. Hasil analisis kekerabatan dengan isozim esterase dan AAT menunjukkan bahwa variasi genetik pada sembilan varietas yang diamati relatif rendah, terutama pada varietas 01, 02, 03, 04, 05, 06, dan 08, dimana pola pita yang muncul sama persis. Gambar 3. Hasil amplifikasi DNA sembilan varietas C. sativus menggunakan primer OPO-06: L. 1Kb Ladder; 01. var. 01; 02. var. 02; 03. var. 03; 04. var. 04; 05. var. 05; 06. var. 06; 07. var. 07; 08. var. 08; 09. var. 09. Hasil analisis kekerabatan sembilan varietas mentimun menggunakan pendekatan RAPD-PCR menunjuk-kan adanya jarak dan variasi genetik antar varietas. Sampel yang memiliki hubungan kekerabatan paling dekat adalah varietas 02 dan 03. Varietas 02, 03, 05, 07, dan 08 membentuk suatu kelompok. Varietas 01 dan 04 membentuk kelompok sendiri dan memiliki kedekatan dengan varietas 06, sedang varietas 09 terpisah dari kedua kelompok di atas, artinya varietas 09 memiliki hubungan kekerabatan paling jauh dengan varietas-varietas yang lain. Analisis dengan metode ini menunjukkan adanya variasi genetik yang cukup tinggi pada sembilan varietas yang diuji. Isozim Pola pita isozim esterase pada C. sativus yang dihasilkan dari penelitian ini tampak pada Gambar 1. Jarak migrasi setiap pita dapat dihitung dengan rumus Rf (relative ferguson) yaitu: dengan membandingkan jarak pita yang terbentuk dari sumuran dengan jarak migrasi terjauh (Hames dan Rickwood, 1990). Analisis terhadap isozim esterase menghasilkan tiga pola pita yang berbeda. Pola pertama tampak pada varietas 01, 02, 03, 04, 05, 06, dan 08, yaitu muncul tiga pita pada Rf 0,0875; 0,4375; 0,4625. Pola kedua tampak pada varietas 07. Pola ini memiliki kemiripan dengan pola pertama yaitu adanya pita pada Rf 0,0875; 0,4375; 0,4625, tetapi terdapat tambahan dua pita pada Rf 0,8125 dan 0,8375. Pola ketiga tampak pada varietas 09, pola ini sangat berbeda dengan varietas lain, dimana terdapat dua pita yang berada pada posisi Rf 0,3875 dan 0,5000. Varietas 01, 02, 03, 04, 05, 06, dan 08 tampak sama, sehingga diduga di antara varietas -varietas tersebut tidak terdapat variasi genetik. Sedangkan varietas 07 memiliki perbedaan walaupun masih terdapat kemiripan pada pola pita yang dihasilkan, dapat dinyatakan varietas ini memiliki hubungan kekerabatan yang relatif dekat dengan tujuh varietas di atas. Varietas yang tampak memiliki hubungan terjauh di antara kesembilan varietas yang diuji adalah varietas 09. Hasil analisis kekerabatan dengan isozim esterase ini, menunjukkan variasi genetik yang cukup rendah pada sembilan varietas C. sativus . Pola pita isozim aspartat amino transferase (AAT) pada sembilan varietas C. sativus pada penelitian ini tampak pada Gambar 2. Analisis terhadap isozim AAT menghasilkan dua pola pita yang berbeda dan satu varietas tidak memiliki pita. Pola pita pertama tampak pada varietas 01, 02, 03, 04, 05, 06, dan 08, yaitu muncul enam pita pada Rf 0,1500; 0,1625; 0,1875; 0,2125; 0,2625; 0,3250 (Gambar 2.). Pola pita kedua tampak pada varietas 09. Pola pita ini memiliki kemiripan dengan pola pita pertama, yaitu: adanya pita pada Rf 0,1500; 0,1625; 0,1875, tetapi pita selanjutnya tidak ditemukan, sedangkan pada varietas 07 tidak muncul pita, dimungkinkan kandungan enzim AAT dalam varietas Gambar 1. Pola pita isozim esterase C. sativus: 1. var. 01; 2. var. 02; 3. var. 03; 4. var. 04; 5. var. 05; 6. var. 06; 7. var. 07; 8. var. 08; 9. var. 09. Gambar 2. Pola pita isozim AAT C. Sativus: 1. var. 01; 2. var. 02; 3. var. 03; 4.var. 04; 5.var. 05; 6. var. 06; 7. var. 07; 8. var. 08; 9. var. 09. Penggabungan data hasil analisis RAPD-PCR dan isozim menunjukkan bahwa variasi genetik di antara sembilan varietas C. sativus yang diuji cukup tinggi. Adanya perbedaan hasil antara metode RAPD-PCR dan analisis isozim dapat diterima dengan penjelasan bahwa metode RAPD-PCR menganalisis pada tingkatan DNA, dimana DNA sebagai cetak biru makhluk hidup merupakan materi genetik yang membawa informasi genetik induk kepada keturunannya. Semua makhluk hidup memiliki materi genetik untuk mempertahankan kelangsungan struktur, sifat, fungsi, dan aktivitas kimia dalam sel (Toha, 2001), sehingga DNA merupakan bahan dasar keturunan atau substansi dari gen (Kimball, 1994). Isozim merupakan molekul multiple suatu enzim yang dijumpai baik dalam satu individu atau pada anggota yang berbeda dari satu spesies (Sofro, 1994). Wirahadikusumah (2001) menjelaskan enzim sebagai golongan protein dalam jumlah paling banyak di dalam sel, mempunyai fungsi sebagai katalisator reaksi biokimia. Protein itu sendiri merupakan ekspresi dari DNA dimana proses sintesisnya melalui penerjemahan kodonkodon pada mRNA menjadi polipep-tida, dan setiap kodon tersusun atas basa-basa nukleotida. Analisis kekerabatan Diagram fenetik yang diperoleh dari analisis ini (Gambar 3.), menunjukkan bahwa varietas 01 dan 02 memiliki hubungan paling dekat di antara sembilan varietas yang diuji. Nilai kedekatan keduanya adalah 0,916667 atau 91,67%. Varietas 03 dekat dengan varietas 08 pada nilai 0,86111. Varietas 05 memiliki kedekatan dengan kelompok varietas 01 dan 02 pada nilai 0,847222. Kelompok ini dekat dengan varietas 04 dengan nilai kedekatan 0,824074. Selanjutnya kelompok yang terbentuk dari varietas 01, 02, 04, 05, memiliki kedekatan 78,24% dengan kelompok 102 B I O D I V E R S I T A S Vol. 9, No. 2, April 2008, hal. 99-102 varietas 03 dan 08. Varietas 06 memiliki kedekatan dengan kelompok di atas sebesar 0,709105. Sedangkan varietas 09 memiliki nilai kedekatan 0,527263. Varietas yang terjauh hubungan kekerabatannya adalah varietas 07 yang memiliki nilai kedekatan 0,486625. Berdasarkan Gambar 3, varietas terjauh adalah varietas 07. Di sini tampak bahwa data yang diperoleh dari hasil analisis isozim memberikan kontribusi yang cukup besar pada penyusunan diagram fenetik. Hal ini dimungkinkan karena hasil dari analisis isozim AAT menunjukkan tidak adanya pita sama sekali pada varietas 07, dalam perhitungan nilai similaritas dan penyusunan diagram phenethyc cukup berpengaruh karena jumlah pita yang dihasilkan pewarnaan isozim AAT relatif besar, yaitu enam pita. Analisis kekerabatan ini menunjukkan adanya variasi genetik yang cukup tinggi pada sembilan varietas yang diuji. Variasi genetik yang muncul, diduga dipengaruhi oleh asal tetua. Varites-varietas yang memiliki kedekatan genetik, diduga beasal dari tetua yang berkerabat dekat, sebaliknya varietas -varietas yang jarak genetiknya relatif tinggi, diduga berasal dari tetua yang jauh hubungan kekerabatannya dengan tetua varietas yang lain. yang lebih akurat. Perbedaan hasil dari metode RAPD-PCR dan isozim ini, dimungkinkan juga karena sedikitnya jumlah isozim yang digunakan sebagai penanda, sehingga hasil yang diperoleh kurang beragam. KESIMPULAN DAN SARAN Analisis kekerabatan C. sativus dengan penanda RAPD-PCR dan isozim menunjukkan bahwa varietas terjauh adalah varietas 07, sedangkan yang memiliki nilai kedekatan paling tinggi adalah varietas 01 dan 02 dengan nilai kedekatan 0,916667. Analisis kekerabatan ini menunjukkan adanya variasi genetik yang cukup tinggi pada sembilan varietas yang diuji. Berdasarkan hasil penelitian, maka disarankan untuk dilakukan penelitian serupa dengan jumlah varietas, jumlah primer dan jumlah isozim yang lebih banyak. UCAPAN TERIMA KASIH Penulis mengucapkan terimakasih kepada: P.T. BISI, Kediri, Jawa Timur, serta Dr. Aulanni’am dan Dr. Sri Widyarti dari Universitas Brawijaya, Malang. DAFTAR PUSTAKA Gambar 3. Diagram fenetik (genetic identity) sembilan varietas C. sativus menggunakan metode RAPD-PCR serta analisis isozim esterase dan AAT. Hasil di atas dapat digunakan sebagai acuan dalam penentuan induk untuk pembuatan bibit. Semakin jauh hubungan kekerabatan antar sampel, maka semakin kecil keberhasilan persilangan, tetapi kemungkinan untuk memperoleh genotip unggul lebih besar jika persilangan berhasil. Semakin beragam genetik, maka semakin besar kemungkinan diperoleh genotipe unggul. Perkawinan antara individu berjarak genetik dekat atau hubungan kekerabatannya sama mempunyai efek pening-katan homozigositas, sebaliknya perkawinan antara individu berjarak genetik besar atau kekerabatannya jauh mempunyai efek peningkatan heterozigositas. Informasi ini berdampak baik bagi proses pembuatan bibit unggul. Perkawinan tetua dengan variasi genetik yang relatif tinggi akan menghasilkan individu dengan heterozigositas lebih tinggi. Penggunaan dua metode dalam analisis kekerabatan mentimun ini, juga memberikan informasi bahwa analisis isozim lebih efisien dari segi biaya dan waktu pengerjaan serta metode yang dilakukan cukup sederhana, sedangkan keuntungan metode RAPD-PCR adalah diperoleh informasi Brewer, G.J. and C.F. Sing. 1970. An Introduction to Isozymes Techniques. New York: Academic Press. Elsya, T. 2003. Mentimun, Obat Awet Muda dan Antistres. Artikel. Pikiran Rakyat Cyber Media. Minggu, 06 Juli 2003. Hames. B.D. dan D. Rickwood. 1990. Gel Electrophoresis of Proteins. A Practical Approach. 2nd ed. Oxford: Oxford University Press. Horejsi, T. and J.E. Staub. 1998. Genetic variation in cucumber (Cucumis sativus L) as assessed by random amplified polymorphic DNA. Abstrak TEKTRAN. Washington D.C.: United States Department of Agriculture, Agricultural Research Service, USA. Hu, J. and G.F. Quiros. 1991. Identification of broccoli and cauliflower cultivars with RAPD marker. Plant Cell Reports 10: 505-511. Kimball, J.W. 1994. Biologi. Jilid I. Penerjemah: Sutarmi, S. dan N. Sugiri. Jakarta: Penerbit Erlangga. Nei, M. and W. Li. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceeding of National Academic of Science, USA. 76: 5269-5273 Newton, C.R. and Graham. 1994. PCR (Introduction to Biotechniques Series). Oxford: Bios Scientific Publishers. Pratamaningtyas, S. 1997. Optimasi Kondisi Polymerase Chain Reaction Untuk Analisis Random Amplyfied Polymorphic DNA’s Pada Genom Tebu. [Tesis]. Malang: Program Pascasarjana Universitas Brawijaya. Rafalski, J.A., S.V. Tingey, and J.G.K. Williams. 1991. RAPD markers-a new technology for genetic mapping and plat breeding. AgBitech News and Information 3: 645-648. Rahayu, S.E., dan A. Hartana 2002. Biosistematika Cucumis (Cucurbitaceae) di Jawa. Floribunda 2: 38-43. Sofro. A.S.M. 1994. Keanekaragaman Genetik. Yogyakarta: Andi Offset. Staub, J.E. and V. Ivandic. 2000. Genetic assessment of the United States National cucumber collection. In: ISHS Acta Horticulture 510: Eucarpia Meeting on Cucurbit Genetics and Breeding. Leuven, Belgium: International Society for Horticultural Science. Sukartini. 2001. Analisis Jarak Genetik dan Hubungan Kekerabatan Pisang (Musa spp) menggunakan Penanda Morfologis dan Random Amplified Polymorphic DNA. [Tesis]. Malang: Program Pascasarjana Universitas Brawijaya. Thorman, C.E., M.E. Ferreira, L.E.A. Camargo, J.G. Tivang, and T.C. Osborn. 1994. Comparison of RFLP and RAPD markers to estimating genetic relationship within and among cruciferous spesies. Theoritical and Applied Genetic 88: 973-980. Toha. A.H.A. 2001. Deoxyribo Nucleac Acid: Keanekaragaman, Ekspresi, Rekayasa, dan Efek Pemanfaatannya. Seri Belajar Biokimia. Bandung: Alfabeta. Wardani. N.M.R. 1999. Pembandingan Penanda Morfologi dan Isozim untuk Pemilihan Tetua Tanaman Tebu yang Berpotensi Produksi Tinggi. [Tesis]. Malang: Program Pascasarjana Universitas Brawijaya. Waugh, R. and W. Powell. 1992. Using RAPD markers for crop improvement. TIBTECH. 10: 186-191. Wirahadikusumah, M. 2001. Biokimia Protein Enzim dan Asam Nukleat. Bandung: Penerbit ITB. Yang, X. and C. Quiros. 1993. Identification and classification of celery cultivars with RAPD markers. Theorical and Applied Genetics 86: 205-212.