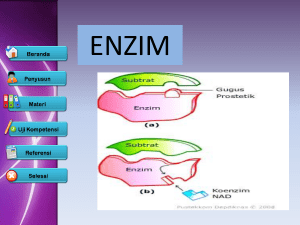
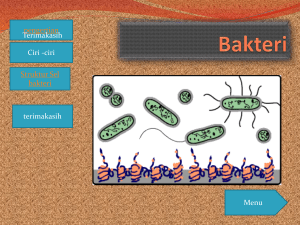
deteksi molekuler komunitas bakteri dari perairan pulau pari
advertisement

DETEKSI MOLEKULER KOMUNITAS BAKTERI DARI PERAIRAN PULAU PARI TELUK JAKARTA YANG TERCEMAR MINYAK ARIANI HATMANTI SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR BOGOR 2011 PERNYATAAN MENGENAI TESIS DAN SUMBER INFORMASI Dengan ini saya menyatakan bahwa tesis berjudul “Deteksi Molekuler Komunitas Bakteri dari Perairan Pulau Pari Teluk Jakarta yang Tercemar Minyak” ini merupakan hasil karya saya sendiri dengan arahan dan masukan dari tim komisi pembimbing. Tesis ini belum diajukan dalam bentuk apapun kepada Perguruan Tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini. Bogor, Agustus 2011 Ariani Hatmanti NRP P051080051 ABSTRACT ARIANI HATMANTI. Molecular Detection of Bacterial Community in Oilcontaminated Seawaters of Pari Island, Jakarta Bay. Under direction of ANJA MERYANDINI and ARIS TJAHJOLEKSONO A research on molecular detection of bacterial community was done in Pari Island Jakarta Bay from January 2010 – June 2011. This study was aimed to understand bacterial community and the succesion of bacteria in oilcontaminated seawater. This research was also conducted to know the domination of exogenous bacteria in the substrates and their effect on crude oil degradation. Twenty-eight samples or substrates were analyzed using Denaturing Gradient Gel Electrophoresis (DGGE). The substrates were mixture of oil-contaminated seawater and sediment. The results of this study showed that marine bacterium SCRIPP 413, uncultured bacterium clone VH-FL6-50 and uncultured bacterium clone W1-16 were dominant in the bacterial community in Pari Island contaminated environment. This study also indicated that the exogenous bacteria were dominant in absence of indigenous bacteria. Keywords: bacterial community, marine bacteria, DGGE, oil-contaminated seawater RINGKASAN ARIANI HATMANTI. Deteksi Molekuler Komunitas Bakteri dari Perairan Pulau Pari Teluk Jakarta yang Tercemar Minyak. Dibimbing oleh ANJA MERYANDINI and ARIS TJAHJOLEKSONO Pada umumnya pencemaran minyak bumi dapat ditanggulangi dengan menggunakan teknik fisika dan kimia. Cara penanggulangan tersebut masih menyisakan cemaran minyak bumi di perairan maupun sedimen di sekitarnya sehingga masih berpotensi mencemari lingkungan. Penanganan sisa bahanbahan cemaran ini biasanya dilakukan dengan menggunakan teknik-teknik bioremediasi, yaitu penggunaan agen biologi, termasuk mikroorganisme untuk menghilangkan limbah atau buangan yang bersifat toksik dari lingkungan. Bioremediasi dapat dilakukan berdasarkan pendekatan bioaugmentasi maupun biostimulasi. Dalam aplikasi biostimulasi maupun bioaugmentasi, perlu dipelajari ketersediaan bakteri indigenous dan struktur komunitas mikroba pada perairan tersebut agar upaya bioremediasi dapat dilakukan secara lebih optimal dan berdaya guna. Penelitian ini bertujuan untuk mendeteksi struktur komunitas dan suksesi bakteri pendegradasi minyak, serta dominansi bakteri eksogenous terhadap populasi bakteri yang terdapat di dalam media yang tercemar minyak. Bakteri yang digunakan di dalam penelitian ini adalah strain RCO/B/08_006, RCO/B/08_008, RCO/B/08_009, dan RCO/B/08_013. Kelima strain tersebut telah diidentifikasi pada penelitian sebelumnya sebagai bakteri potensial pendegradasi minyak dan Poly-aromatics Hydrocarbon (PAH). Substrat yang digunakan dalam penelitian ini adalah sampel air laut dan sedimen dari perairan Pulau Pari Teluk Jakarta yang dicampur untuk menggambarkan kondisi alam yang mendekati sebenarnya. Sebelum dicampur dengan air laut, sampel sedimen yang diambil dari beberapa titik di perairan Pulau Pari Teluk Jakarta dikomposit terlebih dahulu, kemudian didistribusikan ke dalam tabung-tabung dan ditambah dengan waterair laut dengan perbandingan 1:1. Pada penelitian ini digunakan 84 tabung (28 x 3 ulangan) yang terdiri atas: 4x3 tabung disterilisasi dan ditambah bakteri RCO/B/08_008, 4x3 tabung disterilisasi dan ditambah konsorsium A (Strain RCO/B/08_006, RCO/B/08_008, dan RCO/B/08_013), 4x3 tabung tidak disterilisasi dan ditambah bakteri RCO/B/08_008, 4 tabung tidak disterilisasi dan ditambah konsorsium A, 8x3 tabung tidak disterilisasi dan tidak ditambah bakteri, serta 4x3 tabung disterilisasi tanpa ditambah bakteri. Substrat dalam tabung-tabung tersebut diinkubasikan selama 0, 7, 14 dan 28 hari. Setelah masa inkubasi, masing-masing substrat dalam tabung disimpan pada suhu 80oC. Bahan yang digunakan untuk isolasi DNA adalah ISOIL Bead-beating DNA Extraction Kit dan Applied Biosystems Prepman Ultra Sample Preparation Reagent. Bahan yang digunakan untuk PCR dan elektroforesis meliputi HotstarTag PCR Mix (QIAGEN), Primer 341F (5’-CCTACGGGAGGCAGCAG-3’), Primer 907R (5’-CCGTCAATTCMTTTGAGTT T-3’), Primer 341F-GC (40pb GC clamp 5’-CCTACGGGAGGCAGCAG-3’), MilliQ PCR grade, Etanol 70%, QIAquick PCR purification kit (QIAGEN), Urea, Formamida, Akrilamida/bisakrilamida, Ammonium Persulphate (APS), TEMED, Agarosa, Akuades, TAE Buffer 1x, Dye-solution, Gel-loading solution, Etidium Bromida, Etanol 90%, dan air murni. Hasil analisis DGGE menunjukkan bahwa komunitas bakteri dalam lingkungan tercemar di perairan Pulau Pari Teluk Jakarta didominasi oleh marine bacterium SCRIPP 413, uncultured bacterium clone VH-FL6-50 dan uncultured bacterium clone W1-16. Beberapa jenis bakteri lainnya terdeteksi pada hari ke-7 dan 14, namun kemudian tidak terdeteksi pada hari ke-28. Dominansi bakteri eksogenous yaitu strain RCO/B/08_006, RCO/B/08_008, dan RCO/B/08_013 terdeteksi hanya di dalam substrat yang tidak mengandung bakteri indigenous (substrat steril). Namun, pada substrat yang mengandung bakteri indigenous (substrat tidak steril), bakteri eksogenous tidak terdeteksi. Hal ini mengindikasikan bahwa bakteri eksogenous yang ditambahkan tidak kompetitif. Kata kunci: komunitas bakteri, bakteri laut, DGGE, pencemaran laut © Hak Cipta milik IPB, 2011 Hak Cipta dilindungi Undang-undang Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB. Dilarang mengumumkan dan memperbanyak sebagian atau seluruh Karya Tulis ini dalam bentuk apapun tanpa izin IPB. DETEKSI MOLEKULER KOMUNITAS BAKTERI DARI PERAIRAN PULAU PARI TELUK JAKARTA YANG TERCEMAR MINYAK ARIANI HATMANTI Tesis Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada Program Studi Bioteknologi SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR BOGOR 2011 Penguji Luar Komisi pada Ujian Tesis : Dr. Ir. Iman Rusmana, MSi. Judul Tesis : Nama NRP : : Deteksi Molekuler Komunitas Bakteri dari Perairan Pulau Pari Teluk Jakarta yang Tercemar Minyak Ariani Hatmanti P051080051 Disetujui, Komisi Pembimbing Dr. Anja Meryandini, M.S Ketua Dr. Ir. Aris Tjahjoleksono, DEA Anggota Diketahui, Ketua Program Studi Bioteknologi SPs IPB, Dekan Sekolah Pascasarjana IPB Prof. Dr. Ir. Suharsono, DEA. Dr. Ir. Dahrul Syah, M.Sc. Agr. Tanggal Ujian: 29 Juli 2011 Tanggal Lulus: HALAMAN PERSEMBAHAN KARYA ILMIAH ini penulis persembahkan kepada : Bapak dan Ibu.. Yang tanpa beliau berdua tak akan Ariani Hatmanti menjadi seperti saat ini.. Terimakasih yang sebesar-besarnya atas segala bimbingan, arahan, dorongan, cinta, kasih, doa dan ketulusan yang tak ada duanya.. Maafkan ananda.. yang belum bisa membalasnya sampai kini.. & Anak-anak tercinta : Ardana Zahra Septanti & Ardana Dzaka Septanto Terimakasih banyak atas segala pengertian dan pengorbanannya.. atas segala doa dan semangatnya yang membuat bunda masih kuat bertahan.. hingga kini.. dan nanti.. Semoga Allah SWT masih memberikan kesempatan bagi kita ‘tuk mengganti semua waktu, perhatian dan kebersamaan yang sempat tercabik oleh kesibukan selama ini.. Semoga Allah Subhanahuwata’ala senantiasa meridhoi dan melindungi kita.. Amien yaa rabbalalamiin.. PRAKATA Segala puji dan syukur penulis panjatkan ke hadirat Allah SWT atas limpahan nikmat dan karunia-NYA sehingga karya tulis ilmiah ini berhasil diselesaikan. Judul penelitian yang dilaksanakan sejak Bulan Januari 2010 – Juni 2011 ini adalah “Deteksi Molekuler Komunitas Bakteri dari Perairan Pulau Pari Teluk Jakarta yang Tercemar Minyak”. Pelaksanaan penelitian dan penulisan karya ilmiah ini tidak lepas dari bantuan banyak pihak, karena itu penulis mengucapkan terimakasih kepada: 1. Dr. Anja Meryandini, M.S dan Dr. Ir. Aris Tjahjoleksono, DEA, selaku pembimbing, yang telah memberikan segenap waktu, pikiran dan tenaganya untuk membimbing dan mengarahkan penulis baik selama penelitian maupun dalam penulisan tesis. 2. Dr. Ir. Iman Rusmana, MSi. selaku penguji luar komisi yang telah menguji, menelaah dan memberikan masukan demi kesempurnaan tesis ini. 3. Prof. Dr. Ir. Suharsono, DEA. selaku Ketua Program Studi Bioteknologi SPs IPB yang telah mengarahkan dan memberi semangat penulis selama melaksanakan pendidikan di Program Studi Bioteknologi SPs IPB. 4. Program DIPA Puslit Oseanografi LIPI penelitian bioremediasi pantai berpasir dan proyek kerjasama LIPI – NITE yang telah menyediakan bahan dan alat-alat untuk penelitian ini. 5. Beasiswa Bantuan Penulisan Tesis Program Mitra Bahari-COREMAP II tahun 2010 atas bantuan dana demi kelancaran penulisan tesis ini. 6. Kepala Pusat Penelitian Oseanografi LIPI Prof. Dr. Suharsono, MSc., yang telah memberikan izin belajar bagi penulis. 7. Dr. Zainal Arifin, MSc., Dr. Suhartati M. Natsir, MS., Drs. Ruyitno Nuchsin, MSc., Ir. Yeti Darmayati, MSc., Dra. Lies Indah Sutiknowati, MSi., Ir. Dwi Hindarti, MSc., Drs. Muswerry M, MSc., Drs. Helfinalis, MSc., Drs. Djoko Hadi K, MSc., yang selalu memberikan dorongan materi dan nonmateri bagi penulis. 8. Direktur Research and Development Center Charoen Pokphand Indonesia dan Dr. Fitri Fegatella atas izin dan kerjasamanya dalam penggunaan alat DGGE. Mbak Ekasari Hendra, Aster, dan rekan-rekan di Charoen Pokphand atas kerjasama, diskusi dan supportnya. 9. Dr. Achmad Farajallah dan Wildan atas bantuan penggunaan laboratorium untuk silver stain dan diskusinya. 10. Ibu, Bapak, dan adik-adik penulis Dyah dan Adi beserta keluarganya atas doa, dorongan dan kasih sayang serta bantuannya selama penulis menyelesaikan pendidikan. 11. Anak-anak penulis : Ardana Zahra Septanti dan Ardana Dzaka Septanto, atas doa dan pengorbanannya selama penulis melaksanakan pendidikan. 12. Sahabat-sahabat penulis : Ahmad Riyadi, Nurul Fitriya, Syofia, Nilda, Nila, Nano, Mas Tonthowi, Bu Noor, Rahman, Rachma, Yoni, Afdal, Febri, Yulia, Cipon, Mourly atas doa, dorongan dan semangatnya. 13. Rekan-rekan Prodi Bioteknologi SPs IPB : Etty, Eka, Mbak Rere, Duti, Dedi, dan Pak Zul atas kebersamaan dan semangatnya. 14. Seluruh pihak yang tidak dapat penulis sebutkan satu-persatu, namun besar kontribusinya bagi kelancaran pendidikan penulis hingga selesai. Semoga Karya Ilmiah ini bermanfaat. Bogor, Agustus 2011 Ariani Hatmanti NRP. P051080051 RIWAYAT HIDUP Penulis dilahirkan di Tegal pada tanggal 8 Juli 1976 dari pasangan Bapak Drs. Tjipto Hartono dan Ibu Sri Amini Susilowati, B.A. Penulis merupakan putri sulung dari tiga bersaudara. Penulis lulus dari SMAN 1 Pemalang Jawa Tengah pada tahun 1994 dan pada tahun yang sama diterima sebagai mahasiswa Fakultas Biologi Universitas Jenderal Soedirman melalui Ujian Masuk Perguruan Tinggi Negeri (UMPTN). Pada kesempatan selanjutnya penulis memilih untuk mendalami bidang Biologi Lingkungan, khususnya Mikrobiologi Lingkungan. Setelah menamatkan kesarjanaan pada bulan April 1999, penulis diterima bekerja sebagai staf peneliti di Laboratorium Mikrobiologi Laut Pusat Penelitian Oseanografi LIPI terhitung sejak Bulan Maret 2000, dan sampai sekarang masih mengabdikan diri dalam penelitian-penelitian mikrobiologi laut di puslit tersebut, terutama dalam bidang lingkungan. Pada tahun 2008 penulis diterima sebagai mahasiswa pada Program Studi Bioteknologi Sekolah Pascasarjana IPB, Bogor. Selama mengikuti perkuliahan pada program tersebut, penulis aktif menjadi anggota dalam organisasi Ikatan Sarjana Oseanologi Indonesia (ISOI) dan Perhimpunan Mikrobiologi Indonesia (PERMI) cabang Jakarta. Selain itu penulis juga telah menghasilkan beberapa tulisan ilmiah dan semi populer yang diterbitkan pada jurnal terakreditasi nasional dan jurnal internasional. DAFTAR ISI Halaman DAFTAR TABEL.............................................................................................. xiii DAFTAR GAMBAR.......................................................................................... xiv DAFTAR LAMPIRAN....................................................................................... xv PENDAHULUAN............................................................................................. Latar Belakang............................................................................................ Tujuan Penelitian........................................................................................ 1 1 2 TINJAUAN PUSTAKA..................................................................................... Komposisi Minyak....................................................................................... Pencemaran Minyak................................................................................... Efek Pencemaran Minyak.......................................................................... Definisi Bioremediasi.................................................................................. Bioaugmentasi............................................................................................ Bakteri yang digunakan dalam bioaugmentasi........................................... Hubungan pencemaran minyak dan komunitas bakteri............................. DGGE : definisi dan hubungannya dengan komunitas bakteri................... Prinsip DGGE............................................................................................. 3 3 4 6 7 8 9 10 11 12 BAHAN DAN METODE................................................................................... Waktu dan Tempat Penelitian..................................................................... Materi Penelitian......................................................................................... Metode Penelitian....................................................................................... 15 15 15 16 HASIL DAN PEMBAHASAN............................................................................ Struktur komunitas bakteri indigenous pada lingkungan tercemar minyak di perairan Pulau Pari Teluk Jakarta........................................................... Perubahan struktur komunitas bakteri pada substrat dengan penambahan bakteri eksogenous (tunggal dan konsorsium)..................... Komposisi bakteri yang terdeteksi dalam komunitas bakteri dari perairan Pulau Pari Teluk Jakarta............................................................................. 19 SIMPULAN DAN SARAN................................................................................ Simpulan..................................................................................................... Saran.......................................................................................................... 37 37 37 DAFTAR PUSTAKA........................................................................................ 39 LAMPIRAN...................................................................................................... 46 19 24 32 DAFTAR TABEL Halaman 1. Komposisi Elemental Minyak Bumi.......................................................... 3 2. Matrik sampel yang dianalisis komunitas bakterinya................................ 15 3. Strain bakteri koleksi Laboratorium Mikrobiologi Laut Puslit Oseanografi LIPI yang digunakan dalam penelitian...................................................... 16 Pita bakteri yang terdeteksi pada gel akrilamida (profil DGGE) dalam proses bioremediasi pada substrat tidak steril dan substrat steril tanpa penambahan bakteri (tunggal atau konsorsium)...................................... 25 Pita bakteri yang terdeteksi pada gel akrilamida (profil DGGE) dalam proses bioremediasi pada substrat tidak steril dan substrat steril dengan penambahan bakteri (tunggal atau konsorsium)......................... 33 4. 5. DAFTAR GAMBAR Halaman 1. Prinsip Denaturing Gradient Gel Electrophoresis (DGGE)....................... 13 2. Profil DGGE pada proses bioremediasi secara alamiah dalam substrat tidak steril dan substrat steril tanpa penambahan bakteri (tunggal atau konsorsium).............................................................................................. 20 Hubungan antara total biomassa sel dengan penurunan konsentrasi minyak dalam substrat tidak steril tanpa penambahan strain RCO/B/08_008......................................................................................... 22 Profil DGGE pada proses bioremediasi dalam substrat tidak steril dan substrat steril dengan penambahan bakteri (tunggal atau konsorsium).............................................................................................. 26 Hubungan antara total biomassa sel, penurunan konsentrasi minyak, dan penambahan strain tunggal RCO/B/08_008 ..................................... 28 Hubungan antara total biomassa sel, penurunan konsentrasi minyak, dan penambahan konsorsium A............................................................... 31 Komposisi komunitas bakteri dari Perairan Pulau Pari Teluk Jakarta berdasarkan kemampuan kulturisasi........................................................ 32 Komposisi komunitas bakteri dari Perairan Pulau Pari Teluk Jakarta berdasarkan filum..................................................................................... 34 Komposisi komunitas bakteri dari Perairan Pulau Pari Teluk Jakarta berdasarkan kelas.................................................................................... 35 3. 4. 5. 6. 7. 8. 9. DAFTAR LAMPIRAN Halaman 1. Rekapitulasi hasil sekuensing pita DGGE................................................ 48 2. Data sekuen masing-masing isolat dalam format fasta............................ 49 PENDAHULUAN Latar Belakang Minyak bumi merupakan senyawa yang bersifat rekalsitran sehingga tidak mudah terdegradasi secara alami dalam jangka waktu yang relatif pendek (Nugroho 2006). Pada umumnya pencemaran minyak bumi dapat ditanggulangi dengan menggunakan teknik fisika dan kimia. Cara penanggulangan tersebut masih menyisakan cemaran minyak bumi di perairan maupun sedimen di sekitarnya sehingga masih berpotensi mencemari lingkungan. Penanganan sisa bahan-bahan cemaran ini biasanya menggunakan teknik-teknik bioremediasi. Prince et al. (2003) menyatakan bahwa metode bioremediasi merupakan cara penanggulangan tumpahan minyak yang paling aman bagi lingkungan. Menurut Sudrajat (1996) teknik bioremediasi yaitu pemanfaatan mikroorganisme perombak polutan untuk membersihkan lingkungan yang tercemar. Kemampuan merombak tersebut berkaitan dengan gen-gen penyandi berbagai enzim perombak polutan. Bioremediasi dapat dilakukan melalui dua metode yaitu biostimulasi dan bioaugmentasi. Biostimulasi adalah penggunaan nutrien untuk memacu pertumbuhan dan meningkatkan aktivitas bakteri indigenous, sedangkan bioaugmentasi adalah penambahan bakteri eksogenous ke lingkungan yang tercemar (Barnum 2005). Dalam aplikasi biostimulasi maupun bioaugmentasi, perlu dipelajari ketersediaan bakteri indigenous dan struktur komunitas mikroba pada perairan tersebut, agar upaya bioremediasi dapat dilakukan secara lebih optimal dan berdaya guna. Dari penelitian yang dilakukan sebelumnya di perairan Teluk Jakarta, diperoleh beberapa strain bakteri yang mampu mendegradasi minyak dan Polyaromatics Hydrocarbon (PAH). Di antara strain-strain tersebut adalah: strain RCO/B/08_006, RCO/B/08_008, RCO/B/8_009, strain 68 MRa, dan RCO/B/08_013. Strain RCO/B/08_008 mampu mendegradasi senyawa-senyawa PAH yaitu fluoranthene, fenanthrene, naphtalene, pyrene, phenotiazene, dibenzothiophene, fluorene dan campuran PAH, serta menguraikan arabian light crude oil (ALCO) yang ditambahkan pada substrat agar (Hatmanti dan Darmayati 2009, Darmayati 2009). Darmayati (2009) menyatakan strain RCO/B/08_009 mampu mendegradasi dibenzothiophene, fluorene, fenanthrene, pyrene, fluoranthene, dan campuran PAH serta ALCO. Darmayati et al. (2008) juga menyebutkan bahwa RCO/B/08_008, RCO/B/08_009 dan RCO/B/08_006 dapat dijadikan agen bioremediasi di laut maupun pantai karena mampu mendegradasi dan/atau mengemulsifikasi minyak dengan baik dalam kondisi salinitas 10 – 34 ppt dan pH 6 – 8. Walaupun dipilih secara random, strain 68 MRa dan RCO/B/08_013 mempunyai kemampuan mendegradasi setidaknya 4-5 jenis PAH dari 7 yang diujikan (Hatmanti dan Darmayati 2009). Kelima jenis bakteri potensial ini telah ditapis lebih lanjut untuk agen bioaugmentasi skala laboratorium dalam kultur tunggal oleh Kusuma (2009) serta dalam kultur campuran oleh Arifah (2010). Berdasarkan kedua penelitian tersebut diketahui bahwa setiap bakteri yang diintroduksikan ke dalam substrat, baik secara individu maupun konsorsium, memberikan hasil yang bervariasi dalam menguraikan minyak dan PAH. Hasil dari penelitian Kusuma (2009) menunjukkan strain RCO/B/08_008 memiliki persentase degradasi minyak tertinggi yaitu 89% selama 28 hari, sedangkan penelitian Arifah (2010) menunjukkan konsorsium A (Strain RCO/B/08_006, RCO/B/08_008, dan RCO/B/08_013) mampu menurunkan konsentrasi minyak dan total PAH paling tinggi yaitu secara berturut-turut 71% dan 84%. Untuk melengkapi informasi tentang komunitas mikroba di perairan yang tercemar, perlu dilakukan penelitian lebih lanjut dengan menggunakan teknikteknik molekuler. Salah satu teknik molekuler yang dapat digunakan adalah Denaturing Gradient Gel Electrophoresis (DGGE). Teknik DGGE dapat digunakan untuk memisahkan fragmen DNA yang berukuran sama tetapi sekuen nukleotidanya berbeda. Tujuan Penelitian Penelitian ini dilakukan untuk mengetahui struktur komunitas bakteri indigenous, serta dominansi bakteri eksogenous terhadap komunitas bakteri indigenous yang terdapat di dalam substrat dari perairan Pulau Pari Teluk Jakarta yang tercemar minyak. TINJAUAN PUSTAKA Komposisi Minyak Minyak bumi tersusun atas berbagai jenis senyawa hidrokarbon. Komposisi spesifiknya tergantung dari bentuknya, apakah masih berupa minyak bumi atau telah mengalami destilasi. Proses destilasi dilakukan untuk memisahkan komponen-komponen minyak bumi berdasarkan berat molekul yang berbeda menjadi bermacam-macam produk seperti bensin, solar dan minyak tanah. Tumpahan minyak bumi dari kapal tanker dapat menyebabkan kerusakan lingkungan yang serius dan mempengaruhi kehidupan satwa yang ada di lingkungan yang tercemar (Barnum 2005). Budhiarto (2009) menyatakan minyak bumi memiliki campuran senyawa hidrokarbon sebanyak 50-98% berat, sisanya terdiri atas zat-zat organik yang mengandung belerang, oksigen, dan nitrogen serta senyawa-senyawa anorganik seperti vanadium, nikel, natrium, besi, aluminium, kalsium, dan magnesium. Secara umum, komposisi minyak bumi dapat dilihat pada tabel berikut: Tabel 1 Komposisi Elemental Minyak Bumi Komposisi Karbon (C) Hydrogen (H) Sulfur (S) Nitrogen (N) Oksigen (O) Persentase 84-87 11-14 0-3 0-1 0-2 Berdasarkan kandungan senyawanya, minyak bumi dapat dibagi menjadi golongan hidrokarbon dan non-hidrokarbon serta senyawa-senyawa logam. Golongan hidrokarbon-hidrokarbon yang utama adalah parafin, olefin, naften, dan aromatik. 1. Parafin Parafin adalah kelompok senyawa hidrokarbon jenuh berantai lurus (alkana), CnH2n+2. Contohnya adalah metana (CH4), etana (C2H6), n-butana (C4H10), isobutana (2-metil propana, C4H10), isopentana (2-metilbutana, C5H12), dan isooktana (2,2,4-trimetil pentana, C8H18). Jumlah senyawa yang tergolong ke dalam senyawa isoparafin jauh lebih banyak daripada senyawa yang tergolong n-parafin. Tetapi, di dalam minyak bumi, kadar senyawa isoparafin biasanya lebih kecil daripada n-parafin. 2. Olefin Olefin adalah kelompok senyawa hidrokarbon tidak jenuh, CnH2n. Contohnya etilena (C2H4), propena (C3H6), dan butena (C4H8). 3. Naftena Naftena adalah senyawa hidrokarbon jenuh yang membentuk struktur cincin dengan rumus molekul CnH2n. Senyawa-senyawa kelompok naftena yang banyak ditemukan adalah senyawa yang struktur cincinnya tersusun dari lima atau enam atom karbon. Contohnya adalah siklopentana (C5H10), metilsiklopentana (C6H12) dan sikloheksana (C6H12). Umumnya, di dalam minyak bumi, naftena merupakan kelompok senyawa hidrokarbon yang memiliki kadar terbanyak kedua setelah n-parafin. 4. Aromatik Aromatik adalah hidrokarbon-hidrokarbon tak jenuh yang berintikan atomatom karbon yang membentuk cincin benzen (C6H6). Contohnya benzen (C6H6), metilbenzen (C7H8), dan naftalena (C10H8). Minyak bumi dari Sumatera dan Kalimantan umumnya memiliki kadar aromatik yang relatif besar. Selain senyawa-senyawa yang tersusun dari atom-atom karbon dan hidrogen, di dalam minyak bumi ditemukan juga senyawa non hidrokarbon seperti belerang, nitrogen, oksigen, vanadium, nikel dan natrium yang terikat pada rantai atau cincin hidrokarbon. Unsur-unsur tersebut umumnya tidak dikehendaki berada di dalam produk-produk pengilangan minyak bumi, sehingga keberadaannya akan sangat mempengaruhi langkah-langkah pengolahan yang dilakukan terhadap suatu minyak bumi. Pencemaran Minyak Petroleum hidrokarbon merupakan polutan utama pada lingkungan laut yang merupakan hasil dari kegiatan di daratan dan buangan dari sungai-sungai di sekitarnya, seperti kegiatan di kilang minyak di pantai, produksi minyak di lepas pantai, kegiatan pelayaran, dan kecelakaan tanker serta tumpahnya minyak dan produk bahan bakar petroleum lainnya (Yakimov 1998). Pencemaran minyak di perairan paling sering terjadi dibandingkan di darat. Pencemaran minyak di laut bukan hanya akibat dari kecelakaan kapal, tetapi juga bersumber dari kegiatan pengeboran, produksi minyak dan turunannya, pengilangan, transportasi minyak, perembesan minyak bumi dari reservoirnya, serta kegiatan pemuatan dan pembongkaran muatan kapal tanker di pelabuhan. Meningkatnya frekuensi pencemaran akan mengancam kebersihan lingkungan perairan. Bila hal ini tidak segera ditanggulangi, dalam waktu singkat laju pencemaran laut akan menjadi tidak terkendali (Fahruddin 2004). Minyak bumi merupakan salah satu jenis polutan yang masuk ke dalam ekosistem perairan pantai dan laut. Sebagian dari polutan tersebut larut dalam air, sebagian tenggelam ke dasar dan terkonsentrasi di sedimen dan sebagian masuk ke dalam jaringan tubuh organisme laut, termasuk fitoplankton, ikan, udang, cumi-cumi, kerang, rumput laut dan lain-lain. Polutan di dalam tubuh organisme tingkat rendah termakan oleh jenjang organisme di atasnya sehingga terikut dalam rantai makanan mulai dari fitoplankton sampai ikan predator dan pada akhirnya terakumulasi di dalam tubuh manusia. Bila dalam jaringan tubuh organisme laut terdapat polutan dengan konsentrasi tinggi, kemudian organisme tersebut dijadikan bahan makanan, maka akan berbahaya bagi kesehatan manusia (Nurhariyati 2006). Sesaat setelah terlepas ke lingkungan laut, minyak akan mengalami perubahan sifat-sifat fisik, kimia dan biologis. Perubahan sifat ini terjadi sebagai akibat dari faktor-faktor fisika di laut, diantaranya : 1) Evaporasi. Evaporasi alkana (C15) dan aromatik berlangsung antara 1 – 10 hari (Yakimov 1998, Zhu et al. 2001). Faktor lingkungan yang mempengaruh evaporasi adalah angin, gelombang air dan temperatur. 2) Pelarutan. Komponen minyak aromatik dengan berat molekul kecil dan paling toksik bersifat paling larut air dibanding senyawa minyak lainnya, maka proses ini juga penting dalam degradasi. Kecepatan pelarutan dipengaruhi oleh proses foto-oksidasi dan proses biologis (NAS, 1985). 3) Foto-oksidasi. Dalam kondisi aerobik dan terpapar sinar matahari, minyak aromatik dapat ditransformasi menjadi senyawa lebih sederhana. Senyawa lebih sederhana ini (hydroperoxides, aldehydes, ketones, phenols, dan carboxylic acids) bersifat lebih larut air sehingga meningkatkan laju biodegradasi tetapi lebih toksik (Nicodem et al. 1997, Yakimov 1998). 4) Dispersi. Proses ini terjadi karena gradien konsentrasi yang membentuk formasi emulsi minyak-air (butiran minyak dalam kolom air) sehingga memperluas permukaan butir minyak. Emulsi minyak-air dapat terjadi karena adanya agitasi (angin dan gelombang adalah contoh agitasi alamiah), atau dengan penambahan dispersan (Fahruddin 2004). 5) Emulsifikasi. Emulsifikasi adalah proses perubahan bentuk dari butiran minyak dalam air menjadi butiran air dalam minyak (disebut juga chocolate mousse). Bahan asphaltik dapat meningkatkan emulsifikasi, tetapi akan mempersulit pembersihan minyak (Kusuma 2009). 6) Biodegradasi oleh mikroflora laut terutama bakteri (Yakimov 1998). 7) Lain-lain. Termasuk adsorpsi minyak pada zat padat air, sedimentasi dan formasi butir tar (Fahruddin 2004) Efek Pencemaran Minyak Menurut Syakti (2008), pencemaran minyak bumi di laut memberikan pengaruh terhadap beberapa hal yang meliputi (1) pengaruh langsung terhadap organisme; (2) pengaruh langsung terhadap kegiatan perikanan; (3) pengaruh terhadap ekosistem. Pengaruh langsung terhadap organisme meliputi efek letal (kematian) dan subletal. Pengaruh langsung terhadap kegiatan perikanan dapat berupa tainting (bau lantung) terhadap ikan-ikan yang dibudidayakan di dalam karamba, sehingga ikan mempunyai bau dan cita rasa yang tidak enak. Pada kegiatan budidaya, pencemaran minyak bumi dapat mematikan biota budidaya dan merusak peralatan. Pengaruh terhadap ekosistem dapat berupa rusaknya daerah perkembangbiakan dan daerah penyedia makanan, serta terganggunya ketersediaan makanan dalam rantai makanan. Senyawa hidrokarbon yang terkandung dalam minyak bumi berupa benzena, toluena, ethylbenzena, dan isomer xylena, dikenal sebagai BTEX, merupakan komponen utama dalam minyak bumi, bersifat mutagenik dan karsinogenik pada manusia. Senyawa ini bersifat rekalsitran, yang artinya sulit mengalami perombakan di alam, baik di air maupun di darat, sehingga hal ini dapat mengalami proses biomagnition pada ikan ataupun pada biota laut yang lain. Bila senyawa aromatik tersebut masuk ke dalam darah, akan diserap oleh jaringan lemak dan mengalami oksidasi dalam hati membentuk fenol, kemudian pada proses berikutnya terjadi reaksi konjugasi membentuk senyawa glucuride yang larut dalam air, kemudian masuk ke ginjal. Senyawa antara yang terbentuk adalah epoksida benzena yang beracun dan dapat menyebabkan gangguan serta kerusakan pada tulang sumsum. Keracunan yang kronis menimbulkan kelainan pada darah, termasuk menurunnya sel darah putih, zat beku darah, dan sel darah merah yang menyebabkan anemia. Kejadian ini akan merangsang timbulnya preleukemia, yang pada akhirnya menyebabkan leukemia. Dampak lain adalah menyebabkan iritasi pada kulit (Fahruddin 2004). Selain itu Fahruddin (2004) juga menyatakan bahwa komponen minyak tidak larut di dalam air akan mengapung di permukaan air laut sehingga menyebabkan air laut berwarna hitam. Hal ini mengakibatkan penetrasi cahaya menurun di bawah oil slick atau lapisan minyak. Proses fotosintesis akan terhalang pada zona eufotik sehingga rantai makanan yang berawal pada fitoplankton akan terputus. Lapisan minyak juga menghalangi pertukaran gas dari atmosfer dan mengurangi kelarutan oksigen yang akhirnya sampai pada tingkat tidak cukup untuk mendukung bentuk kehidupan laut yang aerob. Beberapa komponen minyak tenggelam dan terakumulasi di dalam sedimen sebagai deposit hitam pada pasir dan batuan-batuan di pantai. Kondisi ini menyebabkan kematian hewan dan tumbuh–tumbuhan yang hidup di batu-batuan dan pasir di wilayah pantai, juga merusak area mangrove serta daerah air payau secara luas. Hutan mangrove merupakan sumber nutrien dan tempat pemijah bagi ikan. Pencemaran minyak dapat menyebabkan sistem perakaran dari tanaman hutan mangrove dapat tertutup minyak sehingga pertukaran CO2 dan O2 terhambat. Komponen hidrokarbon yang bersifat toksik berpengaruh terhadap reproduksi, perkembangan, pertumbuhan, dan perilaku biota laut, terutama pada plankton dan dapat mematikan ikan. Proses emulsifikasi merupakan sumber mortalitas bagi organisme, terutama pada telur, larva, dan perkembangan embrio karena pada tahap ini biota sangat rentan pada lingkungan tercemar. Definisi Bioremediasi Meskipun evaporasi dan fotooksidasi berperan utama dalam detoksifikasi minyak namun pada akhirnya degradasi sempurna komponen-komponen minyak dilakukan oleh mikroflora laut dan bakteri mendominasi fungsi ini. Sayangnya degradasi oleh mikroba secara alami berjalan relatif lambat dalam lingkungan laut, karena suhu yang rendah, keterbatasan nitrogen dan fosfor serta besarnya jumlah residu minyak yang merubah bentuk minyak dari emulsi menjadi tarballs yang akan mengendap dalam sedimen (Yakimov 1998). Metode bioremediasi merupakan cara penanggulangan tumpahan minyak yang paling aman bagi lingkungan (Prince et al. 2003). Bioremediasi didefinisikan sebagai proses penguraian limbah organik maupun anorganik polutan secara biologis dalam kondisi terkendali dengan tujuan mengontrol atau bahkan mereduksi bahan pencemar dari lingkungan. Kelebihan teknologi ini ditinjau dari aspek komersil adalah relatif lebih ramah lingkungan, biaya penanganan yang relatif lebih murah dan bersifat fleksibel. Penanganan bioremediasi dapat dilakukan secara in situ maupun ex situ (Syakti 2008). Fahruddin (2004) mengartikan bioremediasi hidrokarbon sebagai suatu proses penguraian senyawa-senyawa hidrokarbon kompleks menjadi air, karbondioksida dan senyawa organik sederhana secara biologis. Dalam proses tersebut terjadi oksidasi senyawa organik kompleks menjadi senyawa anorganik. Substrat hidrokarbon dari minyak bumi digunakan oleh mikroorganisme sebagai sumber karbon dan nutrien untuk pertumbuhan dan perolehan energi. Senyawa organik diubah menjadi CO2, komponen sel dan produk lain sesuai jalur metabolisme yang ditempuh. Menurut Glick dan Pasternak (2003) bioremediasi adalah proses penggunaan agen biologi untuk menghilangkan limbah atau buangan yang bersifat toksik dari lingkungan. Proses bioremediasi dapat dilakukan secara bioaugmentasi yaitu penambahan atau introduksi satu jenis atau lebih mikroorganisme baik yang alami maupun yang sudah mengalami perbaikan sifat (improved/genetically engineered strains), dan biostimulasi yaitu suatu proses yang dilakukan melalui penambahan zat gizi tertentu yang dibutuhkan oleh mikroorganisme atau menstimulasi kondisi lingkungan sedemikian rupa (misalnya pemberian aerasi) agar mikroorganisme tumbuh dan beraktivitas lebih baik (Irianto 2007). Dua pendekatan yang dapat digunakan dalam bioremediasi tumpahan minyak: (1) bioaugmentasi, di mana mikroorganisme pengurai ditambahkan untuk melengkapi populasi mikroba yang telah ada, dan (2) biostimulasi, di mana pertumbuhan pengurai hidrokarbon asli lingkungan tersebut dirangsang dengan cara menambahkan nutrien dan/atau mengubah habitat (Venosa dan Zhu 2003). Bioaugmentasi Bioaugmentasi didefinisikan sebagai penambahan kultur mikroba untuk melakukan tugas resubstratsi spesifik di dalam lingkungan tercemar. Mikroba dalam kultur tersebut diisolasi secara khusus, pada umumnya dari lingkungan yang sama, ditapis untuk aktivitas biologi yang diinginkan, dan ditumbuhkan dalam jumlah yang besar dalam suatu reaktor (United-tech 2009). Ractliffe (2002) menyatakan bioremediasi hidrokarbon baik dilakukan menggunakan suatu proses yang disebut bioaugmentasi. Proses ini dilakukan dengan menambahkan sejumlah besar mikroorganisme yang telah diisolasi, diseleksi dan ditumbuhkan di laboratorium pada lingkungan yang terkontaminasi. Mikroba tersebut mampu mendegradasi komponen-komponen dalam hidrokarbon menjadi CO2 dan air. Mikroba tersebut akan bertahan hidup dengan mengkonsumsi hidrokarbon sampai polutan tersebut teresubstratsi. Agar proses bioaugmentasi berhasil dengan baik, maka dibutuhkan beberapa kriteria diantaranya : kemampuan mikroba untuk mencapai kontaminan, keberadaan oksigen untuk metabolisme mikroba, suhu antara 5 – 45 oC (28 oC merupakan suhu optimum), pH antara 6,5 – 8,5 dan penambahan nutrien. Selama mikroba dapat mencapai kontaminan, tersedia oksigen serta suhu dan pH yang sesuai, maka proses remediasi akan berlangsung dengan sempurna. Bakteri yang digunakan dalam bioaugmentasi Bakteri dianggap sebagai salah satu mikroorganisme yang bertanggung jawab terhadap degradasi hidrokarbon di lingkungan (Leahy et al. 1990) dan bakteri hidrokarbonoklastik bersifat kosmopolitan, dapat ditemukan di berbagai jenis lingkungan. Lebih dari 20 genera bakteri pendegradasi hidrokarbon terdistribusi dalam beberapa subphylum (α−, β−, γ−proteobacteria; gram positif; Flexibacter-Cytophaga-Bacteroides) telah dilaporkan (Bruns and Corti 1999; Macnaughton et al. 1999; Yakimov et al. 1998). Sejumlah bakteri pendegradasi hidrokarbon telah diisolasi dari lingkungan laut dan telah dikarakterisasi (Kasai et al. 2002, Ozaki et al. 2006, Teramoto et al. 2010), meskipun informasi mengenai bakteri tersebut pada lingkungan tropis masih langka (Zhuang et al. 2003). Harwati et al. (2007, 2009) telah mengisolasi sejumlah bakteri laut dari air laut Pelabuhan Tanjung Mas Semarang, dan beberapa diantaranya mempunyai kemampuan mendegradasi hidrokarbon. Zhu et al. (2001) melaporkan beberapa bakteri yang mempunyai kemampuan mendegradasi hidrokarbon di daerah subtropis, diantaranya Achromobacter, Acinetobacter, Alcaligenes, Arthrobacter, Bacillus, Brevibacterium, Corynebacterium, Flavobacterium, Nocardia, Pseudomonas dan Vibrio, sedangkan Darmayati (2003) dalam penelitiannya telah menemukan beberapa bakteri pendegradasi minyak dari perairan tropis, diantaranya Pseudomonas cepacia dan P. gladioli yang diisolasi dari perairan Kalimantan Timur, demikian juga Achromobacter putrefasciens, Acinetobacter haemolyticus, dan Vibrio algynolyticus yang berhasil diisolasi dari perairan laut Jawa. Feliatra (1998) mengisolasi Acinetobacter, Arthrobacter, Micrococcus dan Bacillus dari perairan Dumai dan Selat Malaka. Genus Alcanivorax, Marinobacter, Bacillus dan Achromobacter merupakan genera yang umum ditemukan di lokasi penelitian. Menurut Teramoto et al. (2009) Marinobacter dan Alcanivorax terdapat di mana-mana di perairan laut tropis, namun Oceanobacter-related menjadi dominan di perairan tropis yang diperkaya dengan pupuk. Diperkirakan bakteri ini merupakan bakteri pemakan n-alkana di perairan tropis. Darmayati (2008) serta Hatmanti dan Darmayati (2009) menyatakan bahwa Alcanivorax merupakan genus yang umum ditemukan di semua lokasi penelitian di Teluk Jakarta, sehingga dianggap mempunyai penyebaran yang kosmopolitan. Genus ini meliputi Alcanivorax dieselolei, Alcanivorax sp TE-9, Alcanivorax sp. EPR 6 dan Alcanivorax sp B 1084. Alcanivorax sebagai genus yang kosmopolitan telah dilaporkan di beberapa tempat diantaranya di perairan Indonesia (Thontowi 2008; Darmayati 2009), Jerman (Bruns dan Berthe-Corti 1999), Inggris (Rolling et al. 2002), Italia (Yakimov et al. 2005), dan Jepang (Kasai et al. 2002). Lebih dari 250 spesies dari genus afiliasi Alcanivorax telah diisolasi dan dideteksi menggunakan sekuens 16S rRNA. Bakteri ini dapat berada di beberapa tipe lingkungan laut, baik dalam komunitas bakteri maupun kultur tunggal bakteri yang diisolasi dari lingkungan subtropis (Darmayati 2008). Hubungan pencemaran minyak dan komunitas bakteri Bakteri sebagai kultur tunggal yang spesifik, hanya mampu mengurai sejumlah kecil komponen yang terdapat dalam minyak, namun biodegradasi minyak pada umumnya dilakukan oleh konsorsium yang terdiri atas bermacammacam spesies bakteri (Roling et al. 2002). Penguraian minyak biasanya dilakukan oleh suatu komunitas bakteri. Setiap jenis bakteri memiliki kemampuan yang berbeda-beda dalam mengurai minyak. Ada yang mampu menguraikan senyawa aromatik, senyawa hidrokarbon bercabang, maupun senyawa hidrokarbon sederhana, namun ada pula yang mampu mendegradasi beberapa jenis senyawa dalam hidrokarbon, seperti Alcanivorax borkumensis yang dikenal mampu mengurai ikatan jenuh dan aromatik. Bakteri tertentu yang dinyatakan dominan dan relatif memiliki kemampuan tinggi dalam mendegradasi minyak adalah Marinobacter, Oceanobacter, Alcanivorax, Thalassospira, Stappia, Bacillus, Novospingobium, Pseudomonas, Spingobium, dan Rhodobacter. Pseudomonas sp biasanya banyak dikenal sebagai bakteri pendegradasi hidrokarbon di Indonesia (Hariyadi 2009). DGGE : definisi dan hubungannya dengan komunitas bakteri Selama dekade terakhir ini banyak perubahan metode yang digunakan dalam menganalisis komposisi komunitas mikroba. Teknik klasik yang digunakan seperti kultivasi dan identifikasi secara mikroskopik tidak cukup untuk menganalisis kelimpahan jenis bakteri dalam sampel dari alam (lingkungan). Di satu sisi, kurangnya perbedaan yang mencolok pada identifikasi secara morfologi dan ukuran sel yang sangat kecil tidak memungkinkan penggunaan identifikasi mikroskopis pada sebagian besar bakteri yang diisolasi dari sampel alam, di sisi lain substrat yang digunakan untuk mengkultivasi galur mikroba adalah substrat selektif sehingga menjadi bias ketika mendeskripsikan komposisi komunitas. Selain itu, isolasi sebagian besar bakteri dari alam terhambat oleh kurangnya pengetahuan kita tentang kondisi kultur yang spesifik dan terdapatnya interaksi dalam komunitas (quorum sensing). Perbandingan antara sel yang dapat dikulturkan dan jumlah sel total dalam habitat yang berbeda menunjukkan kekurangan pendekatan culture-dependent untuk menganalisis komposisi komunitas mikroba (Amann et al. 1995). Studi mengenai komunitas bakteri memunculkan pertanyaan mengenai komposisi komunitas tersebut, struktur, stabilitas, aktivitas dan fungsinya baik dalam komunitas tersebut maupun secara individu. Teknik mikrobiologi tradisional dan observasi menggunakan mikroskop konvensional tidak cukup untuk menjawab pertanyaan tersebut. Sebagian besar bakteri dalam sampel dari lingkungan tidak dapat dideteksi menggunakan mikroskop konvensional, karena melekat pada tanah dan partikel sedimen dan tetap tidak terlihat. Pewarna fluorescence, seperti DAPI dan acridine orange, telah meningkatkan penggunaan teknik ini, namun tetap tidak diperoleh informasi mengenai identitas spesies. Selain itu hanya sebagian kecil dari bakteri alam yang dapat diisolasi dan dikarakterisasi sampai saat ini. Kultur substrat selektif pengayaan masih belum bisa digunakan untuk menggambarkan kondisi khusus yang dibutuhkan oleh bakteri untuk berkembangbiak di habitat alam mereka, sehingga pemahaman mengenai keragaman bakteri di alam sangat terbatas. Bagaimanapun aplikasi teknik biologi molekuler menawarkan peluang baru untuk menganalisis komunitas mikroba, diantaranya Teknik Denaturing Gradient Gel Electrophoresis (DGGE). Pada DGGE, fragmen DNA yang mempunyai panjang sama, namun dengan sekuens pasangan basa yang berbeda dapat dipisahkan. Pemisahan ini didasarkan kepada pengurangan mobilitas elektroporesis dari molekul DNA yang terdenaturasi secara parsial dalam gel poliakrilamid yang mengandung gradien DNA denaturant yang bertambah secara linear (Muyzer et al. 2004). Schafer dan Muyzer (2001) menyatakan bahwa komposisi komunitas bakteri yang berperan dalam proses bioremediasi minyak bumi dapat diketahui dengan menggunakan teknik molekuler yang disebut DGGE. PCR-DGGE fingerprinting adalah suatu teknik untuk memonitor variasi keragaman genetik mikroba, yang menyediakan estimasi minimum kekayaan atau dominansi suatu bakteri di dalam komunitas tersebut. Lebih lanjut DGGE memudahkan identifikasi populasi individu dengan analisis hibridisasi pola pita dengan probe spesifik atau dengan melakukan analisis sekuensing terhadap pita individual. PCR-DGGE telah digunakan untuk menginvestigasi keragaman komunitas mikroba, mendeterminasi variabilitas spasial dan temporal suatu populasi bakteri, dan memonitor perilaku komunitas setelah tercemarnya suatu lingkungan baik secara alami ataupun induksi secara buatan. Perkembangan teknologi molekuler dalam menganalisis ekologi mikroba, termasuk penggunaan sekuens gen rRNA sebagai marker molekular untuk mengidentifikasi mikroorganisme telah mengubah persepsi tentang keragaman komunitas mikroba. Gen yang menyandikan subunit kecil rRNA merefleksikan hubungan evolusi mikroorganisme (Woese 1987). Teknik penyidikan secara molekuler merupakan teknik yang unggul untuk membandingkan sejumlah besar sampel. Genetic fingerprinting komunitas mikroba memberikan profil yang merefleksikan kelimpahan genetik dari komunitas tersebut. DGGE dari fragmen gen yang diamplifikasi dengan PCR adalah salah satu dari teknik penyidikan secara genetik dalam ekologi mikroba (Muyzer 2000). Prinsip DGGE Sekumpulan DNA diekstrak dari konsorsium komunitas mikroba dalam suatu substrat, kemudian diamplifikasi dengan primer yang spesifik untuk fragmen gen 16S rRNA sehingga menghasilkan campuran produk PCR. Produk PCR ini mempunyai ukuran yang sama, sehingga tidak dapat dipisahkan dengan elektroforesis menggunakan gel agarosa. Variasi sekuen di antara gen 16S rRNA dari bakteri yang berbeda-beda memberikan sifat ”melting” yang berbeda, sehingga molekul-molekul DNA yang bervariasi sekuennya tersebut dapat dipisahkan dengan menggunakan elektroforesis gel poliakrilamid yang mengandung gradien denaturan DNA. Contoh gradien denaturan DNA yang dapat digunakan adalah campuran urea dan formamida. Produk PCR memasuki gel sebagai molekul DNA utas ganda (double strands) dan bergerak (bermigrasi) di dalam gel yang konsentrasi denaturannya meningkat secara bertahap. Produk PCR dengan sekuens yang berbeda mulai terdenaturasi pada posisi yang berbeda (pada konsentrasi denaturant yang berbeda) di dalam gel. Melting proceeds sering juga disebut sebagai melting domain, yaitu daerah di mana suatu DNA mengalami denaturasi. Bila suatu DNA mencapai melting domain pada gradien denaturan, akan terjadi transisi molekul DNA dari utas ganda menjadi utas tunggal secara parsial. Utas tunggal yang mencuat menyebabkan berhentinya pergerakan molekul DNA pada posisi tersebut. Untuk mencegah penguraian komplet dua utas DNA, maka suatu sekuens 40 nukleotida yang kaya akan GC (GC clamp) dilekatkan pada 5’-end pada satu primer PCR (Schafer and Muyzer 2001). Gambar 1 Prinsip Denaturing Gradient Gel Electrophoresis (DGGE) DNA yang diisolasi dari campuran spesies mikroba yang berbeda diamplifikasi menggunakan primer universal untuk suatu kelompok organisme yang disisipi dengan susunan GC berulang (sepanjang 40 bp, yang disebut GCclamp). GC-clamp ini berfungsi sebagai penjepit rantai ganda DNA sehingga tidak terpisah menjadi rantai tunggal pada saat dielektroforesis pada gel yang mengandung zat pendenaturasi. Ketahanan rantai ganda DNA terhadap zat pendenaturasi berbeda-beda tergantung dari komposisi (urutan) nukleotida yang ada. Perbedaan urutan nukleotida ini menyebabkan DNA terdenaturasi pada konsentrasi zat pendenaturasi tertentu. Perenggangan rantai ganda DNA menyebabkan pergerakan DNA berhenti dalam matrik gel pada saat dielektroforesis (Gambar 1). Dengan demikian, sekuen DNA yang berbeda, bahkan perbedaan hanya satu pasang basa nukleotida, akan muncul sebagai pita pada posisi yang berbeda di dalam gel akrilamid (Muyzer et al. 1993). BAHAN DAN METODE Waktu dan Tempat Penelitian Penelitian dan analisis dilakukan mulai bulan Januari 2010 – Juni 2011 di Laboratorium Mikrobiologi Laut Puslit Oseanografi LIPI dan P.T. Charoon Phokpand. Materi Penelitian Substrat yang digunakan adalah sampel air laut dan sedimen dari perairan Pulau Pari Teluk Jakarta yang dicampur untuk menggambarkan kondisi alam yang mendekati sebenarnya (untuk tujuan aplikasi). Sebelum dicampur dengan air laut, sampel sedimen yang diambil dari beberapa titik di perairan Pulau Pari Teluk Jakarta dikomposit terlebih dahulu, kemudian didistribusikan ke dalam tabung-tabung dan ditambah dengan air laut dengan perbandingan 1:1. Sampelsampel tersebut terdiri atas 264 tabung, yaitu 88 x 3 ulangan tabung (Kusuma 2009; Arifah 2010). Pada penelitian ini digunakan 84 tabung (28 x 3 ulangan) yang terdiri atas: 4x3 tabung disterilisasi dan ditambah bakteri RCO/B/08_008, 4x3 tabung disterilisasi dan ditambah konsorsium A (Strain RCO/B/08_006, RCO/B/08_008, dan RCO/B/08_013), 4x3 tabung tidak disterilisasi dan ditambah bakteri RCO/B/08_008, 4 tabung tidak disterilisasi dan ditambah konsorsium A, 8x3 tabung tidak disterilisasi dan tidak ditambah bakteri, serta 4x3 tabung disterilisasi tanpa ditambah bakteri. Substrat dalam tabung-tabung tersebut diinkubasikan selama 0, 7, 14 dan 28 hari. Setelah masa inkubasi masingmasing, substrat dalam tabung disimpan pada suhu -80oC. Matrik perlakuan sampel yang dianalisis dapat dilihat pada Tabel 2. Tabel 2 Matrik sampel yang dianalisis komunitas bakterinya RCO/B/08_ 008 Substrat Konsorsium A Tanpa inokulum-1 Tidak disterilisasi 0 7 14 28 0 7 14 28 0 7 14 28 Disterilisasi 0 7 14 28 0 7 14 28 0 7 14 28 Keterangan: Tanpa inokulum-2 0 7 14 28 Penambahan konsorsium A terdiri atas strain RCO/B/08_006, RCO/B/08_008, dan RCO/B/08_013; Perlakuan tanpa penambahan bakteri dilakukan dua ulangan; Pengambilan sampel dilakukan pada hari ke-0, 7, 14 dan 28; masing-masing tabung dilakukan 3 kali ulangan Strain bakteri yang digunakan di dalam penelitian ini yaitu RCO/B/08_006, RCO/B/08_008, RCO/B/08_009, RCO/B/08_013 adalah koleksi Laboratorium Mikrobiologi Laut Puslit Oseanografi LIPI. Masing-masing strain memiliki tingkat kesejajaran 99% atau lebih dengan Pseudomonas balearica, Alcanivorax sp. TE9, Bacillus sp. L41 dan Bordetella sp F2 (Tabel 3). Bahan yang digunakan untuk isolasi DNA adalah ISOIL Bead-beating DNA Extraction Kit dan Applied Biosystems Prepman Ultra Sample Preparation Reagent. Bahan yang digunakan untuk PCR dan elektroforesis meliputi HotstarTag PCR Mix (QIAGEN), Primer 341F (5’-CCTACGGGAGGCAGCAG-3’), Primer 907R (5’- CCGTCAATTCMTTTGAGTT T-3’), Primer 341F-GC (40pb GC clamp 5’CCTACGGGAGGCAGCAG-3’), MilliQ PCR grade, Etanol 70%, QIAquick PCR purification kit (QIAGEN), Urea, Formamida, Akrilamida/bis-akrilamida, Ammonium Persulphate (APS), TEMED, Agarosa, Akuades, TAE Buffer 1x, Dyesolution, Gel-loading solution, Etidium Bromida, Etanol 90%, air murni. Tabel 3 Strain bakteri koleksi Laboratorium Mikrobiologi Laut Puslit Oseanografi LIPI yang digunakan dalam penelitian No. 1. 2. 3. 4. Kode Isolat RCO/B/08_006* RCO/B/08_008* RCO/B/08_009* RCO/B/08_013* Kesejajaran dengan Gen Bank NCBI AM905859.1 Pseudomonas balearica st101 (99%) AB055207.1 Alcanivorax sp. TE-9 (100%) DQ249996.1 Bacillus sp. L41 (99%) DQ453689.1 Bordetella sp. F2 (99%) Keterangan : * adalah isolat koleksi Laboratorium Mikrobiologi Laut Puslit Oseanografi LIPI (Hatmanti dan Darmayati 2009) yang digunakan sebagai perlakuan dalam penelitian Kusuma (2009) dan Arifah (2010) Metode Penelitian Sampel Ekstraksi DNA (ISOIL beads beating) PCR dengan primer GC clamp Sekuensing Analisis Sekuensing DGGE Analisis DGGE Isolasi DNA dari poliakrilamid gel Analisis Sampel Komunitas mikroba dilihat secara kualitatif dengan menggunakan metode Denaturing Gradient Gel Electrophoresis (DGGE). Metode ini diawali dengan isolasi asam nukleat sampel (Rosello-Mora et al. 1999), yang kemudian digunakan sebagai sampel untuk analisis DGGE (Muyzer et al. 1993, 1996). Urutan kerja analisis sampel adalah sebagai berikut : Isolasi DNA Isolasi DNA dilakukan dengan menggunakan metode bead beating, yaitu penggunaan beads (manik-manik) untuk melisiskan sel bakteri yang menempel pada permukaan minyak. Sebanyak 1 ml sampel disentrifuse pada kecepatan lambat dan suhu dingin untuk mengumpulkan biomassa selnya. Supernatan dibuang dan pada pellet ditambahkan campuran 50 mg Skim Milk, 10 μl Triton X dan 950 μl Lysis Solution BB dan dicampur pelan dengan menggunakan vortex. Campuran diinkubasi pada suhu 65 oC selama 5 menit, kemudian dikocok pada kecepatan 3.000 rpm selama 5 menit (300 detik) dengan Beads Cells Disrupter. Campuran kemudian diinkubasi kembali pada suhu 65 oC selama 30 menit, dan dibolak-balik 10 kali setiap 10 menit. Tahap ini dilakukan sebanyak dua kali. Tabung disentrifuse pada 12.000 x g selama 1 menit pada suhu ruangan. Sebanyak 600 μl supernatan ditransfer ke tabung baru, kemudian ditambahkan 400 μl “Purification Solution” dan dihomogenkan dengan pengocokan. Pada campuran ditambahkan 600 μl “Chloroform”, dan divortex selama 15 detik, kemudian disentrifuse pada 12.000 x g selama 15 menit pada suhu kamar. Sebanyak 800 μl supernatant bagian atas (tidak di bagian tengah) diambil dan dipindahkan ke tabung baru serta ditambahkan 800 μl “Precipitation Solution”. Larutan disentrifuse pada 20.000 x g selama 20 menit pada 4 oC, supernatan dibuang, dan pada pelet ditambahkan 1 ml “Wash Solution”. Larutan disentrifuse lagi pada 20.000 x g selama 10 menit pada 4o C, supernatan dibuang pelanpelan. Sebanyak 1 ml 70% etanol dan 2 μl Ethacinmate ditambahkan pada pelet dan disentrifuse 20.000 x g selama 5 menit pada suhu 4 o C kemudian supernatan dibuang dan pelet dikeringkan dengan meletakkannya terbalik pada tissue di dalam laminair airflow. Pelet dilarutkan dalam 30 μl TE, kemudian disimpan pada suhu -20 oC atau segera dianalisis lebih lanjut. PCR untuk DGGE PCR untuk DGGE dilakukan menggunakan primer dengan GC clamp (341F-GC) dan 907R. Kondisi PCR yang dilakukan sebagai berikut aktivasi dilakukan selama 5 menit pada 95oC, diikuti dengan 30 siklus yang terdiri dari : melting selama 30 detik pada 94oC, annealing selama 1 menit pada 55oC dan ekstensi selama 1 menit pada 72 oC, diikuti dengan 10 menit final ekstensi pada 72 oC, dan pendinginan selama 5 menit pada suhu 4oC (modifikasi dari Muyzer et al. 1993, 1996). Produk PCR kemudian dicek dalam 1,5% gel agarose. DGGE DGGE dilakukan dengan menggunakan alat Dcode™ Universal Mutation Detection System BioRad dengan gel berukuran 16 x 16 cm dan ketebalan 1,0 mm. DGGE dilakukan dalam 7 liter 1 x larutan TAE buffer (20 mM Tris acetat, 0,5 mM EDTA, pH 8,0) pada suhu 60 oC selama 210 menit pada voltase 200 Volt. Gradien yang digunakan berkisar antara 35% - 65% denaturant (100% denaturant yang terdiri dari 7 M urea ditambah 40 % vol/vol formamide). Gel diwarnai menggunakan Etidium Bromida (0,5 mg/L) selama 30-45 menit dan dibilas dalam 1 x TAE buffer selama 15 menit pada suhu kamar. Gel kemudian difoto menggunakan Gel-Doc UV Transluminator. Ekstraksi DNA dari Gel Polyacrilamide dan Analisis Sekuen Pada pita yang ditargetkan, pemotongan dilakukan menggunakan pisau/cutter yang telah disterilisasi menggunakan alkohol 70%. Pita yang telah dipotong direndam dalam 30 μl akuades untuk PCR selama semalam pada suhu 4 oC. Sebanyak 2 μl cairan elusi DNA diambil dan digunakan sebagai cetakan dalam PCR untuk sekuensing. Produk PCR kemudian dielektroforesis pada 1,5% gel agarose dan dipurifikasi lebih lanjut menggunakan Gene-Clean Kit (Bio 101). DNA kemudian disekuensing menggunakan Sekuenser ABI Prisma 373. Sekuen yang diperoleh kemudian dianalisis lebih lanjut menggunakan fasilitas BLAST dari NCBI (www.ncbi.nlm.nih.gov). Analisis Sekuen Nukleotida Database 16S rDNA yang diperoleh dari penelitian ini dideterminasi menggunakan BLAST program dan database GenBank. Teknik profil alignment (penyejajaran) ClustalX versi 1.7 digunakan untuk mensejajarkan sekuen. HASIL DAN PEMBAHASAN Lingkungan, baik mikroorganisme yang air, tanah beragam. maupun Menurut udara diyakini Nakatsu mengandung (2007), keragaman mikrobiologi yang tertinggi di planet bumi terdapat pada tanah. Pemahaman mengenai keragaman mikrobiologi mencakup pengetahuan tentang ekologi mikroba dan struktur komunitasnya. Jumlah spesies di dalam sesuatu komunitas (spesies richness) dan ukuran populasi spesies di dalam suatu komunitas (spesies evenness) merupakan parameter penting dalam menentukan struktur dan keanekaragaman dalam suatu komunitas (Liu et al. 1997). Studi mengenai keragaman bakteri dan dinamika komunitas bakteri berkembang pesat dalam ekologi mikroba, dipercepat dengan kemajuan dalam teknik-teknik molekuler. Analisis komunitas bakteri yang berperan dalam aktivitas biodegradasi hidrokarbon in situ menggunakan pendekatan molekuler merupakan suatu tantangan, karena hampir sebagian besar (>90 – 99%) spesies bakteri yang berkompeten dalam komunitas pendegradasi hidrokarbon tidak dapat ditumbuhkan dalam substrat artifisial (Wilkinson 1988). Metode molekuler yang dapat digunakan dalam analisis komunitas bakteri diantaranya adalah DGGE. Metode ini menggunakan molekul 16S rDNA yang dibawa oleh semua bakteri, dengan sekuen yang merupakan marker molekuler untuk identifikasi spesies (Macnaughton et al. 1999). Metode ini pada awalnya digunakan untuk menampilkan profil populasi mikroba dari sampel alam oleh Muyzer et al. (1993). Struktur komunitas bakteri indigenous pada lingkungan tercemar minyak di perairan Pulau Pari Teluk Jakarta Berdasarkan pita-pita pada profil DGGE (Gambar 2), diketahui bahwa bakteri indigenous secara alami telah terdapat pada substrat tercemar minyak dari Pulau Pari Teluk Jakarta. Hal ini dapat dilihat pada substrat yang tidak steril1 dan 2. Pada substrat tidak steril-1 pada hari ke-0 terdapat uncultured bacterium clone VH-FL6-50 (100%) dan uncultured bacterium clone W1-16 (92%), dan kedua bakteri ini bertahan dalam substrat sampai hari ke-28. Uncultured bacterium clone VH-FL6-50 adalah bakteri laut yang ditemukan menempel pada partikel dan dapat hidup bebas pada perairan di pelabuhan Victoria Hongkong China (Zhang et al. 2007). Bakteri ini ditemukan pula di lingkungan perairan Pulau Pari, dalam kondisi yang sama yaitu perairan tercemar minyak. Uncultured bacterium clone W1-16 ditemukan di sedimen dan air perairan Northern Yellow Sea, Korea (Zhao 2008), namun karena yang ditemukan di perairan Pulau Pari hanya mempunyai keidentikan 92% maka diduga merupakan spesies yang berbeda. Hagstrom et al. (2002) mengusulkan untuk mengelompokkan bakteri yang mempunyai keidentikan sekuen 16S rDNA minimal 97% ke dalam spesies yang sama, sehingga bila nilai keidentikannya di bawah 97% dapat diduga merupakan spesies yang baru. Namun dugaan ini harus diuji kebenarannya menggunakan metode identifikasi morfologi dan biokimia. Dalam penelitian ini tidak dilakukan identifikasi berdasarkan uji-uji tersebut. a b c e Pb Br Al Bc Bakteri Kontrol 0 7 14 28 Substrat tidak steril-1 tanpa inokulum f g d 0 h 7 14 f g 28 Substrat tidak steril-2 tanpa inokulum 0 7 14 28 hari Substrat steril tanpa inokulum Gambar 2 Profil DGGE pada proses bioremediasi secara alamiah dalam substrat tidak steril dan substrat steril tanpa penambahan bakteri (tunggal atau konsorsium), bakteri kontrol: Pb: RCO/B/08_006 (Pseudomonas balearica st101 99%)), Br: RCO/B/08_013 (Bordetella sp. F2 99%), Al: RCO/B/08_008 (Alcanivorax sp. TE-9 100%) Bc: RCO/B/08_009 (Bacillus sp. L41 99%); Pita yang terdeteksi: a: Marine bacterium SCRIPP 413 (98%), b: Uncultured bacterium clone VH-FL6-50 (100%), c: Pseudomonas sp. clone HY2 (98%), d: Uncultured bacterium clone W1-16 (92%), e: Uncultured bacterium Uday 0-58 (100%), f: Iron-reducing bacterium clone HN17 (98%), g: Uncultured bacteria clone N704B_48 (99%) dan h: Alteromonas sp. MOLA 3 (98%) Pada substrat tidak steril-1 hari ketujuh diperoleh pita dari marine bacterium SCRIPP 413 (98%). Marine bacterium SCRIPP 413 merupakan bakteri yang ditemukan oleh Hold et al. (2001) menempel pada dinoflagelata toksin dan nontoksin. Beberapa spesies marine bacterium dilaporkan dapat menghasilkan hydroxy-acid siderophore yang dapat mereduksi besi dalam degradasi minyak (Barbaeu et al. 2002). Diduga marine bacterium SCRIPP 413 (98%), uncultured bacterium clone VH-FL6-50 (100%) dan uncultured bacterium clone W1-16 (92%) merupakan bakteri indigenous yang dominan pada lingkungan perairan Pulau Pari yang tercemar minyak. Dominansi ini tidak dapat dihubungkan dengan peranan bakteri dalam degradasi minyak pada substrat yang tercemar minyak. Hal ini karena primer yang digunakan adalah sekuen fragmen 16S rDNA yang merupakan primer universal untuk kelompok bakteri. Bakteri-bakteri tersebut terdapat melimpah di dalam substrat mungkin juga karena kehadiran sumber karbon atau pencemar lain untuk metabolisme selnya. Untuk mengetahui kemampuannya dalam mendegradasi minyak perlu dilakukan analisis lanjutan DGGE, misalnya dengan melakukan hibridisasi gel DGGE menggunakan probe spesifik gen fungsional seperti gen alkB, yaitu gen penyandi enzim alkana monooksigenase. Alkana monooksigenase merupakan enzim yang berperan dalam jalur degradasi senyawa alkana (Whyte et al. 2002). Penggunaan probe tersebut dapat mengkonfirmasi keberadaan bakteri pendegradasi alkana di dalam substrat. Meskipun kehadiran bakteri indigenous pendegradasi hidrokarbon tidak dapat dipastikan di dalam substrat tidak steril-1, namun menurut Kusuma (2009) terjadi penurunan konsentrasi cemaran minyak dari 9,5 mg/20 ml menjadi 5,3 mg/20 ml atau sebesar 4,2 mg/20 ml (44% b/b) selama 28 hari. Penurunan ini menurut Nugroho (2006) terjadi karena di dalam substrat tercemar minyak mentah telah terdapat bakteri tertentu. Penurunan konsentrasi minyak ini berlangsung cepat sampai hari ke-14 di mana terdapat pertambahan jumlah total biomassa sel bakteri. Peningkatan jumlah bakteri ini dimungkinkan karena bertambahnya jumlah sel bakteri yang dapat memetabolisme komponenkomponen minyak, sehingga menyebabkan peningkatan jumlah sel. Peningkatan jumlah sel ini menyebabkan pita bakteri yang awalnya tidak terlihat menjadi terdeteksi di dalam gel poliakrilamida. Hal ini dibuktikan dengan diperolehnya empat pita yang berbeda pada profil DGGE pada substrat tidak steril-1 (Gambar 2). Gambar 3 menunjukkan bahwa proses penurunan konsentrasi minyak melambat mulai hari ke-14 sampai ke-28, seiring dengan menurunnya jumlah total biomassa bakteri. Hal ini terjadi karena ketersediaan sumber karbon yang dapat digunakan oleh bakteri semakin rendah. Kondisi ini menunjukkan bahwa konsentrasi cemaran minyak pada substrat yang tercemar minyak dapat menurun secara alami tanpa perlakuan apapun. Penguraian minyak secara alami dilakukan oleh bakteri indigenous yang terdapat di dalam substrat. Gambar 3 Hubungan antara total biomassa sel dengan penurunan konsentrasi minyak dalam substrat tidak steril tanpa penambahan strain RCO/B/08_008 (Kusuma 2009). : total sel; : berat minyak Penurunan konsentrasi cemaran minyak pada substrat tidak steril-2 yaitu sebesar 56% (berat/berat) selama 28 hari. Menurut Arifah (2010) hal ini menunjukkan bahwa secara alami bakteri pendegradasi hidrokarbon sudah ada di alam dan memiliki kemampuan yang cukup besar untuk menurunkan konsentrasi minyak mentah dari konsentrasi minyak awalnya. Bakteri hidrokarbonoklastik yang terdapat pada substrat tidak steril muncul ketika ada cemaran minyak yang terpenetrasi dalam sedimen dan air laut, sehingga walaupun tanpa dilakukan penambahan bakteri hidrokarbonoklastik eksogenous pun dalam substrat tetap terjadi aktivitas degradasi hidrokarbon. Hal ini dapat dilihat pada profil DGGE lajur substrat tidak steril-2 tanpa penambahan inokulum (Gambar 2). Perbedaan profil DGGE maupun persentase penurunan konsentrasi minyak pada substrat tidak steril-1 dan 2 terjadi karena kedua substrat tersebut disiapkan dalam waktu yang berbeda, walaupun menggunakan teknik pencampuran yang sama dan konsisten. Muyzer dan Smalla (1998) menyatakan bahwa DGGE dapat digunakan untuk memonitor perbedaan teknik penyimpanan dan penyiapan materi yang dilakukan. Seperti halnya pada substrat tidak steril-1, bakteri indigenous yang terdeteksi pada profil DGGE pada substrat tidak steril-2 pun tidak semuanya dapat diasumsikan sebagai bakteri yang ikut serta dalam proses degradasi minyak. Untuk mengetahui peranan bakteri-bakteri tersebut, maka perlu dilakukan analisis DGGE lebih lanjut dengan menggunakan probe-probe spesifik gen penyandi enzim pada bakteri pendegradasi hidrokarbon, misalnya monooksigenase dan dioksigenase (Luz et al. 2004). Pada profil DGGE substrat tidak steril-2 hari ke-0 diperoleh pita marine bacterium SCRIPP 413 (98%), uncultured bacterium clone VH-FL6-50 (100%) dan Pseudomonas sp. clone HY2 (100%). Marine bacterium SCRIPP 413 (98%) dan uncultured bacterium clone VH-FL6-50 (100%) merupakan bakteri indigenous yang sama seperti terdeteksi pada medium tidak steril-1. Pseudomonas sp. clone HY2 (100%) diperkirakan juga merupakan bakteri indigenous di perairan Pulau Pari Teluk Jakarta. Pseudomonas sp. clone HY2 merupakan bakteri yang mampu mengoksidasi hidrokarbon di Victoria Land coast Antartika (Lo dan Michaud 2010). Pseudomonas sp. banyak dilaporkan merupakan bakteri yang mampu memetabolisme berbagai jenis hidrokarbon, yaitu alifatik dan aromatik (Whyte et al. 1997; Darmayati 2009; Hatmanti dan Darmayati 2009). Struktur komunitas bakteri pada hari ketujuh dalam substrat tidak steril-2 berubah, marine bacterium SCRIPP 413 (98%) dan uncultured bacterium clone VH-FL6-50 (100%) masih tetap bertahan, namun Pseudomonas sp. clone HY2 (100%) tidak ditemukan, digantikan oleh keberadaan iron-reducing bacterium clone HN17 (98%) dan uncultured bacteria clone N704B_48 (99%). Iron-reducing bacterium clone HN17 merupakan bakteri pereduksi Fe3+ di ladang padi terkontaminasi arsen di daerah Hunan, Cina. Keberadaan bakteri ini pada lingkungan tercemar minyak dimungkinkan karena bakteri pereduksi besi berperan dalam biodegradasi hidrokarbon (Holba et al. 2004). Uncultured bacteria clone N704B_48 ditemukan di Laut Cina Selatan. Keberadaan bakteri ini dalam hubungannya dengan degradasi minyak belum banyak dilaporkan. Pada substrat tidak steril-2 hari ke-14 sampai hari ke- 28 hanya ditemukan uncultured bacterium clone VH-FL6-50 (100%). Menurut Muyzer et al. (1993) dan Stephen et al. (1999), aplikasi PCR-DGGE hanya dapat mendeteksi 1 - 2 % dari mikroorganisme yang mewakili grup target, sedangkan Macnaughton et al. (1999) menyatakan bahwa metode DGGE tidak dapat digunakan untuk mendeteksi komponen komunitas minor yang mungkin penting dalam degradasi kelas hidrokarbon spesifik. Komunitas bakteri pada substrat steril tanpa penambahan bakteri tunggal maupun konsorsium A, menunjukkan terdapatnya pita iron-reducing bacterium clone HN17 (98%) dan uncultured bacteria clone N704B_48 (99%) yang konsisten sejak hari ke-0 sampai hari ke-28. Alteromonas sp. MOLA 3 (98%) ditemukan pada hari ke-14 sampai ke-28. Alteromonas sp. MOLA 3 merupakan bakteri yang ditemukan dalam penelitian mengenai komunitas bakteri di ekosistem pantai Mediterranian. Keberadaan bakteri ini dalam substrat steril diperkirakan berhubungan dengan kemampuannya mendegradasi senyawa phenanthrene (Zaidi dan Imam 1999). Pita-pita DNA yang terdapat pada profil DGGE (Gambar 2) merupakan bakteri indigenous yang terdapat pada substrat dari perairan Pulau Pari Teluk Jakarta yang tercemar minyak. Komunitas bakteri indigenous di perairan Pulau Pari Teluk Jakarta yang terdeteksi sampai dengan hari ke-28 dirangkum di dalam Tabel 4. Perubahan struktur komunitas bakteri pada substrat dengan penambahan bakteri eksogenous (tunggal dan konsorsium) Gambar 4 menunjukkan suksesi struktur komunitas bakteri selama proses a a bioaugmentasi. Bioaugmentasi adalah penambahan bakteri baik secara tunggal maupun konsorsium pada suatu substrat yang tercemar. Bakteri yang diintroduksikan biasanya bakteri yang telah diuji kemampuannya dalam mendegradasi bahan pencemar. Penambahan bakteri eksogenous pada substrat yang mengandung bakteri indigenous (substrat tidak steril) dapat dilihat pada lajur 5 - 12, sedangkan penambahan bakteri eksogenous pada substrat yang tidak mengandung bakteri indigenous (substrat steril) terdapat pada lajur 13 – 20 (Gambar 4). Tabel 4 Pita bakteri yang terdeteksi pada gel akrilamida (profil DGGE) dalam proses bioremediasi pada substrat tidak steril dan substrat steril tanpa penambahan bakteri (tunggal atau konsorsium) Hari Hari ke-0 Hari ke-7 Hari ke-14 Hari ke-28 Perlakuan Uncultured bacterium Uncultured bacterium Uncultured bacterium Uncultured bacterium clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) Uncultured bacterium Uncultured bacterium Uncultured bacterium Uncultured bacterium Substrat tidak steril 1 clone W1-16 (92%) clone W1-16 (92%) clone W1-16 (92%) clone W1-16 (92%) tanpa penambahan Marine bacterium SCRIPP Marine bacterium SCRIPP Marine bacterium SCRIPP bakteri 413 (98%) 413 (98%) 413 (98%) Uncultured bacterium clone Uday 0-58 (92%) Uncultured bacterium Uncultured bacterium Uncultured bacterium Uncultured bacterium clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) Marine bacterium SCRIPP Marine bacterium SCRIPP Substrat tidak steril 2 413 (98%) 413 (98%) tanpa penambahan Iron-reducing bacteria Pseudomonas sp. clone bakteri clone HN17 (98%) HY2 (100%) Uncultured bacterium N704B_48 (98%) Iron-reducing bacteria Iron-reducing bacteria Iron-reducing bacteria Iron-reducing bacteria clone HN17 (98%) clone HN17 (98%) clone HN17 (98%) clone HN17 (98%) Substrat steril tanpa Uncultured bacterium Uncultured bacterium Uncultured bacterium Uncultured bacterium penambahan bakteri N704B_48 (98%) N704B_48 (98%) N704B_48 (98%) N704B_48 (98%) Alteromonas sp. MOLA3 Alteromonas sp. MOLA3 (100%) (100%) 25 a a a a b c b c d f e e Br Al Bakteri Kontrol Bc 0 7 14 28 Substrat tidak steril + strain RCO/B/08_008 0 7 14 28 Substrat tidak steril + konsorsium A g g h i f h i i j j Pb a 0 7 14 28 Substrat steril + strain RCO/B/08 008 j 0 7 14 28 hr Substrat steril + konsorsium A Gambar 4 Profil DGGE pada proses bioremediasi dalam substrat tidak steril dan substrat steril dengan penambahan bakteri (tunggal atau konsorsium), bakteri kontrol: Pb: RCO/B/08_006 (Pseudomonas balearica st101 99%), Br: RCO/B/08_013 (Bordetella sp. F2 99%), Al: RCO/B/08_008 (Alcanivorax sp. TE-9 100%) Bc: RCO/B/08_009 (Bacillus sp. L41 99%); Pita yang terdeteksi: a: Marine bacterium SCRIPP 413 (98%), b: Uncultured bacterium clone VH-FL6-50 (100%), c: Pseudomonas sp. clone HY2 (98%), d: Uncultured gamma-proteobacterium (99%), e: Bordetella sp. F2 (98%), f: Bacillus sp. L41 (99%), g: Uncultured Bordetella sp. (100%), h: Alcanivorax sp. TE9 (99%), i: Iron-reducing bacterium clone HN-HFO91 (98%), j: Uncultured Firmicuted bacteria (100%) Pada profil DGGE Gambar 4 lajur 5 – 8, pita bakteri RCO/B/08_008 tidak terdeteksi. Hal ini dapat disebabkan karena bakteri eksogenous yang ditambahkan tidak dapat berkompetisi jumlahnya dengan komunitas bakteri indigenous. Tidak semua bakteri eksogenous dapat hidup dan berkembang pada suatu lingkungan walaupun diisolasi dari substrat dengan karakter yang sama dengan lingkungan yang diremediasi (Macnaughton et al. 1999). Selain itu, tidak terdeteksinya RCO/B/08_008 sejak hari ke-0 dapat disebabkan karena jumlah inokulum yang kurang sesuai. Berdasarkan Kusuma (2009), RCO/B/08_008 diinokulasikan pada substrat dengan kepadatan 1 x 108 sel/ml. Pita RCO/B/08_008 tidak terdeteksi di dalam profil DGGE. Namun, hasil penelitian sebelumnya (Kusuma 2009) menunjukkan bahwa penambahan RCO/B/08_008 (Alcanivorax sp. TE9 100%) menurunkan konsentrasi minyak pada substrat sebesar 89% (b/b) setelah diinkubasi 28 hari. Penurunan ini relatif lebih besar dibandingkan pada substrat tidak steril tanpa penambahan bakteri eksogenous. Hal ini menunjukkan bahwa pemberian RCO/B/08_008 memberi pengaruh yang signifikan terhadap degradasi minyak di dalam substrat walaupun mungkin tidak secara langsung. Diasumsikan bahwa aktivitas degradasi minyak pada substrat tersebut didominasi oleh bakteri indigenous, namun hal ini perlu diuji lebih lanjut. Berbeda dengan hasil yang diperoleh pada substrat yang mengandung bakteri indigenous, pada substrat yang tidak mengandung bakteri indigenous, terdapat pita Alcanivorax sp. TE9 (99%) yang dominan sejak hari ke-0 sampai dengan hari ke-28. Bakteri Alcanivorax sp. TE9 (99%) dimungkinkan merupakan bakteri eksogenous yang telah mengalami mutasi. Vogel et al. (1996) menyatakan bahwa stress lingkungan dapat meningkatkan terjadinya mutasi. Seperti diketahui bahwa RCO/B/08_008 mempunyai kesejajaran 100% dengan Alcanivorax sp. TE9 dalam GenBank NCBI. Terdapatnya pita Alcanivorax sp. TE9 (99%) pada substrat steril dan tidak terdeteksinya pita tersebut pada substrat tidak steril menunjukkan bahwa bakteri tersebut tidak dapat berkompetisi dengan bakteri indigenous. Dalam Kusuma (2009) penurunan konsentrasi minyak pada substrat ini adalah sebesar 68% (b/b), lebih rendah dibandingkan dengan penambahan RCO/B/08_008 pada substrat yang mengandung bakteri indigenous (89%), namun lebih tinggi dibandingkan dengan hanya bakteri indigenous saja (44%). Hal ini menunjukkan bahwa penambahan RCO/B/08_008 memberi pengaruh bagi penurunan konsentrasi minyak dalam substrat. Gambar 5a dan b menunjukkan bahwa total biomassa sel yang terdeteksi pada substrat yang tidak steril lebih rendah daripada total biomassa sel pada substrat steril. Pada substrat yang tidak steril, total biomassa sel selama 28 hari didominasi oleh uncultured bacterium clone VH-FL6-50 (100%), namun pada hari ketujuh muncul marine bacterium SCRIPP 413 (98%) (Gambar 4). a b Gambar 5 Hubungan antara total biomassa sel, penurunan konsentrasi minyak, dan penambahan strain tunggal RCO/B/08_008 (Kusuma 2009). (a) substrat tidak steril; (b) substrat steril; : total biomassa : berat minyak Beberapa jenis bakteri lain tidak dapat terdeteksi pada profil DGGE karena kemungkinan jumlahnya relatif sedikit. Total biomassa bakteri pada substrat steril (Gambar 5b) didominasi oleh Alcanivorax sp. TE9 (99%) pada hari ke-0 dan ketujuh, sedangkan total biomassa sel pada hari ke-14 diperkirakan didominasi oleh Alcanivorax sp. TE9 (99%), marine bacterium SCRIPP 413 (98%), Bacillus sp. L41 (99%) dan iron-reducing bacteria clone HN-HFO91 (98%) (Gambar 4). Lebih tingginya penurunan konsentrasi minyak pada substrat tidak steril-1 dengan penambahan RCO/B/08_008 dibandingkan pada substrat steril dengan penambahan RCO/B/08_008 menunjukkan bahwa strain tersebut, meskipun tidak terdeteksi oleh teknik PCR-DGGE, namun mampu mengaktivasi atau memacu bakteri indigenous untuk melakukan penguraian minyak menjadi komponen-komponennya. Pada substrat tidak steril dengan penambahan konsorsium A (Gambar 4), bakteri indigenous yang terdiri atas marine bacterium SCRIPP 413 (98%), uncultured bacterium clone VH-FL6-50 (100%) dan Pseudomonas sp. clone HY2 (98%) mendominasi pada hari ke-0. Di samping itu terdeteksi juga bakteri eksogenous Bordetella sp. F2 (98%) dalam intensitas yang relatif rendah. Bordetella sp. F2 (98%) diduga merupakan hasil mutasi dari bakteri eksogenous RCO/B/08_013 (Bordetella sp. F2, 99%) akibat perubahan lingkungan. Pada hari ketujuh Bordetella sp. F2 (98%) tidak terdeteksi lagi dan susbtrat didominasi oleh uncultured bacterium clone VH-FL6-50 (100%) dan Pseudomonas sp. clone HY2 (98%), sementara marine bacterium SCRIPP 413 (98%) digantikan oleh uncultured gamma proteobacterium clone 1 T12d-oil (99%). Bakteri ini bersama dengan uncultured bacterium clone VH-FL6-50 (100%) tetap terdeteksi sampai hari ke-28. Uncultured gamma proteobacterium clone 1 T12d-oil merupakan salah satu bakteri yang ditemukan oleh Paisse et al. (2010) terdapat pada sedimen Etang-de-berre Lagoon yang tercemar minyak. Pada hari ke-28 juga terdapat bakteri uncultured Firmicuted bacteria (100%), yang menandakan terjadinya degradasi hidrokarbon aromatik secara anaerob (Kunapulli et al. 2008). Gambar 4 lajur 17 sampai 20 menggambarkan perubahan struktur komunitas bakteri yang terjadi pada perlakuan substrat steril dengan penambahan konsorsium A. Pada substrat ini, bakteri indigenous marine bacterium SCRIPP 413 (98%), uncultured bacterium clone VH-FL6-50 (100%) dan Pseudomonas sp. clone HY2 (98%) terdeteksi pada hari ke-0. Uncultured Bordetella sp. clone ud3b (100%) terdeteksi pada hari ke-0 dan meningkat pada hari ketujuh. Selain itu Alcanivorax sp. TE-9 (99%) ditemukan pula pada hari ketujuh dalam intensitas yang relatif tinggi. Hal ini diperkirakan karena kemampuan Alcanivorax sp. TE-9 dalam mendegradasi senyawa-senyawa PAH seperti fluoranthene, fenanthrene, naphtalene, pyrene, phenotiazene, dibenzothiophene, fluorene dan Arabian Light Crude Oil (Hatmanti dan Darmayati 2009, Darmayati 2009). Uncultured Bordetella sp. clone ud3b merupakan bakteri yang berperan dalam mineralisasi 1,2,4-trichlorobenzene (Fang et al. 2008), sedangkan Alcanivorax sp. merupakan bakteri kunci dalam degradasi hidrokarbon alifatik dan aromatik (Kasai et al. 2001; Roling et al. 2004), khususnya di lingkungan laut (Harayama et al. 2004). Selain itu pada hari ketujuh terlihat pula adanya bakteri iron-reducing bacteria HN-HFO91 (98%) yang dimungkinkan berperan pada degradasi senyawa benzene dalam substrat tersebut. Pada hari ke-14 Alcanivorax sp. TE-9 (99%) tetap bertahan dalam jumlah yang relatif tinggi, marine bacterium SCRIPP 413 (98%) kembali terdeteksi dan terdapat pertumbuhan Bacillus sp. L41 yang berperan dalam degradasi pyrene, phenanthrene, dibenzothiophene, fluorene dan ALCO (Darmayati 2009; Hatmanti dan Darmayati 2009). Pada hari ke-28 Alcanivorax sp. TE-9 (99%) masih tetap bertahan bersama iron-reducing bacterium HNHFO91 (98%). Pada hari ke-28 ini terdapat uncultured Firmicuted bacteria BC_COM467 (100%) yang menandakan kondisi substrat menjadi anaerob (Abu 2010). Tabel 5 merangkum perubahan struktur komunitas bakteri pendegradasi minyak di perairan Pulau Pari Teluk Jakarta setelah dilakukan penambahan bakteri eksogenous dan diinkubasi sampai 28 hari. Gambar 6 menunjukkan perbandingan antara total biomassa sel dan penurunan konsentrasi minyak. Penurunan konsentrasi minyak pada tiga perlakuan substrat selama 28 hari bervariasi. Konsentrasi minyak dalam substrat secara alami mengalami penurunan sampai 56% (Gambar 6a), sedangkan pada substrat yang mengandung bakteri indigenous dengan penambahan konsorsium A adalah sebesar 71% (Gambar 6b) dan pada substrat dengan penambahan bakteri eksogenous tanpa bakteri indigenous hanya mencapai 40% (Gambar 6c). Berdasarkan Gambar 6a diketahui bahwa tanpa penambahan bakteri eksogenous, total biomassa sel bertambah tidak terlalu tinggi, namun dengan penambahan konsorsium A, maka jumlah sel bakteri tersebut mengalami peningkatan (Gambar 6b dan 7c). Jika dilihat dari penurunan konsentrasi minyaknya, maka penambahan konsorsium A pada substrat yang mengandung bakteri indigenous (Gambar 6b) adalah yang tertinggi, yaitu 71%. Hal ini diperkirakan karena terdapatnya interaksi positif antara bakteri eksogenous dengan bakteri indigenous pada substrat, yang bekerja saling menguntungkan dalam menguraikan minyak. Pada substrat yang tidak mengandung bakteri indigenous, penurunan konsentrasi minyaknya justru lebih rendah bila dibandingkan substrat tidak steril tanpa penambahan konsorsium A (hanya bakteri indigenous saja). Hal ini diperkirakan karena tanpa bakteri indigenous, bakteri eksogenous yang diintroduksikan tidak dapat menguraikan minyak secara maksimal. a b Gambar 6 c Hubungan antara total biomassa sel, penurunan konsentrasi minyak, dan penambahan konsorsium A (Arifah 2010). (a) substrat tidak steril tanpa penambahan konsorsium A; (b) substrat tidak steril dengan penambahan konsorsium A; (c) substrat steril dengan penambahan konsorsium A. Best et al. (1988) menyatakan bahwa ada beberapa macam interaksi di dalam suatu komunitas, antara lain : (i) interaksi antar anggota komunitas di mana yang satu tidak mampu menghasilkan salah satu faktor tumbuh, sementara yang lain memenuhi melalui aktivitas metaboliknya; (ii) interaksi di mana salah satu atau lebih anggota komunitas menghasilkan senyawa metabolit yang menghalangi pertumbuhan anggota lainnya; (iii) interaksi di mana terdapat anggota komunitas yang menghasilkan berbagai enzim untuk meningkatkan metabolisme kooperatif antar anggota; (iv) interaksi di mana terdapat anggota komunitas baru yang berasal dari transfer plasmid antar spesies yang resisten terhadap polutan baru. Pada profil DGGE masih terdapat pita hasil amplifikasi dari substrat yang telah disterilisasi (diautoklaf). Hal ini menunjukkan bahwa proses sterilisasi tidak merusak semua DNA yang ada di dalam substrat. Esser et al. (2006) menyatakan bahwa sterilisasi menggunakan autoklaf dapat mendegradasi molekul DNA menjadi fragmen-fragmen berukuran 20-30 pasang basa. Namun, beberapa molekul DNA berukuran besar masih dapat bertahan sehingga masih dapat diamplifikasi menggunakan PCR bersensitifitas tinggi. Komposisi bakteri yang terdeteksi dalam komunitas bakteri dari perairan Pulau Pari Teluk Jakarta Berdasarkan hasil sekuensing yang dibandingkan dengan seluruh data yang terdapat di bank data (www.ncbi.nlm.nih.gov) menggunakan program BLAST diketahui bahwa dari 70 pita yang terdeteksi pada profil DGGE dalam penelitian ini, 45 pita merupakan bakteri yang belum dapat dikultur dan 25 merupakan bakteri yang dapat diisolasi dan telah diketahui taksonominya (Gambar 4). Terdapatnya lebih banyak pita yang teridentikasi sebagai uncultured bacterium menunjukkan bahwa di lingkungan yang tercemar minyak masih banyak terdapat bakteri yang belum dapat dikultur atau diisolasi. Bakteri-bakteri tersebut kemungkinan mempunyai peranan tertentu dalam komunitasnya. Gambar 7 Komposisi komunitas bakteri dari Perairan Pulau Pari Teluk Jakarta berdasarkan kemampuan pengkulturan. Tabel 5 Pita bakteri yang terdeteksi pada gel akrilamida (profil DGGE) dalam proses bioremediasi pada substrat tidak steril dan substrat steril dengan penambahan bakteri (tunggal atau konsorsium) Hari Hari ke-0 Hari ke-7 Hari ke-14 Hari ke-28 Perlakuan Uncultured bacterium Uncultured bacterium Uncultured bacterium Uncultured bacterium clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) Substrat tidak steril 1 clone VH-FL6-50 (100%) Marine bacterium SCRIPP + RCO/B/08_008 413 (98%) Uncultured bacterium Uncultured bacterium Uncultured bacterium Uncultured bacterium clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) clone VH-FL6-50 (100%) Marine bacterium SCRIPP Pseudomonas sp. clone Uncultured gamma Uncultured gamma Substrat tidak steril 2 HY2 (98%) 413 (98%) proteobacterium (99%) proteobacterium (99%) + Konsorsium A Uncultured gamma Uncultured Firmicuted Pseudomonas sp. clone bacteria BC_COM467 HY2 (100%) proteobacterium (99%) (100%) Bordetella sp. F2 (98%) Alcanivorax sp. TE9 (99%) Alcanivorax sp. TE9 (99%) Alcanivorax sp. TE9 (99%) Alcanivorax sp. TE9 (99%) Marine bacterium SCRIPP Iron-reducing bacteria clone HN17 (98%) 413 (98%) Substrat steril + Bacillus sp. L41 (99%) RCO/B/08_008 Uncultured Firmicuted bacteria BC_COM467 Iron-reducing bacteria (100%) clone HN-HFO91 (98%) Marine bacterium SCRIPP Alcanivorax sp. TE9 (99%) Alcanivorax sp. TE9 (99%) Alcanivorax sp. TE9 (99%) 413 (98%) Marine bacterium SCRIPP Iron-reducing bacteria Uncultured Bordetella sp. (100%) 413 (98%) clone HN17 (98%) Uncultured bacterium clone VH-FL6-50 (100%) Substrat steril + Bacillus sp. L41 (99%) Iron-reducing bacteria Uncultured Firmicuted konsorsium A clone HN-HFO91 (98%) bacteria BC_COM467 Pseudomonas sp. clone HY2 (100%) (100%) Uncultured Bordetella sp. (100%) 33 Dari 25 bakteri yang dapat dikultur, filum proteobakteria mendominasi yaitu sebanyak 20 isolat dan sisanya adalah Firmicutes yaitu 5 isolat (Gambar 5). Hal ini mirip dengan hasil penelitian Paisse et al. (2008) yang menyatakan bahwa analisis komposisi komunitas bakteri di Berre lagoon Prancis didominasi oleh Proteobacteria phylum (γ-, δ-, α-, ε- dan β-proteobacteria), Bacteroidetes and Verrucomicrobium groups and Spirochaetes, Actinobacteria and Cyanobacteria. Menurut Madigan et al. (2003), proteobakteria merupakan filum yang mempunyai anggota terbesar dalam kingdom bakteri. Proteobakteria terdiri atas beberapa kelas yaitu α, β, γ, δ dan ε-proteobakteria. Gambar 8 Komposisi komunitas bakteri dari Perairan Pulau Pari Teluk Jakarta berdasarkan filum. Berdasarkan kelasnya, komunitas bakteri di Pulau Pari didominasi oleh γ−proteobacteria (16 isolat), diikuti oleh β−proteobacteria (4) dan Bacilli (2). Hal ini mirip dengan Thontowi (2008) yang menyebutkan bahwa kelas γproteobakteria dan α-proteobakteria mendominasi bakteri yang ada di Pulau Pari. Hal ini sesuai dengan komposisi bakteri-bakteri laut pada umumnya (Harayama et al. 1999; Yakimov et al. 2003). Berbeda dengan Harwati et al. (2007) yang menyebutkan bahwa keragaman bakteri pendegradasi minyak di perairan laut Semarang didominasi oleh α-proteobakteria. Gambar 8 Komposisi komunitas bakteri dari Perairan Pulau Pari Teluk Jakarta berdasarkan kelas. Rekapitulasi hasil sekuensing pita-pita dari profil DGGE pada penelitian ini dapat dilihat pada Lampiran 1, sedangkan format fasta sekuen fragmen DNA hasil sekuensing tercantum dalam Lampiran 2. SIMPULAN DAN SARAN Simpulan 1. Komunitas bakteri dalam lingkungan tercemar di perairan Pulau Pari Teluk Jakarta didominasi oleh Marine bacterium SCRIPP 413, uncultured bacterium clone VH-FL6-50 dan uncultured bacterium clone W1-16. Beberapa jenis bakteri terdeteksi pada hari ke-7 dan 14, namun kemudian tidak terdeteksi sampai hari ke-28. 2. Dominansi bakteri eksogenous (RCO/B/08_008, RCO/B/08_006, dan RCO/B/08_013) terdeteksi hanya di dalam substrat yang tidak mengandung bakteri indigenous (substrat steril). Namun, pada substrat yang mengandung bakteri indigenous (substrat tidak steril), bakteri eksogenous tidak terdeteksi. Hal ini menunjukkan bahwa bakteri eksogenous yang ditambahkan tidak kompetitif dan tidak efisien digunakan sebagai agen bioremediasi. Saran 1. Untuk mengetahui peranan uncultured bacteria dalam komunitas bakteri pendegradasi minyak perairan Pulau Pari Teluk Jakarta, perlu dilakukan penelitian lebih lanjut dengan menggunakan primer spesifik gen fungsional misalnya gen penyandi alkana monooksigenase (gen AlkB). 2. Perlu dilakukan analisis tentang produk-produk hasil dari degradasi minyak, untuk mengetahui peranan bakteri dalam bioremediasi minyak di lingkungan yang tercemar minyak. DAFTAR PUSTAKA Abu LN, Selesi D, Rattei T, Tischler P, Meckenstock RU. 2010. Identification of enzymes involved in anaerobic benzene degradation by a stricktly anaerobic iron-reducing enrichment culture. Environ. Microbiol. 12: 27832796. Amann RI, Ludwig W, Schleifer KH. 1995. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiological Reviews 59, 143–169. Arifah F. 2010. Pemilihan formulasi konsorsium bakteri untuk mendegradasi minyak mentah pada sedimen pantai Pulau Pari Kepulauan Seribu [Skripsi]. Yogyakarta: Fakultas Biologi Universitas Gajahmada. Barnum S. 2005. Biotechnology: an introduction, 2nd edition. United States: Miami University Thomson Brooks/Cole. Pages : 138 – 139. Baker PW, Harayama S, editor. 2004. An analysis of microorganisms in environments using denaturing gradient gel electrophoresis (DGGE). Di dalam: Walker JM, Spencer JFT, Spencer ALR, editor. Method in biotechnology: environmental biology: method and protocols. hlm 323 – 338. Barbeau K, Zhang G, David HL, Alison B. 2002. Petrobactin, a Photoreactive Siderophore Produced by the Oil-Degrading Marine Bacterium Marinobacter hydrocarbonoclasticus. J. Am. Chem. Soc., 2002, 124 (3), pp 378–379. Best DJ, Jones J, Stafford D. 1988. The Environment and Biotechnology. Di dalam: Higgins J, Best DJ, Jones J, editor. Biotechnology: principles and application. 2nd Edition. Oxford : Blackwell Scientific Publications. hlm 213256. Bruns A, Corty LB. 1999. Fundibacter jadensis gen. nov, sp.nov., a new slightly halophilic bacterium, isolated from intertidal sedimen. Int. J. Systematic Bacteriol 49 : 441 – 448. Budhiarto A. 2009. Crude Distilation Unit (CDU). Di dalam: Teknologi Proses Kilang Minyak Bumi. Jakarta: MIGAS. Darmayati Y. 2003. Marine petroleum hydrocarbon-degrading bacteria distribution in Malaka Strait. Proceeding of Annual Meeting of Indonesian Sociecty for Microbiology II : 555 – 562. Darmayati Y, Harayama S, Yamazoe A, Hatmanti A, Sulistiani, Nuchsin R, Kunarso DH. 2008. Hydrocarbonoclastic bacteria from Jakarta Bay and Seribu Islands. Marine Research in Indonesia, 33: 55 – 64. Darmayati Y. 2008. Pemanfaatan bakteri laut dalam bioremediasi hidrokarbon di perairan pantai: screening dan optimalisasi pertumbuhan. Pusat Penelitian Oseanografi LIPI. Jakarta. Darmayati Y. 2009. Seleksi dan karakterisasi bakteri laut pendegradasi minyak. Dipresentasikan di Seminar Nasional Perikanan 2009. STP Jakarta. Elhafi G, Naylor CJ, Savage CE, Jones RC. 2004. Microwave or autoclave treatments destroy the infectivity of infectious bronchitis virus and avian pneumovirus but allow detection by reverse transcriptase-polymerase chain reaction. Avian Pathology 33: 303-306. Esser KH, Marx WH, dan Lisowsky T. 2006.DNA decontamination: DNAExitusPlus in comparison with conventional reagents. Nature Methode 3. Fahruddin. 2004. Dampak tumpahan minyak pada biota laut. Career Development Network, Faculty of Engineering University of Indonesia. Jakarta. Feliatra. 1998. Isolation, identification and biodegradation of petroleum by bacteria in Malacca Strait. Di dalam: proceeding of the first Indonesian Seminar on Marine Biotechnology’98. Soemodihardjo et al., editor. Jakarta, Widya Graha-LIPI, 14-15 Oktober 1998. hal: 291 – 303. Glick BR, Pasternak JJ. 2003. Molecular biotechnology: principles and applications of recombinant DNA, 3rd edition. Washington United States: American Society for Microbiology. pages : 378 – 379. Haghstrom A, Pommier T, Rohwer F, Simu K, Stolte W, Svensson D, Zweifel UL. 2002. Use of 16S ribosomal DNA for delineation of marine bacterioplankton species. Appl Environ Microbiol 68:3628-3633. Harayama S, Kasai Y, Hara A. 2004. Microbial communities in oil-contaminated seawater. Current Opinions in Biotechnology 15: 205–214. Hariyadi R. 2009. Bakteri Pengunyah Minyak. Gatra Nomor 22 [Terbit Kamis, 9 April 2009]. Hatmanti A, Darmayati Y. 2009. Karakterisasi dan analisis kekerabatan bakteri potensial pendegradasi minyak dan poly-aromatics hydrocarbon (PAH) dari Perairan Teluk Jakarta. Di dalam : Prosiding Seminar Nasional Biologi 2009, Purwokerto : Universitas Jenderal Soedirman, 12 Desember 2009. Harwati UT, Kasai Y, Kodama Y. Susilaningsih D, Watanabe K. 2007. Characterization of diverse hydrocarbon-degrading bacteria isolated from Indonesia Seawater. Microb Environ 22:1-4. Holba AG, Wright L, Levinson R, Huizinga, B, Scheihing M. 2004. Effects and impact of early-stage anaerobic biodegradation on Kuparuk River Field, Alaska. Geological Society Special Publications 237: 53-88. Hold GL, Smith EA, Rappe MS, Maas EW, Moore ERB, Stroempl C, Stephen, JR, Prosser JI, Birkbeck TH, Gallacher S. 2001. Characterization of bacterial communities associated with toxic and non-toxic dinoflagellates: Alexandrium spp. Microbiol. Ecol. 37 (2), 161-173. Kasai Y, Kishira H, Harayama S. 2002. Bacteria belonging to the genus Cycloclasticus play primary role in the degradation of aromatic hydrocarbon released in marine environment. Appl. Environ. Microbiol. 69 : 5625 – 5633. Kumar S, Tamura K, Nei M. 2004. MEGA 3: Integrated software for molecular evolutionary genetics analysis and sequence alignment. Briefings Bioinformatics 5:150-163. Kunapuli U, Lueders L, Meckenstock RU. 2007. The use of stable isotope probing to identify key iron reducing microorganisms involved in anaerobic benzene degradation. ISME J. 1, 643-653. Kunapuli U, Griebler C, Beller HR, Meckenstock RU. 2008. Identification of intersubstrattes formed during anaerobic benzene degradation by an ironreducing enrichment culture. Environ. Microbiol. 10, 1703-1712. Kusuma DH. 2009. Kemampuan lima isolat bakteri hidrokarbonoklastik sebagai agensia bioremediasi lingkungan tercemar minyak mentah di Pulau Pari, Kepulauan Seribu [Skripsi]. Yogyakarta: Univ Gadjahmada. Leahy JG, Collwell RR. 1990. Microbial degradation of hydrocarbons in the environment. Microbiological Review 54:305-315. Liu WT, Marsh TL, Cheng H, Forney LJ. 1997. Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA. Appl. Environ. Microbiol. 63:4516–4522. Liu J, Lee LS, Nies LF, Nakatsu CH, Turco RF. 2007. Biotransformation of 8:2 fluorotelomer alcohol in soil and by soil bacteria isolates. Env. Sci. Technol. 41:8024-8030. Lo GA, Michaud L. 2010. Occurrence and characterization of psychrotolerant hydrocarbon-oxidizing bacteria from surface seawater along the Victoria Land coast (Antarctica). Polar Biol. 33 (7), 929-943. Luz AP, Pellizari VH, Whyte LG, Greer CW. 2004. A survey of indigenous microbial hydrocarbon degradation genes in soils from Antarctica and Brazil. Can. J. Microbiol. 50:323-333. Macnaughton SJ, Stephen JR, Venosa AD, Davis GA, Chang YJ, White DC. 1999. Microbial population changes during bioremediation of an experimental oil spill. Appl. and Environ. Microbiol. 65: 3566 – 3574. Muyzer G, De Waal HR, Uiterlinden AG. 1993. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Appl. Environ Microbiol 59: 695 – 700. Muyzer G, Hottentrager S, Teske A, Wawer C. 1996. Denaturing gradient gel electrophoresis of PCR-amplified 16s rDNA – a new molecular approach to analyse the genetic diversity of mix microbial communities. Di dalam: Molecular Microbial Ecology Manual. Akkermans ADL, Elsas JD van, Bruijn de, editor. pp 1-23. Dordrecht: Kluwer Academic Publishers. Muyzer G. 2000. Genetic fingerprinting of microbial communities – present status and future perspective. Di dalam : Microbial. Biosystem : New Frontiers. Proceedings of the 8th International. Symposium on Microbial. Ecology. Bell CR, Brylinski M, Johnson-Green P, editor. Canada: Atlantic Canada Society for Microbial Ecology. hal : 503-572 Muyzer G, Brinkhoff T, Nubel U, Santegoeds C, Schafer H, Wawer C. 2004. Denaturing gradient gel electrophoresis (DGGE) in microbial ecology. Molecular microbial ecology manual, 2nd edition. Netherlands: Kluwer Academic Publishers. hal 743 – 770. Muyzer G, Smalla K. 1998. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology. Antonie van Leeuwenhoek 73: 127-141. Nakatsu CH. 2007. The basics and application of denaturing gradient gel electrophoresis for soil microbial community analysis. Soil Sci. Soc. J. Am. 71:562-571. Nicodem DE, Fernandes MC, Guedes CLB, Correa RJ. 1997. Photochemical processes and the environmental impact of petroleum spills. Biogeochemistry 39:121-138. Nugroho A. 2006. Bioremediasi Hidrokarbon Minyak Bumi. Yogyakarta, Graha Ilmu, 160 hal. Nurhariyati T, Ni’matuzzahroh, Surtiningsih T. 2006. Biodegradasi minyak oleh rhodotorula dan candida hasil isolasi dari Pelabuhan Tanjung Perak Surabaya. Berkala Penelitian Hayati 12: 27-31. Paissé S, Coulon F, Goñi-Urriza M, Peperzak L, McGenity TJ, Duran R. 2008. Structure of bacterial communities along a hydrocarbon contamination gradient in a coastal sediment. FEMS Microbiol Ecol. 66:295-305. Païssé S, Goñi-Urriza M, Coulon F, Duran R. 2010. How a bacterial community originating from a contaminated coastal sediment responds to an oil input. FEMS Microb Ecol. 60:394-405. Pramudianto B. 1999. Sosialisasi PP No.19/1999 tentang pengendalian pencemaran dan atau perusakan laut. Di dalam : Prosiding Seminar Sehari Teknologi dan Pengelolaan Kualitas Lingkungan Pesisir dan Laut. Bandung: Jurusan Teknologi Lingkungan ITB. Prince RC, Bare RE, Garrett RM, Grossman MJ, Haith CE, Keim LG, Lee K, Holtom GJ, Lambert P, Sergy GA, Owens EH and Guénette CC. 2003. Bioremediation of stranded oil on an Arctic shoreline. Spill Science and Tech Bull 8:303 – 312. Ratcliff R. 2009. Petro-clear hydrocarbon and pesticide remediation: microbial solutions. http://www.virtualviz.com/petrochemical.htm [15 Mei 2009] Rolling WF, Milner MG, Jones DM, Lee K, Daniel F, Swannell RJ, Head IM. 2002. Robust hydrocarbon degradation and dinamycs of bacterial communities during nutrient-enhanced oil spill bioremediation. Appl. Environ. Microbiol 68:5537 – 5548. Rolling WF, Milner MG, Jones DM, Fratepietro F, Swannell RP, Daniel F, Head IM. 2004. Bacterial community dynamics and hydrocarbon degradation during a field-scale evaluation of bioremediation on a mudflat beach contaminated with buried oil. Appl Environ Microbiol 70:2603-2613. Rossello-Mora R, Thamdrup B, Schäfer H, Weller R, Amann R. 1999. The response of the microbial community of marine sediments to organic carbon input under anaerobic conditions. Syst. Appl. Microbiol. 22:237-248. Schafer H, Muyzer G. 2001. Denaturing gradient gel electrophoresis in marine microbial ecology. Di dalam: Methods in microbiology Vol 30. America: Academic Press Ltd. hal 423 – 465. Stephen JR, et al. 1999. Microbial Characterization of a JP-4 fuel contaminated site using a combined lipid biomarker/PCR-DGGE based approach. Environ Microbiol 1:231-243. Sudrajat. 1996. Karakteristik Limbah Minyak dan Pelaksanaan Bioremediasi. Samarinda. PPLH. Syakti AD. 2008. Multi-proses Resubstratsi di dalam Penanganan Tumpahan Minyak (Oil Spill) di Perairan Laut dan Pesisir. http://www_pksplipb_or_idSEMINAR BIOREMEDIASI.htm [29 Mei 2009]. Syutsubo K, Kishira H, Harayama S. 2001. Development of specific oligonucleotide probes for the identification and in situ detection of hydrocarbon-degrading Alcanivorax strains. Environ Microbiol 3:371-379. Teramoto M, Suzuki M, Okazaki F, Hatmanti A, Harayama S. 2009. Oceanobacter-related bacteria are important for the degradation of petroleum aliphatic hydrocarbons in the tropical marine environment. Microbiology 155:3362-3370. Thontowi A. 2008. Potensi bakteri pendegradasi hidrokarbon alkana sebagai agen bioremediasi pencemaran minyak di laut Indonesia [Tesis]. Bogor: Institut Pertanian Bogor. United-tech. 2009. Bioaugmentation vs indigenous microbial populations for waste remediation projects. http://www.united-tech.com/bioaugmentation .html. [29 Mei 2009] Venosa AD and Zhu X. 2003. Biodegradation of Crude Oil Contaminating Marine Shorelines and Freshwater Wetlands. Spill Science & Tech Bull 8(2): 163– 178. Whyte LG, Bourbonniere L, Greer CW. 1997. Biodegradation of petroleum hydrocarbons by psycrotrophic Pseudomonas strains possesing both alkane (alk) and naphthalene (nah) catabolic pathways. App. Environ. Microbiol. 63: 3719 – 3723. Whyte LG, Greer CW, Inniss EW. 1999. Assessment of the biodegradation potential of psychrotrophic microorganisms. Can J Microbiol 42:99-106. Whyte LG et al. 2002. Prevalence of Alkane Monooxygenase Genes in Arctic and Antarctic Hydrocarbon-Contaminated and Pristine Soils. FEMS Microbiol. Ecol. 41: 141-150. Wilkinsons SG. 1988. Gram-negative bacteria. Di dalam: Microbial Lipids. Ratledge, Wilkinsons SG, editor. London, England: Academic Press. p.299488 Yakimov MM, Golyshin PN, Lang S, Edward R, Moore B, Abraham WR, Lunsdorf H, Timmis KN. 1998. Alcanivorax borkumensis gen nov., sp nov., a new hydrocarbon-degrading and surfactant-producing marine bacterium. IJSM 48:339-348. Yakimov MM,.Timmis KN, Golyshin PN. 2007. Obligate oil-degrading marine bacteria. Curr Opin. Biotech 18:1-10. Zaidi BR, Imam SH. 1999. Factors affecting microbial degradation of polycyclic aromatic hydrocarbon phenanthrene in the Caribbean coastal water. Marine Pollution Bulletin 38:737-742. Zhang R, Liu B, Lau SC, Ki JS, Qian PY. 2007. Particle-attached and free-living bacterial communities in a contrasting marine environment: Victoria Harbor, Hong Kong. Microbiol Ecol. 61:496-508. Zhuang WQ, Tay JH, Maszenan AM, Tay ST. 2003. Isolation of naphthalenedegrading bacteria from tropical marine sediments. Water Sci Technol 47: 303-308. Zhu X, Venosa AD, Suidan MT, Lee K. 2001. Guideline for the bioremediation of marine shorelines and freshwater wetlands. Cincinnati: U.S. Environmental Protection Agency. 156 pp. LAMPIRAN Lampiran 1 Rekapitulasi hasil sekuensing pita DGGE No. 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 13. 14. 15. 16. 17. Nama Sampel A0 (4) A28 (2) 8‐14 (2) 8‐14 (3) 8‐28 (3) A‐0 (1) A‐0 (2) A‐0 (3) A‐0 (4) A‐14 (2) A‐28 (3) K814 (3) K814 (4) KA0 (3) KA7 (3) K‐7 (2) K‐14 (1) Iden‐ tity DQ453689.1 Bordetella sp F2 98% FM242405.1 Uncultured gamma proteobacterium 99% AB055207.1 Alcanivorax sp TE‐9 99% FJ269102.1 Iron‐reducing bacterium clone HN‐HFO91 100% EU369166.1 Uncultured bacterium clone MBFOS‐07 100% AF359548.1 Marine bacterium SCRIPPS_413 98% HQ190475.1 Uncultured bacterium clone BR130 99% EF379688.1 Uncultured bacterium clone VH‐FL6‐50 100% FJ529835.1 uncultured Bordetella sp 100% DQ249996.1 Bacillus sp. L41 99% HQ727565.1 uncultured Firmicutes bacterium BC_COM467 100% FJ545511.1 Uncultured bacterium clone W1‐16 92% HQ011565.1 Uncultured bacterium clone Uday0‐58 100% GU176052.1 Pseudomonas sp. clone HY2 100% FJ269053.1 iron‐reducing bacterium clone HN17 98% GU941107.1 Uncultured bacterium clone N704B_48 99% EF061427.1 Alteromonas sp. MOLA 3 100% Kode Akses Nama Isolat Lampiran 2 Data sekuen masing-masing Isolat dalam format fasta >Bordetella sp. F2 (98%) GGGGGTTTGGAATGGGGGGCACCCTGATCCAGCCATCCCGCGTGTGCGATGAAGG CCTTCGGGTTGTAAAGCACTTTTGGCAGGAAAGAAACGGCCCTGGTTAATACCTG GGGCAACTGACGGTACCTGCAGAATAAGCACCGGCTAACTACGTGCCAGCAGCCG CGGTAATACGTAGGGTGCAAGCGTTAATCGGAATTACTGGGCGTAAAGCGTGCGC AGGCGGTTCGGAAAGAAAGATGTGAAATCCCAGGGCTTAACCTTGGAACTGCATT TTTAACTACCGGGCTAGAGTGTGTCAGAGGGAGGTGGAATTCCGCGTGTAGCAGT GAAATGCGTAGAGATGCGGAGGAACACCGATGGCGAAGGCAGCCTCCTGGGATAA CACTGACGCTCATGCACGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTA GTCCACGCCCTAAACGATGTCAACTAGCTGTTGGGGCCTTCGGGCCTTGGTAGCG CAGCTAACGCGTGAAGTTGACCGCCTGGGGAGTACGGTCGCAAGATTAAAACTCA AAGGAATTGACGGAGGNNNGNNGGAGGGNNGGGGGGNGGGGGAGAGGAGGTTATA GATTGTGGGTATCTTAAGCTTCCGGTTGAGCCGGTAGAGTTATT > Uncultured gamma proteobacterium (99%) ATGGGCGCACCCTGATCCAGCCATGCCGCGTGTGTGAAGAAGGCCTTCGGGTTGT AAAGCTCTTTCTATAGGGAAGAAAAGGCCTGTGTTTATACCGTAAGGCCTTGACG TTACCTGCAAAGCAAGCACCGGCTAACTCCGTGCCCCGCCCCGCTGCAATCTTTC TGGTGGTTATCCGGAATCAAATTACTGGGCGGGTCCGCGGGCGGATCGGGTAGTT AAGGGTGATGTGAAACGGCGGGCTTAACCTGTGAACTGCGTCCGATACTGGTTGA GTTGAGTATGGAAGAGGGAAATGGAATTTCAGGTGTAGCGGTGAAATGCGTTTAT ATCTGAAAGAACATCAGTGGCCAAGGCGGCTTCCTGGTCCTATCGCTGATCTAAG GTGCCAAAGCGTGGGGAGCAAAATTAGATACCATACCCTGGTACTCCGTCCCTAT ACGATACTAAACTAGTTGTTGGAAGGGTCTGCCTTTCAGTGACACGTCTAACGCG TCCACCTCTCCGAGTACGTGAGTACGGCCGCAACGTTAAAACTTTGACGGATTGA CGG > Alcanivorax sp TE‐9 (99%) GGGGGGTTTGGAAATGGGCGCAGCTGATGCCAGCACATGCCGCGTGTGTGAAGAA GGCCTTCGGGTTGTAAAGCTCTTTTGGTAGGGAGGAAGGCTTTGCGGCTAATACC CTGGAGTACTTGACGTTACCTACAGAAGAAGCCCCGGCTAATTTCGTGCCAGCAG CCGCGGTAATACGAAAGGTGCGAGCGTTAATCGGAATTACTGGGCGTAAAGCGCG CGTAGGCGGTGTGTTAAGTCGGATGTGAAAGCCCAGGGCTCAACCTTGGAATTGC ATCCGATACTGGCACGCTAGAGTGCAGTAGAGGGAGGTGGAATTTCCGGTGTAGC GGTGAAATGCGTAGAGATCGGAAGGAACACCAGTGGCGAAGGCGGCCTCCTGGAC TGACACTGACGCTGAGGTGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTG GTAGTCCACGCCGTAAACGATGTCTACTAGCCGTTGGGGTCCTTAGTGACTTTGG TGGCGCAGCTAACGCGATAAGTACACCGCCTGGGGAGTACGGCCGCAAGGTTAAA ACTCAAATGAATTGACGGAANNNCCTNCGACGAAGTAGGGTTGGGATCGAGGGAA GTAGTTGATTGGGGCGTGGTGGGTGGGCTTTTGGTTTTGGGTGCCGCGG >Iron‐reducing bacterium clone HN‐HFO91 (100%) GGGGTTTTTCCAATGGCCGAAAGGCTGATGCAGCAACGCCGCGTGAGTGAAGAAG GCCTTCGGGTTGTAAAGCTCTGTTGCCCGTGAAGAAGTTCCTGCTCAGGAAATGG GGCAGGATTGACGGTAACGGGAGAGGAAGCCCCGGCAAACTACGTGCCAGCAGCC GCGGTAATACGTAGGGGGCGAGCGTTGTCCGGATTTATTGGGCGTAAAGGGCGTG TAGGCGGTCTGGTAAGTCAGGGGTGAAACCCCGGGGCTCAACCCCGGGCGTGCCT CTGATACTGCTGGACTTGAGTGCTCTAGAGGGCAGTGGAATTGCCGGTGTAGCAG TGAAATGCGTAGAGATCGGCAGGAACACCAGTGGCGAAGGCGACTGCCTGGGGAG TAACTGACGCTGAGGCGCGAAAGCTAGGGGAGCGAACAGGATTAGATACCCTGGT AGTCCTAGCCGTAAACGATGAGTGCTATGTGTGAGGGGTATCGACCCCCCTCGTG CAGCAGTTAACGCAATAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGTTGAAAC TCAAAGGAATTGACGGAAAATTTAATTTTTTATTAAATTATTAATTTCGTACACA GCTCGTCGTTGGCGGGGGTTGACGCGGGTT > Uncultured bacterium clone MBFOS‐07 (100%) GCCTGACCGAGCGACGCCGCGTGAGGGAAGAAGGCCTTCGGGTCGTAAACCTCTG TCCTTGGGGAAGAAGGTGACGGTACCCAAGGAGGAAGCCCCGGCTAACTACGTGC CAGCAGCCGCGGTAATACGTAGGGGGCGAGCGTTGTCCGGAATTACTGGGCGTAA AGGGTGCGTAGGCGGCCATTTAAGTCTTGTGTGAAAGGCCCCGGCTTAACCGGGG AGCTGCATGGGAAACTGGATGGCTTGAGTGCTGGAGAGGGTTGCGGAATTCCCAG TGTAGCGGTGAAATGCGTAGATATTGGGAAGAACACCAGTGGCGAAGGCGGCAAC CTGGACAGTAACTGACGCTGAGGCACGACAGCTGGGGGAGCGAACAGGATTAGAT ACCCTGGTAGTCCCAGCCGTAAACGATGAGTGCTAGGTGTTGCGGGTATCGACCC CTGCAGTGCCGCAGTTAACGCAATAAGCACTCCGCCTGGGGAGTACGGCCGCAAG GCTGAAACTCAAAGGAATTGACGGA > Marine bacterium SCRIPPS_413 (98%) GGGGAAAGGAATGGGGGCAAACCTGATCCAGCCATGCCGCGTGCAGGAAGACTGC CCTATGGGTTGTAAACTGCTTTTATACGGGAAGAAACACCCCTACGTGTAGGGGC TTGACGGTACCGTAAGAATAAGGATCGGCTAACTCCGTGCCAGCAGCCGCGGTAA TACGGAGGATCCAAGCGTTATCCGGAATCATTGGGTTTAAAGGGTCCGTAGGCGG ATAATTAAGTCAGAGGTGAAATCCTGCAGCTCAACTGTAGAACTGCCTTTGATAC TGGTTGTCTTGAGTTATTATGAAGTAGTTAGAATATGTAGTGTAGCGGTGAAATG CATAGATATTACATAGAATACCGATTGCGAAGGCAGATTACTAACAATATACTGA CACTGATGGACGAAAGCGTGGGGAGCGAACAGGATTAGATACCCTGGTAGTCCAC GCCGTAAACGATGGTCACTAGCTGTTCGGATTTCGGTCTGAGTGGCTAAGCGAAA GTGATAAGTGACCCACCTGGGGAGTACGTTCGCAAGAATGAAACTCAAATGAATT GACGGACGGTCTCTTTTTTGGGTCGCTGGTGTTTTTAGGTCTACTCTTAGCGTGG GTACGAAAAGAAGCAAGGAAGAG > Uncultured bacterium clone BR130 (99%) GCGCAGCCTGATCCAGCCATGCCGCGTGCAGTGAAGAAGGCCTTAGGGTTGGAAA ACTGTTTTTATACGGAAGAAAAAGAGAGTACCTGTACTCCAATGACCGGACCATA CCAAGCCAGCCCGGCTAACTCCACGCCAGGAGCCGCGGTTATACGGAAGGTCCGG GCGTTATCCGGAATCATTGGGTTTAAAGGGTCCGGGGTGAAATCATTGGGTTCAG GGTGAAAGGTGCCGGCTCAACTGTAGCACTGCCCTTGATACTGATTGTCTTGAGT TATGGTGAAGTGGCCGGAATATGTAGTGTTTCGGTGAAATGCATATATATTAGGC GAACACCGATTGCGAATGGACGTCACTAACCATTGACTGACGCTGAAAAAAGAAA GCGTGGCGAGGGAACTGCATTAGATACCCCAGTAGTCCACGCTGTAAACGATGGA TACTATCTGTCGGGCTTTCTGGCTCGGCGGCCAAGCGAAAGTGATAAGTATCCCA CCTGGTGAGTACGTTCGCAAGAATGAAACTCAAAGGAATTGACGGA > Uncultured bacterium clone VH‐FL6‐50 (100%) AACCCTGATCCAGCTATGCCGCGTGTGTGAAGAAGGCCTTAGGGTTGTAAAGCAC TTTAAGTTGTGAGGAAAGGTTAGTAGTTAATACCTGCTAGCTGTGACGTTAGCAA CAGAATAAGCACCGGTCTAACTCCGTGCCAGCAGCCGCGGTAATACGGAGGGTGC AAGCGTTAATCGGAATTACTGGGCGTAAAGCGCGCGTAGGTGGTTTGTTAAGTTA GATGTGAAAGCCCCGGGCTTAACCTGGGAACTGCATCTGATACTGGCAGGCTAGA GTACGAAAGAGGGGAGTGGAATTTCCAGTGTAGCAGTGAAATGCGTAGAGATTGG AAGGAACACCAGTGGCGAAGGCGACTCCCTGGTTTGATACTGACACTGAGGTGCG AAAGCGTGGGTAGCGAACGGGATTAGATACCCCGGTAGTCCACGCCGTAAACGAT GTCTACTAGCCGTTGGGAATCTTGAATTCTTAGTGGCGCAGTTAACGCGATAAGT AGACCGCCTGGGGAGTACGGCCGCAAGGCTAAAACTCAAATGAATTGACGGG > uncultured Bordetella sp(100%) CCGCGTGTGCGATGAAGGCCTTCGGGTTGTAAAGCACTTTTGGCAGGGAAGAAAC GGCCCTGGTTAATACCTGGGGCAACTGACGGTACCTGCAGAATAAGCACCGGCTA ACTACGTGCCAGCAGCCGCGGTAATACGTAGGGTGCAAGCGTTAATCGGAATTAC TGGGCGTAAAGCGTGCGCAGGCGGTTCGGAAAGAAAGATGTGAAATCCCAGGGCT TAACCTTGGAACTGCATTTTTAACTACCGGGCTAGAGTGTGTCAGAGGGAGGTGG AATTCCGCGTGTAGCAGTGAAATGCGTAGAGATGCGGAGGAACACCGATGGCGAA GGCAGCCTCCTGGGATAACACTGACGCTCATGCACGAAAGCGTGGGGAGCAAACA GGATTAGATACCCTGGTAGTCCACGCCCTAAACGATGTCAACTAGCTGTTGGGGC CTTCGGGCCTTGGTAGCGCAGCTAACGCGTGAAGTTGACCGCCTGGGGAGTACGG TCGCAAGATTAAAACTCAAAGGAATTGACGG > Bacillus sp. L41 (99%) GGGGGTTTGCATGGAGAAAGTCTTGACGGAGCAACGCCGCGTGAACGATGAAGGC CTTCGGGTCGTAAAGTTCTGTTGTTAGGGAAGAACAAGTACCAGAGTAACTGCTG GTACCTTGACGGTACCTAACCAGAAAGCCACGGCTAACTACGTGCCAGCAGCCGC GGTAATACGTAGGTGGCAAGCGTTGTCCGGAATTATTGGGCGTAAAGCGCGCGCA GGCGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTG GAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCACGTGTAGCGGTG AAATGCGTAGAGATGTGGAGGAACACCAGTGGCGAAGGCGACTCTCTGGTCTGTA ACTGACGCTGAGGCGCGAAAGCGTGGGGAGCGAACAGGATTAGATACCCTGGTAG TCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCT GCAGCAAACGCATTAAGCACTCCGCCTGGGGAGTACGGTCGCAAGACTGAAACTC AAATGAATTGACGG > uncultured Firmicutes bacterium BC_COM467 (100%) GCCTGACCGAGCGACGCCGCGTGAGGGAAGAAGGCCTTCGGGTCGTAAACCTCTG TCCTTGGGGAAGAAGGTGACGGTACCCAAGGAGGAAGCCCCGGCTAACTACGTGC CAGCAGCCGCGGTAATACGTAGGGGGCGAGCGTTGTCCGGAATTACTGGGCGTAA AGGGTGCGTAGGCGGCCATTTAAGTCTTGTGTGAAAGGCCCCGGCTTAACCGGGG AGCTGCATGGGAAACTGGATGGCTTGAGTGCTGGAGAGGGTTGCGGAATTCCCAG TGTAGCGGTGAAATGCGTAGATATTGGGAAGAACACCAGTGGCGAAGGCGGCAAC CTGGACAGTAACTGACGCTGAGGCACGACAGCTGGGGGAGCGAACAGGATTAGAT ACCCTGGTAGTCCCAGCCGTAAACGATGAGTGCTAGGTGTTGCGGGTATCGACCC CTGCAGTGCCGCAGTTAACGCAATAAGCACTCCGCCTGGGGAGTACGGCCGCAAG GCTGAAACTCAAAGGAATTGACGGA > Uncultured bacterium clone W1‐16 (92%) GCGCAGCTGATGCAGCAATGCCGCGTGCGGGATGAAGGCCTTCGGGTTGTAAACC CCTTTCAGCAGGGACGAAAATGACGGTACCTGCAGAAGAAGGTACGGCCAACTAA ATGCCATCAGCCGCGGTAACGCATAGGGGGGGACGTAGTGTGGTTTTGAATTATT GGGAGAACTGCGCGGGGGTGCGGCCCGTCAAGTGTGAAGACTCAGGGCTCAGCCC AACCACGCCACTGCATTCTGTACTGGCGGGCTTGAGGTAGGTGAGGGAGGATTTC ATGGTGTGTGTGTCAATGGAATGCATATCTATAGGAAGGAACACCGCTGAGGCAG GCCTCTGGCCCGAGCTCGCCTCTGACTAACAAACGAAAGCGTGGGTAACAAAATT AAATACCTTGGTTGTCCATCCCTGACACATACGATAGTAGGTGTGGGTGGGGACC AATTAACTGCCTCCCGGTCGAAACTAATTCATTGCCCCCCCCGGCGGGGGACGAC GGCCGCCTAACAAAAAAACAAATTGATTGACGGA > Uncultured bacterium clone Uday0‐58 (100%) AGCCTGATGCAGCAACGCCGCGTGCGGGATGAAGGCCCTAGGGTTGTAAACCGCT TTCAGCAGGGAAGAAACTGACGGTACCTGCAGAAGAAGGTGCGGCCAACTACGTG CCAGCAGCCGCGGTGACACGTAGGCACCAAGCGTTGTCCGGATTTATTGGGCGTA AAGAGCTCGTAGGCGGTTCAGTAAGTCGGGTGTGAAAACTCTGGGCTCAACCCAG AGACGCCACCCGATACTGCTGTGACTTGAGTACGGTAGGGGAGTGGGGAATTCCT GGTGTAGCGGTGAAATGCGCATATATCAGGAGGAACACCAGTGGCGAAAGCGCCA CTCTGGGCCGTAACTGACGCTGAGGACCGAAAGCGTGGGTAGCAAACAGGATTAG ATACCCTGGTAGTCCACGCCGTAAACATTCGGCACTAGGTGTGGGTCTCAACCAA CGAGATCCGCGCCGTCGCTAACGCATTAAGTGCCCCGCCTGGGGAGTACGGTCGC AAGACTAAAACTCAAATGAATTGACGG > Pseudomonas sp. clone HY2 (100%) CCTGATCCAGCTATGCCGCGTGTGTGAAGAAGGCCTTAGGGTTGTAAAGCACTTT AAGTTGGGAGGAAAGGTTAGTAGTTAATACCTGCTAGCTGTGACGTTAGCAACAG AATAAGCACCGGCTAACTCCGTGCCAGCAGCCGCGGTAATACGGAGGGTGCAAGC GTTAATCGGAATTACTGGGCGTAAAGCGCGCGTAGGTGGTTTGTTAAGTTAGATG TGAAAGCCCCGGGCTTAACCTGGGAACTGCATCTGATACTGGTGGGCTAGAGTGC GAAAGAGGGGAGTGGAATTTCCAGTGTAGCAGTGAAATGCGTAGAGATTGGAAGG AACACCAGTGGCGAAGGCGACTCCCTGGTTTGATACTGACACTGAGGTGCGAAAG CGTGGGTAGCGAACGGGATTAGATACCCCGGTAGTCCACGCCGTAAACGATGTCT ACTAGCCGTTGGGAATCTTGAATTCTTAGTGGCGCAGTTAACGCGATAAGTAGAC CGCCTGGGGAGTACGGCCGCAAGGCTAAAACTCAAATGAATTGACGG > iron‐reducing bacterium clone HN17 (98%) GGGTTAGCAATGGGCGAAAGCCTGACGCAGCCATGCCGCGTGGGTGATGAAGGCC TTAGGGTTGTAAAACCCTTTCACCGGGGACGATAATGACGGTACCCGGAGAAGAA GCCCCGGCTAACTTCGTGCCAGCAGCCGCGGTAATACGAAGGGGGCTAGCGTTGT TCGGAATTACTGGGCGTAAAGGGCGCGTAGGCGGACAGTTAAGTTGGGGGTGAAA GCCCGGGGCTCAACCTCGGAATTGCCTTCAATACTGGCTGTCTTGAGTTCGGGAG AGGTGAGTGGAACTCCGAGTGTAAAGGTGAAATTCGTAGATTTTCGGAAGAACAC CAGTGGCGAAGGCGACTCACTGGCCCGTTACTGACGCTGAGGCGCGAAAGCGTGG GGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAG TTGTCGGCGTGCATGCATGTCAGTGACGCAGCTAACGCATTAAGCACTCCGCCTG GGGAGTACGGTCGCAAGATTAAAACTCAAAGGAATTGACGGACGGATAAGA > Uncultured bacterium clone N704B_48 (99%) GGGGTAAGAAATGGGGGGCAACCCTGATCCAGCCACGCCGCGTGAGTGATGAAGG CCTTAGGGTTGTAAAGCTCTTTCGGCGGGGACGATAATGACGGTACCCGGAGAAG AAGCCCCGGCTAACTTCGTGCCAGCAGCCGCGGTAATACGAAGGGGGCTAGCGTT GTTCGGAATCACTGGGCGTAAAGCGCACGTAGGCGGACCGTTAAGTCGGAGGTGA AAGCCTGGGGCTCAACCCCAGAACTGCCTTCGATACTGGCGGTCTTGAGTGTGGT AGAGGTTGGTGGAACTCCGAGTGTAGAGGTGAAATTCGTAGATATTCGGAAGAAC ACCAGTGGCGAAGGCGGCCAACTGGACCATTACTGACGCTGATGTGCGAAAGCGT GGGTAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGAGCT AGCCGTTGGCCTGCATGCATGTCAGTGACGCAGCTAACGCGTTAAGCTCTCCGCC TGGGGAGTACGGTCGCAAGATTAAAACTCAAAGGAATTGACGGAGG > Alteromonas sp. MOLA 3 (100%) ACCTGATGCAGCCATGCCGCGTGTGTGAAGAAGGCCTTCGGGTTGTAAAGCACTT TCAGTTGTGAGGAAAAGTTAGTAGTTAATACCTGCTAGCCGTGACGTTAACAACA GAAGAAGCACCGGCTAACTCCGTGCCAGCAGCCGCGGTAATACGGAGGGTGCGAG CGTTAATCGGAATTACTGGGCGTAAAGCGCACGCAGGCGGTTTGTTAAGCTAGAT GTGAAAGCCCCGGGCTCAACCTGGGATGGTCATTTAGAACTGGCAGACTAGAGTC TTGGAGAGGGGAGTGGAATTCCAGGTGTAGCGGTGAAATGCGTAGATATCTGGAG GAACATCAGTGGCGAAGGCGACTCCCTGGCCAAAGACTGACGCTCATGTGCGAAA GTGTGGGTAGCGAACAGGATTAGATACCCTGGTAGTCCACACCGTAAACGCTGTC TACTAGCTGTGTGTGAATTTAATTCGTGCGTAGCGAAGCTAACGCGCTAAGTAGA CCGCCTGGGGAGTACGGCCGCAAGGTTAAAACTCAAATGAATTGACGGA