KLONING GEN PENYANDI SUKROSA SINTASE DARI
advertisement

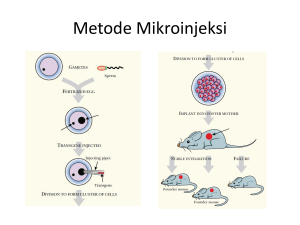
KLONING GEN PENYANDI SUKROSA SINTASE DARI TANAMAN SENGON (Paraserianthes falcataria) HARTATI G451030041 SEKOLAH PASCA SARJANA INSTITUT PERTANIAN BOGOR BOGOR 2007 Kloning Gen Penyandi Sukrosa Sintase Dari Tanaman Sengon (Paraserianthes falcataria) Abstrak Hartati. Kloning Gen Penyandi Sukrosa Sintase Dari Tanaman Sengon (Paraserianthes falcataria). Dibimbing oleh Dr. Djarot Sasongko Hamiseno dan Dr. Enny Sudarmonowati, APU. Salah satu aspek yang dipelajari dan diteliti untuk meningkatkan kualitas kayu sengon (Paraserianthes falcataria) adalah peningkatan deposisi selulosa. Sukrosa sintase merupakan salah satu enzim kunci yang berperan dalam biosintesis selulosa. Sukrosa sintase mengkatalisis reaksi konversi sukrosa + UDP UDP-glukosa + fruktosa. Enzim ini terlibat dalam biosintesis selulosa dengan cara menghubungkan UDP-Glukosa ke mesin sintesis selulosa (selulosa sintase). Penelitian yang terkait dengan gen penyandi sukrosa sintase yang terlibat pada biosntesis selulosa pada sengon masih terbatas. Tujuan dari penelitian ini adalah untuk mengisolasi dan mengkloning gen penyandi sukrosa sintase yang bersumber dari sengon dengan teknik RT-PCR (sintesis cDNA) menggunakan primer yang spesifik. Kandidat gen yang berhasil diisolasi ditransformasikan ke bakteri Escherichia coli menggunakan vektor plasmid pGemT-Easy. Dari hasil sekuensing fragmen cDNA sukrosa sintase diperoleh 341 pb nukleotida yang menyandi 112 asam amino. Studi homologi yang dilakukan pada sekuens basa nukleotida dan protein penyandi sukrosa sintase sengon terhadap basa nukleotida dan protein penyandi sukrosa sintase dari tanaman lain menunjukkan bahwa sekuens gen penyandi sukrosa sintase pada sengon memiliki kesamaan dengan Glycine max, Citrus unshiu, Vigna radiata dan Arabidopsis thaliana dengan tingkat kesamaan masing-masing 79%, 78%, 75% dan 73%. Key words : vektor Paraserianthes falcataria, selulosa, sukrosa sintase, RT-PCR, pGemT-Easy, kloning gen. 2 KLONING GEN PENYANDI SUKROSA SINTASE DARI TANAMAN SENGON (Paraserianthes falcataria) Hartati G451030041 Tesis Sebagai salah satu syarat untuk memperoleh gelar Master Sains pada Program Studi Biokimia SEKOLAH PASCA SARJANA INSTITUT PERTANIAN BOGOR BOGOR 2007 3 Judul Penelitian : Kloning Gen Penyandi Sukrosa Sintase Dari Tanaman Sengon (Paraserianthes falcataria) Nama : Hartati NRP : G451030041 Disetujui Komisi Pembimbing Dr. Djarot Sasongko Hamiseno Dr. Enny Sudarmonowati, APU. Ketua Anggota Diketahui oleh Ketua Program Studi Biokimia Prof. Dr. Maria Bintang Dekan Sekolah Pascasarjana Prof. Dr. Ir. Khairil Anwar Notodiputro, MS Tanggal lulus : 4 RIWAYAT HIDUP Penulis dilahirkan di Porsea, Kabupaten Tapanuli di Propinsi Sumatera Utara pada tanggal 10 Juni 1972 dari bapak Saman (Alm) dan ibu Porman Marpaung. Penulis merupakan anak ketiga dari lima bersaudara. Penulis menyelesaikan pendidikan sarjana Program Studi Kimia, Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Sumatera Utara dan lulus pada tahun 2001. Pada Tahun 2003, penulis diterima di Program Studi Biokimia Sekolah Pascasarjana Institut Pertanian Bogor (IPB). Penulis bekerja sebagai peneliti di Pusat Penelitian Bioteknologi Lembaga Ilmu Pengetahuan Indonesia (LIPI) di Cibinong, Kabupaten Bogor, Jawa Barat. 5 PRAKATA Puji syukur penulis panjatkan kepada Allah SWT atas segala rahmat dan karunia-Nya sehingga penulis dapat menyusun dan menyelesaikan tesis ini. Dan tak lupa, shalawat beriring salam kepada Rasulullah Muhammad SAW atas suri tauladan yang baik dalam hidup dan mencari ilmu. Pada kesempatan ini penulis ingin menyampaikan ucapan terima kasih yang sebesar-besarnya kepada : 1. Dr. Djarot Sasongko Hamiseno dan Dr. Enny Sudarmonowati, APU selaku pembimbing yang telah membimbing dan mengarahkan penulis hingga penelitian dan tesis ini bisa diselesaikan. 2. Prof. Dr. Maria Bintang sebagai Ketua Program Studi Biokimia. 3. Bapak dan Mamak yang telah memberikan dorongan dan semangat kepada penulis selama menempuh kuliah dan menyelesaikan tesis. 4. Kak Sri Sugiarti sebagai penyemangat, penyokong dan pemberi dana bagi penulis selama menempuh kuliah dan menyelesaikan tesis. 5. Kak Ida dan keluarga, Adik-Adikku Zulham dan Leo serta Sanak Saudara yang telah memberikan dukungan moril bagi penulis selama menempuh kuliah dan menyelesaikan tesis. 6. Seluruh teman-teman peneliti di laboratorium Biologi Molekuler Puslit Bioteknologi LIPI. Pada akhirnya, penulis berharap agar tulisan ini bermanfaat bagi pihakpihak yang memerlukan. Bogor, 1 Desember 2007 Hartati 6 DAFTAR ISI Halaman DAFTAR ISI................................................................................................ i DAFTAR TABEL....................................................................................... iii DAFTAR GAMBAR................................................................................... iv DAFTAR LAMPIRAN.............................................................................. v I. PENDAHULUAN 1.1. Latar Belakang...................................................................................1 1.2. Tujuan Penelitian...............................................................................2 II. TINJAUAN PUSTAKA 2.1 Tanaman Sengon ..............................................................................4 2.2. Kimia Kayu Sengon...................................................................... 6 2.3. Biosintesis Selulosa...................................................................... 7 2.4. Sukrosa Sintase ............................................................................. 9 2.5. Kloning Gen ................................................................................. 10 2.5.1. Sintesis cDNA..................................................................... 11 2.5.2. Vektor Kloning................................................................. 12 2.5.3. Plasmid pGemT-Easy dan Strategi Ligasi........................ 13 2.5.4. Transformasi Bakteri......................................................... 14 2.5.5. Seleksi dan Verifikasi Transforman.................................... 15 III. BAHAN DAN METODE 3.1. Tempat dan Waktu............................................................................... 16 3.2. Bahan Penelitian.................................................................................. 16 3.3. Metode Penelitian............................................................................. 16 3.3.1. Isolasi RNA Total Sengon ................................................. 16 3.3.2. Sintesis cDNA Gen Sukrosa Sintase Melalui RT PCR.... 17 3.3.3. Elektroforesis Produk PCR................................................. 17 3.3.4. Isolasi dan Pemurnian cDNA produk PCR................... 17 7 3.3.5. Kloning cDNA ke dalam Vektor pGemT- Easy ..…….. 18 3.3.6. Transformasi Gen ke E. coli DH5α…………… ……… 18 3.3.7. Isolasi DNA Plasmid...................................................... 19 3.3.8. Sekuensing cDNA.......................................................... 20 3.3.9. Analisis Homologi……………....…………………….. 20 IV. HASIL DAN PEMBAHASAN 4.1. Isolasi RNA Total............................................................................ 22 4.2. Sintesis cDNA................................................................................... 23 4.3. Kloning Fragmen............................................................................... 24 4.4. Verifikasi Sisipan dan Analisis Situs Pemotongan Enzim Restriksi.... 25 4.5. Pengurutan Fragmen Sukrosa Sintase.................................................. 26 4.6. Analisis Homologi Sukrosa Sintase….................................................. 27 4.7. Analisis Tingkat Kekerabatan………...…………………………...…. 29 4.8. Pembahasan Umum…………………………………………………… 30 V. SIMPULAN DAN SARAN 5.1. Simpulan....................................................................................... 32 5.2. Saran............................................................................................. 32 Daftar Pustaka.............................................................................................. 33 Lampiran..................................................................................................... 35 8 DAFTAR TABEL Halaman 1. Komposisi Kimia Serbuk Kayu Sengon ..........................................................6 2. Penyejajaran nilai kesamaan basa nukleotida sukrosa sintase dari beberapa tumbuhan........................ ..................................................... 28 3. Penyejajaran deduksi protein sukrosa sintase dari beberapa tumbuhan....... 29 9 DAFTAR GAMBAR Halaman 1. Pohon sengon .......................................................................................... 4 2. Biosintesis selulosa.................................................................................. 7 3. Model biosintesis selulosa pada Populus sp............................................ 9 4. Peta vektor pGemT - Easy....................................................................... 13 5. Integritas pita RNA 28S dan 18S dari RNA total ………………....... 23 6. Síntesis cDNA………………….…………………………………...... 23 7. Pita hasil PCR cDNA............................................................................. 24 8. Koloni E. coli hasil transformasi berupa koloni putih .......................... 25 9. Hasil restriksi fragmen sukrosa sintase dengan EcoR1 …………….25 10. Peta restriksi fragmen sukrosa sintase berdasarkan NEB Cutter…….. 26 11. Urutan basa nukleotida fragmen sukrosa sintase dan deduksi asam aminonya……………………………………………………….27 12. Nilai kekerabatan sukrosa sintase berbagai tumbuhan berdasarkan deduksi asam amino…………………… ……………………........... 30 10 DAFTAR LAMPIRAN Halaman 1. Alur Penelitian Isolasi dan Kloning Gen Penyandi Sukrosa Sintase Pada Tanaman Sengon (Paraserianthes falcataria)……….………………….. 35 2. Deduksi asam amino yang menyandi sukrosa sintase sengon dibandingkan dengan asam amino sucrosa sintase dari berbagai tumbuhan.................…. 36 11 BAB I PENDAHULUAN 1.1. Latar Belakang Sengon merupakan salah satu tanaman asli Indonesia yang dibudidayakan oleh Hutan Tanaman Industri (HTI) sebagai sumber bahan baku, terutama untuk industri pulp dan kertas, konstruksi bangunan, peti kemas, dan lain-lain. Budidaya sengon dilatarbelakangi oleh peningkatan kapasitas produksi industri berbahan dasar kayu yang tidak disertai oleh peningkatan dan jaminan ketersediaan pasokan bahan baku kayu, sementara hutan alam secara kuantitas tidak dapat lagi memenuhi kebutuhan bahan baku kayu yang memadai. Untuk mengatasi kesenjangan tersebut, HTI membudidayakan jenis tanaman kayu cepat tumbuh (fast growing tree), seperti sengon, Acacia mangium, Eucalyptus sp, dan lain-lain (Santoso, 1992). Kayu sengon sudah dapat dipanen pada umur 5 tahun. Jika dibandingkan dengan jati yang baru dapat dipanen pada umur lebih dari 10 tahun, umur panen kayu sengon jauh lebih cepat. Sifat unggul tersebut menjadikan sengon sangat tepat dan cepat digunakan sebagai sumber untuk memenuhi kebutuhan bahan baku kayu yang semakin menipis (Hidayat, 2002) Selain peningkatan kuantitas kayu melalui budidaya kayu kategori cepat tumbuh, perbaikan kualitas pada kayu-kayu yang secara komersial sangat menguntungkan juga terus dilakukan. Penelitian yang terkait dengan bioteknologi kayu saat ini banyak menggunakan teknologi rekayasa genetik untuk meningkatkan kualitas dan perbaikan sifat kayu seperti yang diinginkan (Joshi et.al., 2004). Nilai kualitas kayu sangat tergantung pada pemanfaatan kayu untuk menghasilkan suatu produk. Perbaikan sifat fisik seperti densitas dan sudut mikrofibril sangat mempengaruhi peningkatan kekuatan kayu, sedang perbaikan sifat kimia seperti deposisi jumlah selulosa dan penurunan kadar lignin sangat penting dan menentukan bagi industri, terutama pada industri pulp dan kertas (Bowyer et.al., 2000). Hasil penelitian terhadap sifat fisik dan dimensi serat serta analisis komponen kimia penyusunan kayu sengon termasuk selulosa menunjukkan bahwa kayu sengon merupakan salah satu bahan baku yang baik dan dapat dimanfaatkan sebagai alternatif kayu substitusi pada industri pulp dan kertas (Kartiwa, 1998). 12 Serat kayu sengon merupakan jenis kayu serat pendek yaitu berkisar antara 0,6-1 mm. Kayu serat pendek banyak dimanfaatkan sebagai bahan baku industri pulp terutama industri pulp yang menghasilkan kertas gelombang (Siagian et. al.,2003). Sifat kimia kayu dikendalikan oleh berbagai gen yang bekerja secara kompleks. Pengetahuan tentang gen-gen yang berperan untuk pengendalikan sifat kayu sangat penting untuk meningkatkan sifat dan kualitas kayu dimasa yang akan datang (Neale, 2002). Peningkatan deposisi selulosa merupakan salah satu aspek yang diinginkan untuk perbaikan kualitas kayu, sehingga banyak penelitian yang dilakukan untuk mencari gen yang mengendalikan proses biosintesis selulosa. Biosintesis selulosa pada tumbuhan dikendalikan oleh berbagai gen. Sebagai contoh, biosintesis selulosa pada poplar dikendalikan oleh beberapa gen, seperti selulosa sintase, sukrosa sintase dan korrigan selulase (Joshi et.al., 2004). Sukrosa sintase merupakan enzim kunci pada biosintesis selulosa yang mengkatalisis reaksi konversi sukrosa dan UDP menjadi UDP-glukosa dan fruktosa. Enzim ini terlibat pada biosintesis selulosa dengan cara menghubungkan dan menyediakan UDP-glukosa ke selulosa sintase pada sintesis dinding sel sekunder (Amor et. al., 1995). Sukrosa sintase juga diduga menghubungkan UDP-glukosa dari sukrosa ke berbagai β glukan sintase pada biosintesis selulosa (Konishi et. al., 2004). Informasi mengenai gen yang terlibat pada biosintesis selulosa pada sengon masih belum banyak diteliti. Pada penelitian ini akan dilakukan isolasi dan kloning gen penyandi sukrosa sintase dari tanaman sengon melalui pembuatan pustaka cDNA dengan teknik ”Reverse Transcriptase Polymerase Chain Reaction” (RT PCR). Beberapa penelitian sebelumnya telah berhasil mengidentifikasi dan mengisolasi gen penyandi sukrose sintase pada berbagai tanaman, diantaranya adalah sukrosa sintase pada Vigna radiata (Konishi et. al., 2004), kapas (Amor et.al., 1995), A. thaliana, Populus sp, Eucaliptus sp, dan lainlain. 1.2. Tujuan Penelitian Penelitian ini bertujuan untuk mengisolasi dan mengklon gen penyandi sukrosa sintase dari tanaman sengon. 13 1.3. Manfaat Penelitian Penelitian yang dilakukan merupakan studi awal untuk memperoleh gen yang terlibat dalam pengendalian biosintesis selulosa yang bermanfaat untuk peningkatan kualitas kayu dan perbaikan sifat sengon dimasa yang akan datang. 1.4. Hipotesis Penelitian Teknik RT PCR dan kloning gen dapat digunakan untuk memperoleh gen penyandi sukrosa sintase. 14 BAB II TINJAUAN PUSTAKA 2.1. Tanaman Sengon Sengon yang dalam bahasa latin dikenal sebagai Paraserianthes falcataria, termasuk famili Leguminoseae sub famili Mimosaceae atau keluarga petai-petaian (Gambar 1). Di Indonesia, sengon memiliki beberapa nama daerah seperti jeunjing laut (Sunda), jeunjing, kalbi, sengon landi, sengon laut, atau sengon sabrang (Jawa), seja (Ambon), sikat (Banda), tawa (Ternate), dan gosui (Tidore). Sengon merupakan tanaman asli Indonesia dengan sebaran alami di daerah Papua, Maluku, kep. Solomon, Bismark. Sedangkan untuk hutan tanaman banyak dijumpai di Sulawesi, Jawa, dan Kalimantan (Hidayat, 2002) Gambar 1. Pohon sengon 15 Bagian terpenting yang mempunyai nilai ekonomi pada tanaman sengon adalah kayunya. Ketinggian pohon sengon dapat mencapai sekitar 30–45 meter dengan diameter batang 70–80 cm. Bentuk batangnya bulat dan tidak berbanir. Kulit luarnya berwarna putih atau kelabu, tidak beralur dan tidak mengelupas. Berat jenis kayu rata-rata 0,33. Sengon memiliki akar tunggang yang cukup kuat menembus kedalam tanah, akar rambutnya tidak terlalu besar, tidak rimbun dan tidak menonjol kepermukaan tanah. Akar rambutnya berfungsi untuk menyimpan zat nitrogen, oleh karena itu tanah disekitar pohon sengon menjadi subur. Dengan sifat-sifat kelebihan yang dimiliki sengon, maka banyak pohon sengon ditanam di tepi kawasan yang mudah terkena erosi dan menjadi salah satu kebijakan pemerintah melalui Departemen Kehutanan dan Perkebunan untuk menggalakkan ‘Sengonisasi’ di sekitar daerah aliran sungai (DAS) di Jawa, Bali dan Sumatra (Santoso, 1992) Bunga tanaman sengon tersusun dalam bentuk malai berukuran sekitar 0,5-1 cm, berwarna putih kekuning-kuningan dan sedikit berbulu. Setiap kuntum bunga mekar terdiri dari bunga jantan dan bunga betina, dengan cara penyerbukan yang dibantu oleh angin atau serangga. Buah sengon berbentuk polong, bulat pipih, tipis, dan panjangnya sekitar 6–12 cm. Setiap polong buah berisi 15–30 biji. Bentuk biji mirip perisai kecil dan jika sudah tua biji akan berwarna coklat kehitaman,agak keras, dan berlilin (Star et.al., 2003) Sengon merupakan pohon multifungsi. Sebagai contoh, daun sengon sebagaimana famili Mimosaceae lainnya merupakan pakan ternak yang sangat baik dan mengandung protein tinggi. Sistem perakaran sengon banyak mengandung nodul akar sebagai hasil simbiosis dengan bakteri Rhizobium. Keberadaan nodul akar dapat meningkatkan porositas tanah dan penyediaan unsur nitrogen dalam tanah, sehingga tanah disekitarnya menjadi lebih subur. Batang kayu banyak diusahakan untuk berbagai keperluan dalam bentuk kayu olahan berupa papan papan dengan ukuran tertentu sebagai bahan baku pembuat peti, papan penyekat, pengecoran semen dalam kontruksi, industri korek api, pensil, papan partikel dan bahan baku industri pulp kertas (Hidayat, 2003) 16 2.2. Kimia Kayu Sengon Sengon termasuk dalam kategori pohon yang cepat tumbuh (fast growing tree) dan merupakan kayu kelas awet IV-V. Hasil analisis komposisi kimia serbuk gergajian kayu sengon dapat dilihat pada Tabel 1. Kandungan selulosa tumbuhan ini termasuk tinggi dan kadar ligninnya termasuk dalam kategori sedang yaitu berkisar antara 18-33% (Pari, 1996). Semua jenis kayu yang memiliki serat selulosa dapat digunakan sebagai bahan baku pulp kertas atau rayon. Namun dalam proses pelaksanaannya perlu dilakukan pembatasan terhadap faktor teknis dan ekonomis. Faktor teknis seperti jenis dan sifat kayu merupakan hal yang paling penting dalam pemilihan kayu untuk memperoleh kualitas pulp yang baik, biaya produksi lebih ekonomis dan kertas yang dihasilkan memiliki kekuatan dan nilai jual yang tinggi (Kartiwa, 2006). Tabel 1. Komposisi kimia serbuk kayu sengon (Pari, 1996) Komponen Kandungan (%) Hemiselulosa 70,52 Selulosa 40,99 Lignin 27,88 Pentosan 16,89 Abu 1,38 Air 5,64 Secara umum faktor yang perlu diperhatikan dalam pemilihan kayu untuk pulp diantaranya adalah kulit kayu tipis sampai sedang, batang lurus tidak terpuntir, tidak banyak mata kayu, tidak mengeluarkan banyak getah dan relatif tahan dalam proses penyimpanan. Kayu yang sudah baku dan terseleksi secara komersial untuk pulp sudah banyak digunakan secara rutin dan dibudidayakan. Saat ini ada beberapa kayu yang digunakan sebagai bahan baku alternatif untuk 17 pembuatan pulp, walaupun belum diketahui jenis dan manfaatnya. Akhir-akhir ini dikenal kayu dengan sebutan Mixed Tropical Hardwood (MTH) yang terdiri dari dua jenis atau lebih kayu yang memiliki variasi berbeda. Kelompok ini sering digunakan sebagai bahan baku pulp untuk memenuhi kebutuhan dan menjaga kelangsungan produksi pulp dan memenuhi kebutuhan kertas dunia saat ini. Hasil analisis komponen kimia kayu sengon memungkinkan untuk memanfaatkan sengon sebagai alternatif kayu substitusi pada industri pulp dan kertas (Kartiwa, 2006). 2.3. Biosintesis Selulosa Selulosa merupakan penyusun utama dinding sel kayu berupa polimer alami yang terdiri dari residu β-D-glukosa yang dihubungkan oleh ikatan glikosida seperti pada Gambar 2. Walaupun biosintesis selulosa merupakan salah satu proses biologi yang membedakan tumbuhan dan hewan, mekanisme biosintesis selulosa masih belum sepenuhnya diketahui (Read, 2002). Gambar 2. Biosintesis selulosa (Bohinsky, 1987) Selulosa dibentuk oleh kurang lebih 25000 molekul glukosa membentuk satu rantai polimer. Rantai polimer selulosa akan berikatan dengan rantai selulosa yang lain melalui ikatan hidrogen membentuk mikrofibril. Satu mikrofibril 18 dibentuk dari 30-100 rantai selulosa dan gabungan mikrofibril tersebut akan membentuk struktur dinding sel kayu yang kuat (Lea, 1994) Biosintesis selulosa berlangsung di membran plasma dimana uridin difosfat atau UDP-glukosa bertindak sebagai donor residu glukosa. Proses biosintesis selulosa dikatalisis oleh enzim selulosa sintase. Enzim selulosa sintase memiliki dua situs katalitik yang mentransfer dua unit glukosil dari UDP-glukosa ke rantai selulosa yang sedang mengalami perpanjangan sehingga enzim ini diklasifikasikan sebagai golongan enzim glukosiltransferase (Bohinski, 1987). Sukrosa bertindak sebagai pensuplai glukosa yang terikat ke UDP. Selulosa sintase mengkatalisis pembentukan satu rantai glukan yang akan bergabung dengan rantai lain membentuk mikrofibril. Selain selulosa sintase, enzim yang terlibat dalam sintesis selulosa adalah 1,4 β-glukanase, terutama untuk pembentukan mikrofibril (Lea, 1994). Penelitian biosintesis selulosa yang dilakukan pada pohon poplar (Populus sp) dapat dijadikan sebagai model biosintesis selulosa pada tanaman kehutanan yang lain. Informasi genetik poplar, terutama untuk biosintesis selulosa pada pohon berkayu relatif lebih lengkap. Poplar merupakan salah satu jenis pohon yang banyak terdapat di Amerika dan Eropa. Karena sifatnya yang relatif tahan terhadap serangan hama, membuat tanaman ini banyak dikembangkan sebagai sumber bahan baku kayu untuk industri, terutama untuk industri pulp dan kertas. Selain itu, beberapa tahun terakhir, poplar juga dikembangkan sebagai alternatif bahan baku untuk produksi bioetanol (ORNL Review, 2007). Proses biosintesis selulosa pada poplar secara umum terbagi menjadi tiga tahap dan melibatkan kerja beberapa enzim seperti terlihat pada Gambar 3. Pada tahap pertama, sukrosa sintase pada membran plasma menyediakan substrat UDPglukosa ke mesin sintesis selulosa. Selulosa sintase selanjutnya menggunakan substrat UDP-glukosa untuk membentuk rantai glukan, sedangkan UDP akan digunakan kembali oleh sukrosa sintase. Terakhir, enzim Korrigan selulase pada membran mengedit atau memonitor perubahan rantai glukan menjadi mikrofibril selulosa (Delmer dan Haigler, 2002). 19 Keterangan : SUSY : Sukrosa sintase CESA : Selulosa Sintase KOR : Korrigan Sintase PM : Membran Plasma Gambar 3. Model biosintesis selulosa pada Populus sp (Joshi et.al.,2004). 2.4. Sukrosa Sintase Sukrosa sintase adalah enzim yang mengkatalisis reaksi konversi antara sukrosa dan uridin dipospat membentuk UDP-glukosa dan fruktosa. Dari berbagai penelitian yang terkait aktifitas sukrose sintase pada berbagai jenis tanaman diketahui bahwa sukrose sintase sangat berperan dalam mengatur metabolisme, mengontrol mobilisasi sukrosa ke berbagai jalur metabolisme yang penting, menghasilkan UDP glukosa untuk membentuk dinding sel bersama kompleks selulosa sintase. Enzim ini terlibat dalam biosintesis selulosa dengan menghubungkan UDP-glukosa ke selulosa sintase pada pembentukan dinding sel sekunder pada kapas (Amor, et.al. 1995). Sukrosa sintase bersama Kallosa sintase terlibat pada pembentukan tudung akar (Hong et.al., 2001) dan dengan β 1-3, 1-4 glukan sintase pada membran golgi (Konishi et.al.,2001). Pembentukan UDP glukosa oleh sukrose sintase merupakan mekanisme perlindungan energi ATP untuk pertumbuhan sel karena UDP yang dibentuk dari UDP-glukosa oleh β-glukan sintase dapat diubah kembali menjadi UDP-glukosa oleh sukrose sintase. Penekanan ekspresi sukrosa sintase melalui RNA antisense 20 pada wortel menyebabkan aktifitas sukrose sintase berkurang sehingga kuantitas selulosa juga berkurang. Sebaliknya, ekspresi sukrose sintase dari mutan “mung bean” Vigna radiata didalam Acetobacter xylinum tidak hanya merubah metabolisme sukrosa, tetapi juga meningkatkan produksi selulosa dengan cara mencegah akumulasi UDP selama biosintesis selulosa karena UDP adalah inhibitor selulosa sintase pada A. xylinum. Dengan demikian, akan sangat memungkinkan bagi peneliti untuk menghasilkan tanaman hutan transgenik dengan jumlah selulosa lebih tinggi melalui overekspresi sukrosa sintase (Konishi et.al., 2004). 2.5. Kloning Gen Kloning gen adalah penggandaan jumlah DNA rekombinan melalui proses perkembangbiakan sel bakteri. Teknik pembuatan DNA rekombinan meliputi pemotongan, pemindahan dan penyisipan gen yang diinginkan ke dalam sel inang. Prinsip dari teknik ini adalah ligasi fragmen DNA ke vektor yang dapat bereplikasi sendiri dalam inang. Sel inang yang digunakan dapat berupa sel bakteri atau sel ragi. Selanjutnya hasil ligasi ditransformasikan ke sel inang untuk mengamplifikasi atau memperbanyak jumlah fragmen DNA gen target. Keberhasilan kloning gen tergantung pada pemilihan dan kemampuan vektor untuk melanjutkan siklus hidupnya serta bereplikasi pada sel inang yang disisipi oleh gen target. Tahap berikutnya adalah proses seleksi transforman, yaitu sel-sel yang mengandung DNA rekombinan. Secara keseluruhan, proses kloning harus melewati 3 tahap, yaitu ligasi, transformasi dan seleksi transforman (Brown, 1991). Fragmen DNA gen target dapat diperoleh melalui pustaka DNA. Pustaka DNA terbagi menjadi 2 jenis, yaitu pustaka genom dan pustaka cDNA. Pustaka DNA merupakan kumpulan klon fragmen DNA yang mewakili keseluruhan genom suatu organisme, sedang pustaka cDNA adalah kumpulan klon gen yang mengkode suatu protein. Pemilihan jenis pustaka DNA didasari oleh tujuan kloning yang dilakukan. Kloning pustaka genom dilakukan untuk mengetahui fragmen genom secara utuh, seperti urutan basa nukleotida dari seluruh DNA genom, daerah promoter , daerah exon, dan termasuk intron yang terdapat hanya 21 pada DNA eukariot. Materi yang digunakan untuk pembuatan pustaka genom adalah DNA kromosom. Kloning pustaka cDNA dilakukan untuk mengetahui gen-gen yang mengkode protein dan fungsi protein penyandi gen melalui ekspresi protein tersebut. Kloning pustaka cDNA sangat penting dilakukan terutama untuk eukariot, karena pada eukariot terdapat intron, yaitu sekuen pada genom yang tidak menyandi protein. Tidak seperti pada prokariot yang tidak mempunyai intron, pada eukariot diperlukan sintesis cDNA untuk memperoleh gen penyandi protein tertentu yang terekspresi (Ausubel et.al, 1990). 2.5.1. Síntesis cDNA Sintesis cDNA adalah proses pembuatan copy DNA dengan menggunakan mRNA sebagai cetakan. Hasil sintesis cDNA berupa untai tunggal dan prosesnya disebut sebagai transkripsi balik atau sintesis cDNA untai pertama. Enzim yang digunakan untuk transkripsi balik disebut ”reverse transcriptase”, yaitu enzim DNA polimerase yang mentranskripsi RNA untai tunggal menjadi DNA untai tunggal. Enzim ”reverse transcriptase” yang umum dipakai bersumber dari HIV (human imunodeffyciency virus) tipe 1, M-MLV (Moloney Murine Leukimia virus), dan AMV yang berasal dari Avian Myeloblastosis virus (Protocol anline, 2006) Pemakaian material mRNA secara langsung sebagai sumber gen relatif lebih sulit dilakukan. Hal ini disebabkan molekul RNA bersifat tidak stabil dan mudah terdegradasi oleh RNAse. Oleh karena itu, pembuatan copy DNA dari mRNA melalui mekanisme transkripsi balik akan sangat memudahkan. cDNA hanya dapat disintesis dari mRNA. Hal ini disebabkan oleh karena ujung 3’ pada mRNA mempunyai ekor poli-A. Ekor poli-A tersebut akan berkomplementer dan menempel pada primer oligo d(T) untuk membentuk untai pertama cDNA.(Dale dan Scantz, 2002). Proses sintesis cDNA dengan sekuens yang lengkap merupakan suatu tahap yang sangat penting, terutama untuk mengidentifikasi, kloning dan karakterisasi gen-gen yang fungsional. cDNA dari eukariot dapat dikloning ke prokariot seperti E.coli dan diekspresikan atau ditranslasikan menjadi protein 22 tertentu. Pustaka cDNA yang lengkap menggambarkan total protein yang dapat terekspresi (Protocol online, 2006). Protokol PCR telah dikembangkan untuk sintesis cDNA dari ujung 3’ atau 5’ mRNA. Transkripsi balik dengan primer oligo d(T) menghasilkan untai pertama. Untai kedua cDNA dihasilkan dengan menggunakan enzim Taq DNA polimerase dan primer yang spesifik. Serangkaian proses amplifikasi dengan mesin PCR berlangsung dan menghasilkan jutaan copy DNA untai ganda (Glick, 1998). 2.5.2. Vektor Kloning Vektor berfungsi sebagai biotransport atau kendaraan yang membawa gen target dan memperbanyak DNA yang dibawanya di dalam sel inang. Beberapa jenis vektor kloning yang umum dipakai sebagai biotransport adalah plasmid, virus lambda bactheriophage dan cosmid. Pemilihan vektor tersebut tergantung pada ukuran fragmen DNA yang akan dibawa dan vektor yang paling sering dipergunakan adalah plasmid (Brown, 1991)). Plasmid secara umum diperoleh dari bakteri, yaitu berupa DNA untai ganda yang berbentuk bulat atau linier yang terpisah dari kromosom dengan ukuran yang bervariasi dari 1 kb – 500 kb. Kemampuannya untuk bereplikasi tanpa tergantung pada kromosom induk sangat menguntungkan terkait perannya sebagai vektor kloning. Replikasi plasmid dimulai dari ORI (origin of replication). Kriteria paling penting yang harus dimiliki plasmid agar dapat secara optimal bekerja sebagai vektor adalah adanya ori, marker seleksi (selectable marker) dan situs kloning (multiple cloning site). Beberapa sifat fenotip plasmid yang telah berhasil diidentifikasi adalah ketahanan terhadap antibiotik, produksi antibiotik, degradasi senyawa organik kompleks atau aroma dan lain-lain (Glick, 1998). Plasmid yang digunakan sebagai vektor haruslah berukuran kecil sehingga dapat membawa fragmen DNA asing yang besar dan mudah dikenal oleh peta restriksi. Terobosan dalam kloning telah memacu pengembangan DNA vektor yang baru yang lebih canggih dengan cara memodifikasi plasmid alam. Plasmidplasmid tersebut ditambah atau dikurangi dengan karakteristik tertentu sehingga 23 memudahkan proses kloning (Sambrook dan Russel, 2001). Plasmid yang digunakan untuk penelitian ini adalah pGemT-Easy. 2.5.3. Plasmid pGemT-Easy dan Strategi Ligasi pGemT-Easy merupakan vektor plasmid berbentuk linier berukuran 3015 pb (Gambar 4). Plasmid ini mempunyai beberapa kelebihan, diantaranya adalah mempunyai ukuran yang relatif kecil, mempunyai ori untuk replikasi dalam E. coli, membawa gen ketahanan untuk ampisilin, dan mempunyai multiple cloning site (MCS) dengan 19 sisi unik (Promega, 2005). Gambar 4. Peta vektor pGemT-Easy (Promega, 2005) MCS adalah suatu segmen pada plasmid yang panjangnya beberapa puluh sampai ratusan pasang basa untuk mengenali enzim restriksi tertentu dan merupakan tempat disisipkannya fragmen DNA asing. Situs pengenalan enzim restriksi tersebut, diantaranya EcoR I, BstZ I, Not I dan lain-lain, memudahkan pemutusan gen dari vektor plasmid. Plasmid ini juga mengandung gen lacZ sehingga dapat digunakan untuk seleksi koloni biru putih. Gen lacZ menyandi enzim β-galaktosidase yang mengubah isopropil- β galaktopiranosida (IPTG) dan substrat 5-bromo-4-kloro-3-indoil- β-galaktosidase (X-gal) menjadi biru. Apabila gen lacZ disisipi fragmen DNA asing, maka gen lacZ tidak akan terekspresi sehingga koloni berwarna putih (Promega, 2005). pGemT-Easy merupakan generasi terbaru plasmid pGem yang didisain secara khusus untuk mengkloning gen dari produk-produk PCR (polymerase 24 chain reaction). Salah satu keuntungan penggunaan vektor plasmid pGemT-Easy adalah, plasmid dapat langsung diligasikan ke DNA target tanpa melalui proses restriksi ataupun penambahan adaptor. pGemT-Easy didesain sedemikian rupa dengan penambahan basa timin pada ujung 3’ Vektor ini digunakan pada produk PCR yang proses amplifikasinya menghasilkan basa adenin pada ujung 3’. Basa adenin dari produk PCR akan ‘overhang’ dengan basa timin Produk PCR dengan hasil akhir penambahan basa adenin pada ujung 3’ dapat diperoleh dengan memilih enzim polimerase yang tepat. Beberapa enzim polimerase yang menghasilkan basa adenin pada daerah ujung 3’ diantaranya adalah enzim taq polimerase, Tfl dan Tth (Promega, 2005). 2.5.4. Transformasi Bakteri Transfomasi adalah memasukkan DNA rekombinan hasil ligasi ke dalam sel bakteri, biasanya adalah E. coli. Untuk meningkatkan efisiensi transformasi, beberapa bakteri harus diberi perlakuan fisik atau kimia tertentu yang akan meningkatkan kemampuannya mengambil DNA. Sel-sel yang mengalami perlakuan ini disebut kompeten sel (Brown, 1991). Ada empat teknik transformasi vektor ke dalam sel inang yang biasa digunakan, yaitu : 1. Teknik kejut panas, yaitu inkubasi pada suhu 420 C selama 45 detik 2. Elektroporasi, yaitu transfer DNA dengan bantuan medan listrik 3. Biolistik, yaitu penembakan sel target dengan mikro proyektil berkecepatan tinggi. 4. Mikro injeksi, yaitu injeksi DNA secara langsung ke dalam sel target menggunakan jarum yang sangat runcing. Metode transformasi yang digunakan untuk penelitian ini adalah metode kejut panas. DNA adalah molekul yang sangat hidrofil, sehingga secara normal DNA tidak dapat melewati dinding sel bakteri. Agar DNA gen target dapat melewati membran sel bakteri, maka pada metode kejut panas ini bakteri diberi perlakuan khusus dengan CaCl2 . Komponen ini akan membuat lubang kecil pada dinding sel bakteri dan dengan memberikan kejutan panas pada kompeten sel, maka gen target 25 dapat memasuki sel bakteri. Sel yang telah ditransformasi selanjutnya disebar ke media seleksi (Glick, 1998). 2.5.5. Seleksi dan Verifikasi Transforman Identifikasi sel transforman diseleksi berdasarkan gen penanda seleksi (selectable marker) yang terdapat pada plasmid. Gen yang biasa digunakan adalah gen ketahanan terhadap antibiotik tertentu dan gen lacZ untuk seleksi biru putih. Seleksi dilakukan dengan menumbuhkan bakteri pada media agar yang mengandung antibiotik dan hanya sel transforman yang mengandung gen ketahanan yang bersumber dari plasmid yang akan bertahan hidup. Untuk seleksi biru putih digunakan media yang mengandung X-gal sebagai substrat dan IPTG sebagai inducer. Dalam proses transformasi, sel kompeten yang dicampur dengan molekul DNA rekombinan akan mengalami 3 kemungkinan, yaitu sel kompeten tidak disisipi molekul DNA apapun, sel kompeten disisipi DNA vektor yang tidak membawa fragmen DNA, sel kompeten disisipi DNA vektor yang membawa fragmen DNA rekombinan. Secara teoretis, pada seleksi berdasarkan koloni biru putih, koloni yang berwarna putih dapat dipastikan merupakan sel yang membawa DNA rekombinan. Untuk meyakinkan hal tersebut, ada langkah berikutnya yang harus dilakukan meliputi isolasi DNA rekombinan, pemotongan DNA dengan enzim restriksi yang digunakan dalam pembuatan DNA rekombinan, pemisahan DNA melalui elektroforesis dan visualisasi DNA (Sambrook dan Russel, 2001). 26 BAB III BAHAN DAN METODE 3.1. Tempat dan Waktu Penelitian ini dilakukan di Puslit Bioteknologi LIPI Cibinong dari bulan Desember 2005 sampai Oktober 2007. 3.2. Bahan Penelitian Bahan tanaman yang digunakan dalam penelitian ini adalah jaringan “xylem” batang sengon yang diperoleh dari pohon induk dan anakan sengon kategori pohon plus dengan nomor koleksi 2 yang berasal dari kebun koleksi plasma nutfah Puslit Bioteknologi LIPI. Plasmid yang digunakan adalah pGemTEasy dengan ukuran 3015 pb. Primer yang digunakan adalah primer spesifik untuk sukrosa sintase, yaitu primer maju (AACTTGTTGTGCCACAC) dan primer mundur (ATA AGA ATGCGGCCGCTCTTTCATTCT CTGTTCCAGA). 3.3. Metode Penelitian Penelitian ini dilakukan dalam beberapa tahap. Skema tahapan metode dapat dilihat pada Lampiran 1. 3.3.1. Isolasi RNA Total Sengon Isolasi RNA total dilakukan menggunakan kit dan prosedur yang sama dengan protokol reagen Trizol. Sebanyak 0,1 g jaringan “xylem” batang sengon digerus bersama nitrogen cair hingga halus. Hasil gerusan dimasukkan ke dalam ependorf tube yang berisi 1 ml reagen Trizol lalu diinkubasi selama 10 menit pada suhu 15-300C. Campuran disentrifugasi dengan kecepatan 5000 rpm pada suhu 40C selama 15 menit. Supernatan diambil dan diekstraksi dengan 200 μl kloroform kemudian disentrifugasi kembali dengan kecepatan 5000 rpm pada suhu 40C selama 15 menit. Ke dalam supernatan ditambahkan 0,5 ml isopropanol kemudian disentrifugasi pada kecepatan 12000 rpm dan suhu 40C selama 15 menit. Pelet dipisahkan dari supernatan lalu dibilas dengan etanol 70% dingin lalu disentrifugasi kembali dengan kecepatan 5000 rpm pada suhu 40C selama 15 27 menit. Pelet dikeringkan pada suhu ruang , selanjutnya dilarutkan dengan penambahan 20 µl air bebas RNAse (GibcoBRL, 2005). 3.3.2. Sintesis cDNA Gen Sukrosa Sintase Melalui RT PCR Sintesis cDNA dilakukan dengan metode RT-PCR menggunakan kit ” ready to go RT PCR” dengan prosedur yang sama dengan protokol ready to go RT PCR dari Amersham Biosciences. Sintesis cDNA dilakukan dengan mencampurkan 3 µl RNA total (500 ng) dengan butiran “ ready to go RT PCR “ yang telah dilarutkan dengan 43,9 µl air bebas RNAse, 1,1 µl primer untai pertama pd (T)12-18 konsentrasi 0,5 µg/µl, 1 µl primer maju dengan konsentrasi 100 pmoles, dan 1 µl primer mundur (100 pmoles) dengan volume akhir 50 µl. RT-PCR dilakukan alat PCR Perkin Elmer dengan menginkubasi 0 reaksi pada suhu 42 C selama 30 menit dan campuran 0 suhu 95 C selama 5 menit (Amersham Bioscience, 2005). PCR cDNA sukrosa sintase dilakukan dengan kondisi predenaturasi 950C 5 menit, denaturasi 950C 0,45 menit, penempelan primer 54,40C 0,45 menit, pemanjangan rantai 720C 1 menit pemanjangan , dan pemanjangan akhir 720C 5 menit. PCR dilakukan sebanyak 30 siklus (Amersham Biosciences, 2005) 3.3.3. Elektroforesis Produk PCR DNA hasil amplifikasi dengan PCR dielektroforesis pada gel agarose konsentrasi 1,5% dengan buffer TAE 1x (yang dibuat dari larutan stok TAE 50x dengan komposisi 242 g Tris base; 57,1 ml asam asetat glasial dan 100 ml 0,5 M EDTA pH 8.0) selama 1 jam pada voltase 50 millivolt. Produk elektroforesis selanjutnya direndam dalam EtBr selama 15 menit dan visualisasi pita DNA dilakukan dengan menggunakan foto polaroid diatas lampu UV (Sambrook dan Russel, 2001) 3.3.4. Isolasi dan Pemurnian cDNA Produk PCR Isolasi dan pemurnian fragmen cDNA produk PCR dilakukan dengan menggunakan kit Sephaglas BrandPrep dengan prosedur yang sama dengan protokol Sephaglas BradPrep. Fragmen DNA sukrosa sintase pada gel agarose 28 dipotong dengan pisau steril. Potongan gel dicacah untuk mendapatkan potonganpotongan gel yang lebih kecil. 200mg gel agarose dilarutkan dengan 250 µl buffer pelarut gel. Campuran gel agarose kemudian divorteks dan diinkubasi pada suhu 600C selama 5-10 menit hingga gel agarose larut. Fragmen DNA diikat dengan menambahkan 5 µl Sephaglas BP, lalu diinkubasi selama 5 menit pada suhu ruang dan disentrifugasi dengan kecepatan 5000 rpm selama 30 detik. Pelet fragmen DNA dibilas dengan buffer pencuci. Selanjutnya pelet dilarutkan dengan 30 µl buffer elusi (Amersham Bioscience, 2006). 3.3.5. Kloning cDNA ke dalam Vektor pGemT-Easy Pengklonan cDNA ke vektor dilakukan dengan cara menginkubasi reaksi ligasi yang terdiri dari campuran 1 µl (50 ng ) vektor pGemT-Easy, 3µl DNA hasil purifikasi (10 ng/µl), 5 µl 2x buffer T4 DNA ligase, 1 µl T4 DNA ligase dan air bebas RNAse dengan volume akhir 10µl pada suhu 40C selama semalam (Promega, 2005) 3.3.6. Transformasi Gen ke E. coli DH5α Hasil ligasi fragmen cDNA dimasukkan ke kompeten sel E. coli DH5α. Pembuatan kompeten sel bakteri DH5α dilakukan dengan menggunakan kalsium klorida. Satu koloni bakteri E. coli DH5α dikultur dalam 5 ml media LB (10 g/l tripton, 5 g/l yeast ekstrak, 5 g/l NaCl, pH 7.0) selama semalam dalam inkubator shaker pada 370C dengan kecepatan 150 rpm. Selanjutnya 0,5 ml kultur ini dimasukkan dalam 5 ml LB dan diinkubasi pada kondisi yang sama hingga mencapai OD 0,4-0,6. Sebanyak 1 ml kultur ditransfer ke dalam tabung mikro steril dan disentrifugasi pada 5000 rpm, 40C selama 2 menit. Endapan yang diperoleh ditambah dengan 1 ml larutan 0,1 M CaCl2 dingin dan disuspensikan. Suspensi disentrifugasi dengan kondisi yang sama dan selanjutnya endapan disuspensikan kembali dalam 250 µl larutan 0,1 M CaCl2 dingin. Selanjutnya diinkubasi di es selama 10 menit dan bakteri siap untuk ditransformasi. Kompeten sel bakteri sebanyak 250 µl dicampur dengan 10 µl (50-100 ng) DNA hasil ligasi dengan plasmid dan diinkubasi di es selama 30 menit. Campuran tersebut kemudian diberi kejutan panas dengan cara diinkubasi pada suhu 420C 29 selama 45 detik. Kemudian dimasukkan kembali kedalam es selama 1 jam. Campuran tersebut kemudian diberi kejutan panas kembali pada suhu 420C selama 45 detik lalu dimasukkan ke dalam es. Campuran tersebut kemudian ditambah dengan 2 ml media LB dan diinkubasi pada suhu 370C selama 1 jam. Bakteri disebar pada media seleksi LB yang mengandung 50 mg/l ampisilin, 10 µl 0,1M IPTG dan 50 µl X-gal (20 mg/ml) dan diinkubasi pada suhu 370C selama semalam (Sambrook dan Russel, 2001) Hanya plasmid yang mengandung pGemT-Easy yang dapat hidup pada media seleksi yang mengandung antibiotik ampisilin. Adanya X-gal dan IPTG akan menghasilkan koloni putih dan biru. Koloni bakteri yang berwarna putih diisolasi karena mengandung mengandung pGemT-Easy pGemT-Easy rekombinan tidak rekombinan. mampu Bakteri yang menghasilkan β- galaktosidase sehingga X-gal tidak dapat diuraikan dan koloni tetap berwarna putih, sedang koloni yang mengandung pGemT-Easy non rekombinan akan berwarna biru karena mampu menguraikan X-gal. 3.3.7. Isolasi DNA Plasmid DNA plasmid pGemT-Easy rekombinan yang dikloning ke E. coli DH5α diisolasi menggunakan metode lisis dengan alkali. Satu koloni E. coli DH5α recombinan dikultur dalam 10 ml media LB yang mengandung 10 µl ampisilin 50µl/ml dan diinkubasi pada inkubator shaker 150 rpm pada suhu 370C selama semalam. Kultur bakteri disentrifugasi dengan kecepatan 5000 rpm pada suhu 40C selama 2 menit. Endapan bakteri kemudian disuspensikan dalam 100 µl BRS dan 200 µl LS lalu diamkan selama 3 detik. Setelah lisis tambahkan buffer penetralisasi 150 µl koAC dan sentrifugasi pada suhu 40C selama 10 menit dengan kecepatan 10000 rpm. Supernatan diambil dan ditambahkan dengan 250 µl isopropanol, lalu divorteks sampai bercampur dan disentrifugasi kembali pada suhu 40C selama 5 menit dengan kecepatan 10000 rpm. Kedalam pelet ditambahkan 500 µl aquades dan diberi perlakuan dengan penambahan 0,5 µl RNAse 20 mg/ml selama 2 jam pada suhu 370C untuk menghilangkan sisa RNA. Larutan yang mengandung DNA plasmid diekstraksi dengan campuran fenol dan kloroform dengan perbandingan 24:1 lalu disentrifugasi pada suhu 40C selama 5 30 menit dengan kecepatan 10000 rpm. Fase atas diambil dan ditambah dengan 2 volume etanol absolut dan dibiarkan pada suhu - 200C selama semalam. Selanjutnya larutan tersebut disentrifugasi pada suhu 40C selama 20 menit dengan kecepatan 12000 rpm. Pelet dibilas dengan 500 µl etanol 70% dan disentrifugasi kembali pada suhu 40C selama 5 menit dengan kecepatan 10000 rpm. Pelet dilarutkan dengan penambahan 30 µl H2O (Sambrook dan Russel, 2001) 3.3.8. Sekuensing cDNA Sekuensing DNA dilakukan dengan menggunakan pereaksi Big Dye terminator dan mesin sekuensing ABI Prisma 3. Komposisi reaksi sekuensing terdiri dari 2 µl BigDye terminator, 1,5 µl DNA cetakan (200 ng), 0,2 µl primer M13-FP (20 pmol/ µl) dan 6,3 µl ddH2O dengan volume total 10 µl. Prinsip kerja BigDye terminator adalah pewarnaan ujung ddNTP yang terdiri dari ddATP, ddTTP, ddCTP dan ddGTP dengan pewarna berpendar yang memiliki panjang gelombang berbeda-beda. Selanjutnya gen sukrosa sintase diamplifikasi menggunakan mesin PCR dengan pereaksi yang salah satu komponennya adalah ddNTP yang telah diberi warna berpendar. Hasil amplifikasi secara langsung dielektroforesis melalui suatu kapiler (capillary electrophoresis) dan urutan basa nukleotida yang teramplifikasi dideteksi melalui suatu ”sequence analyzers”. Output yang diperoleh dari ”sequence analyzers” adalah berupa kromatogram urutan basa nukleotida dengan warna 4 puncak basa nukleotida yang spesifik (Applied Biosystem, 2002). 3.3.9. Analisis Homologi Berdasarkan hasil pengurutan basa nukleotida gen penyandi sukrosa sintase yang diperoleh dari sengon, maka selanjutnya dilakukan analisis kesamaan dan tingkat homologi dengan sekuen gen sukrosa sintase dari organisme lain yang ada di dalam bank gen NCBI (National Center for Biotechnology Information), http://www. ncbi. nlm. nih.gov/BLAST) dengan prinsip BLAST (Basic Local Alignment Search Tools). Untuk analisis tingkat kesamaan basa nukleotida digunakan program BLASTN (BLAST Nucleotide), sedang untuk analisis tingkat homologi digunakan program BLASTP (BLAST Protein). Analisis tingkat 31 kekerabatan fragmen sukrosa sintase sengon dengan sukrosa sintase dari organisme lain dapat disajikan dalam bentuk pohon filogenetik. Pohon pilogenetik dibuat dengan cara membandingkan deduksi asam amino gen penyandi sukrosa sintase yang diperoleh dari sengon dan organisme lain yang terdapat pada bank gen dengan program ClustalW (http : //www.ncbi.nlm.nih.gov/ClustalW). 32 BAB IV HASIL DAN PEMBAHASAN 4.1. Isolasi RNA Total RNA total sengon diisolasi dengan reagen Trizol dari jaringan “xylem” batang sengon yang tua (berumur 5-10 tahun) dan bibit sengon yang berumur 3- 4 bulan. Kualitas RNA yang diisolasi dari batang yang tua tidak sebaik RNA yang diperoleh dari batang yang muda dan hampir sebagian besar terdegradasi. Degradasi RNA total ditandai dengan tidak adanya ke dua pita rRNA 28S dan rRNA 18S dan pada bagian bawah lajur migrasi RNA pada elektroforesis muncul pita tebal yang merupakan potongan-potongan RNA yang terdegradasi oleh nuklease (Lin, et. al., 1996). Karena kualitas RNA yang diperoleh akan sangat mempengaruhi optimalisasi sintesis cDNA, maka RNA yang digunakan untuk penelitian ini bersumber dan diisolasi dari bibit sengon dengan umur 3-4 bulan. Perbedaan kualitas RNA yang diperoleh kemungkinan disebabkan oleh karena aktivitas nuklease yang lebih tinggi pada batang sengon tua dibanding dengan sengon yang berumur 3-4 bulan. Selain itu, kandungan senyawa fenolik yang terdapat pada tanaman juga turut mempengaruhi tingkat keberhasilan isolasi RNA. Semakin tua umur pohon, maka kandungan zat ekstraktif, terutama senyawa-senyawa fenolik akan semakin besar. Berbeda dengan tanaman muda yang kandungan senyawa fenoliknya masih relatif sedikit, komponen ini akan mengganggu proses isolasi RNA dari batang kayu yang berumur lebih tua. Hasil kuantifikasi RNA total dengan spektrofotometer pada panjang gelombang 260 nm menunjukkan bahwa konsentrasi RNA total yang diperoleh cukup baik dan memadai untuk sintesis cDNA. Konsentrasi RNA bervariasi dari 126 μg sampai 157 μg dengan variasi rasio OD260/OD280 antara 1.81-1.92. Nilai OD yang berada dalam kisaran range 1.8-2 menunjukkan kualitas RNA dengan kemurnian cukup tinggi (Manchester, 1996). Analisis RNA total melalui elektroforesis pada gel agarose menghasilkan integritas 2 pita RNA yang dominan dan seimbang, yaitu r RNA 28S dan 18S (Gambar 5). 33 1 2 3 4 5 6 28S 18S Gambar 5. Integritas pita RNA 28S dan 18S dari RNA total Keterangan : Nomor 1-6: RNA total batang sengon umur 4 bulan 4.2. Sintesis cDNA cDNA disintesis melalui proses transkripsi balik (riverse transcription) dengan menggunakan RNA total sebagai bahan cetakan. Primer yang digunakan untuk transkripsi balik adalah oligo d(T) sehingga hanya mRNA yang mengandung poli-A pada ujung 3’ yang akan disintesis menjadi cDNA, sedang rRNA dan tRNA yang tidak memiliki poli-A pada ujung 3’ tidak dapat membentuk cDNA. Hasil elektroforesis cDNA yang divisualisasikan dari gel agarose dibawah sinar UV menunjukkan bahwa proses sintesis cDNA berhasil dilakukan. Hal ini dapat dilihat dari kualitas pita cDNA sengon dibandingkan dengan kontrol RNA dari kit “ready to go RT-PCR” seperti pada Gambar 6. 1 2 3 Gambar 6. Sintesis cDNA dari sengon Keterangan : 1. Kontrol positif, 2. Kontrol negatif, 3. Hasil sintesis cDNA dari RNA 34 Proses PCR untuk mengamplifikasi cDNA menggunakan primer spesifik juga berhasil dilakukan dan menghasilkan fragmen dengan ukuran 341 pb. Fragmen cDNA yang berukuran 341 pb ini merupakan kandidat fragmen gen penyandi sukrosa sintase dari tanaman sengon (Gambar 7). Fragmen tersebut selanjutnya diisolasi dan dimurnikan dari gel agarose dan diintroduksikan ke E.coli. 1 2 12000 pb 5000 pb 2000 pb 1650 pb 1000 pb 850 pb 650 pb 500 pb 400 pb 300 pb 200 pb 100 pb 341 pb Gambar 7. Pita hasil PCR cDNA Keterangan : Nomor 1. Marker 1 Kb DNA plus Ladder; 2. Fragmen cDNA 4.3. Kloning Fragmen Plasmid pGemT-Easy yang mengandung gen lac-Z digunakan sebagai vektor ligasi. Hasil ligasi fragmen sukrosa sintase dengan pGemT-Easy diintroduksikan ke bakteri E. coli galur DH5α dan disebar ke media seleksi yang mengandung ampisilin, IPTG dan X-gal. Koloni E. coli yang mengandung transforman (plasmid rekombinan) diseleksi berdasarkan pembentukan koloni biru putih. Koloni E. coli yang berwarna biru tidak mengandung transforman, sehingga gen lacZ yang menyandi enzim β-galaktosidase masih aktif terekspresi dan mengubah substrat X-gal yang tidak berwarna menjadi biru. Koloni target adalah koloni putih yang mengandung transforman, dimana gen lacZ disisisipi oleh fragmen sukrosa sintase sehingga tidak terekspresi dan menyebabkan koloni tetap berwarna putih (Gambar 8). 35 Konfirmasi koloni putih dengan PCR dilakukan untuk mengetahui fragmen sukrosa sintase yang menyisip pada pGemT-Easy. PCR koloni yang menghasilkan pita DNA dengan ukuran 341 pb menunjukkan bahwa koloni putih adalah transforman yang disisipi fragmen sukrosa sintase. Ini berarti, fragmen sukrosa sintase berhasil dikloning ke vektor pGemT-Easy didalam E. coli galur DH5α. Koloni tranforman putih Koloni biru Gambar 8. Koloni E. coli hasil transformasi berupa koloni putih 4.4. Verifikasi Sisipan dan Analisis Situs Pemotongan Enzim Restriksi Hasil analisis peta restriksi fragmen DNA sengon menunjukkan bahwa pada fragmen tersebut tidak terdapat situs restriksi untuk enzim EcoR1 seperti pada plasmid pGemT-Easy. Hal ini tentu saja akan memudahkan pemisahan fragmen DNA sukrosa sintase sengon dari plasmid tersebut. Restriksi fragmen sukrosa sintase dengan enzim EcoR1 berhasil dilakukan. Hal dapat dilihat dari terbentuknya 2 pita spesifik, yaitu pita pada daerah 3015 pb yang spesifik untuk plasmid pGemT-Easy dan pita pada daerah 341 pb untuk fragmen sukrosa sintase (Gambar 9). 1 3015 pb 341 pb 2 2000 pb 1650 pb 1000 pb 850 pb 650 pb 500 pb 400 pb 300 pb 200 pb 100 pb Gambar 9. Hasil restriksi fragmen sukrosa sintase dengan EcoR1; Nomor 1. Restriksi sukrosa sintase; 2. Marker 1 kb plus DNA ladder 36 Analisis peta situs pemotongan enzim restriksi pada fragmen sukrosa sintase dibuat dengan program NEB Cutre (Gambar 10). Hasil pemetaan enzim restriksi menunjukkan bahwa fragmen sukrosa sintase tidak memiliki situs pemotongan yang sama dengan situs pemotongan seperti pada plasmid pGemT-Easy. Perbedaan situs tersebut akan memudahkan pemisahan sisipan fragmen DNA sukrosa sintase dari plasmid pGemT-easy. Peta restriksi sangat bermanfaat untuk menentukan enzim restriksi yang paling tepat untuk pembuatan kontruksi gen dan pustaka genom. Gambar 10. Peta restriksi fragmen sukrosa sintase berdasarkan NEB Cutter 4.5. Pengurutan Fragmen Sukrosa Sintase Hasil pengurutan fragmen Sukrosa sintase menghasilkan 341 pb nukleotida. Basa-basa tersebut menyandi 113 asam amino. Urutan basa nukleotida 37 DNA dan profil asam amino yang diperoleh dari hasil sekuensing DNA plasmid dapat dilihat pada Gambar 11 . 9 18 27 36 45 54 5' GTG TTA AGG GTG AAG AGG AGA ATT AAT TTG TTC AAG ACT GTG ATA TTG AGA TGT --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --Val Leu Arg Val Lys Arg Arg Ile Asn Leu Phe Lys Thr Val Ile Leu Arg Cys 63 72 81 90 99 108 CCG AAA ATG GGG CTG CCG GGT GCT AAC TTG TTG TGC CAC ACA GCT TTC ACC CTT --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --Pro Lys Met Gly Leu Pro Gly Ala Asn Leu Leu Cys His Thr Ala Phe Thr Leu 117 126 135 144 153 162 CCC GTC CTC ACC CGG GTG GTT CAT GGT ATC GAT GTG CTC GAT CCC AAG TTA AAC --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --Pro Val Leu Thr Arg Val Val His Gly Ile Asp Val Leu Asp Pro Lys Leu Asn 171 180 189 198 207 216 ATT GTT CAC ACG GCG ATG TCA TGT GTA TTT ACT TCC CCT TCA AGA GAA GTT AGC --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --Ile Val His Thr Ala Met Ser Cys Val Phe Thr Ser Pro Ser Arg Glu Val Ser 225 234 243 252 261 270 AGA TAT CAG GGC CTT GGA AGT CGA TCT GGG AGA AGT TTT AGG AAG GGG AAG GAA --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --Arg Tyr Gln Gly Leu Gly Ser Arg Ser Gly Arg Ser Phe Arg Lys Gly Lys Glu 279 288 297 306 315 324 AAG AAA ATA GGA TCC GGG ATG AAA AGC GGA ACA AGG CAA ATG TTT TAT AGC AAG --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --- --Lys Lys Ile Gly Ser Gly Met Lys Ser Gly Thr Arg Gln Met Phe Tyr Ser Lys 333 GAA TGG ACC GGG CCC CA 3' --- --- --- --- --- -Glu Trp Thr Gly Pro Gambar 11. Urutan basa nukleotida fragmen sukrosa sintase dan deduksi asam aminonya 4.6. Analisis Homologi Sukrosa Sintase Urutan basa nukleotida fragmen sukrosa sintase dianalisis dan dibandingkan dengan database gen penyandi sukrosa sintase dari Bank DNA menggunakan program BLAST ( Basic Local Alligment Search Tool) untuk mengetahui identitas dan tingkat homologinya dengan fragmen dan gen yang telah diketahui (Tabel 2). BLAST adalah program penyejajaran urutan basa nukleotida atau asam amino berdasarkan skor tertinggi (Mount, 2001). 38 Tabel 2. Penyejajaran nilai kesamaan basa nukleotida sukrosa sintase dari beberapa tumbuhan (Blastn NCBI, 2007) Kode Sumber Skor Maks Perbandingan Nilai Kesamaan Sekuen E AB022092.1 Citrus unshiu, CitSUS1 mRNA untuk sukrosa sintase, cds komplit 91.5 33% 2e-15 78% DQ487290.1 Viscum album subsp. Album sukrosa sintase mRNA, cds sebagian 86.0 34% 8e-14 76% AY341026.1 Populus tremuloides sukrosa sintase mRNA, cds komplit 82.4 32% 1e-12 76% U73588.2 Gossypium hirsutum sukrosa sintase mRNA, cds komplit 82.4 36% 1e-12 75% AJ311496.1 Pisum sativum mRNA sukrosa sintase isoform 3 (sus3 gene) 73.4 31% 5e-10 75% A. thaliana SUS1 (SUKROSA SINTASE 1); UDP-glikosil NM_122090.3 transferase sukrosa sintase (SUS1) mRNA, cds komplit 69.8 36% 6e-09 73% X70990.1 A.thaliana sukrosa sintase 69.8 36% 6e-09 73% D10266.1 Vigna radiata var. radiata vss1 mRNA sukrosa sintase, cds komplit 69.8 32% 6e-09 75% AF030231.1 Glycine max sukrosa sintase (SS) mRNA, cds komplit 68.0 22% 2e-08 79% X69773.1 V. .faba mRNA sukrosa sintase 64.4 31% 3e-07 74% Hasil BLAST basa nukleotida dengan program BLASTN menggunakan fragmen sukrosa sintase yang diperoleh menunjukkan bahwa fragmen sukrosa sintase sengon memiliki kesamaan urutan basa nukleotida dengan Glycine max, Citrus unshiu, Vigna radiata dan Arabidopsis thaliana dengan nilai kesamaan berurutan 79%, 78%, 75% dan 73%. Batas standar nilai E (expectacy) yang dapat digunakan sebagai batas terendah untuk tingkat kepercayaan pada nilai kesamaan dan homologi antar organisme adalah pangkat -4. Nilai E yang berada pada range pangkat -7 sampai -15 dari penyejajaran basa nukleotida sengon dibanding organisme lain seperti pada Tabel 2 menunjukkan bahwa tingkat homologi gen penyandi sukrosa sintase sengon cukup tinggi. Selain BLAST berdasarkan basa nukleotida, juga dilakukan BLAST berdasarkan deduksi asam amino. Hasil BLAST asam amino yang menyandi protein sukrosa sintase menunjukkan asam amino sengon memiliki tingkat 39 homologi yang mirip dengan Glycine max, Citrus unshiu, Vigna radiata dan Arabidopsis thaliana dengan nilai berurutan 54.3, 51.2, 54.3 dan 51.6. (Tabel 3) . Tabel 3. Penyejajaran deduksi protein sukrosa sintase dari beberapa Tumbuhan (Blastp NCBI, 2007) Kode Sumber Skor Nilai E P13708 Glycine max 54.3 2e-06 Q01390 Vigna radiata 54.3 2e-06 ABP88869 Medicago sativa 53.9 3e-06 CAB40795.1 Medicago truncatula 53.9 3e-06 CAA09910.1 Pisum sativum 53.9 3e-06 P31926 Vicia faba 53.9 3e-06 CAA50317.1 A. thaliana 51.6 1e-05 BAA89049.1 Citrus unshiu 51.2 2e-05 ABF50715.1 Viscum album 50.8 2e-05 AAD28641.1 Gossypium hirsutum 50.8 2e-05 ABB53602.1 Eucalyptus grandis 50.4 3e-05 AAR03498.1 Populus tremuloides 50.1 3e-05 Hasil BLAST dan penyejajaran menggunakan deduksi asam amino menunjukkan bahwa, gen penyandi sukrosa sintase yang diperoleh dari sengon belum komplit dan sekuen tersebut berada pada daerah tengah gen penyandi sukrosa sintase dari berbagai spesies yang telah berhasil diidentifikasi (Lampiran 2). Untuk itu, masih perlu dilakukan upaya untuk mendapatkan gen yang utuh. 4.7. Analisis Tingkat Kekerabatan Tingkat kekerabatan sukrosa sintase sengon dibandingkan dengan sukrosa sintase dari berbagai spesies yang lain diuji berdasarkan penyejajaran deduksi asam amino. Perbandingan deduksi asam amino tersebut kemudian digunakan sebagai dasar pembuatan profil pohon pilogenetik gen sukrosa sintase dengan menggunakan program piranti DNASIS (Gambar 12). Analisis kekerabatan berdasarkan pengelompokan/kluster menunjukkan bahwa gen penyandi sukrosa sintase berada satu kluster dengan Glycine max, Vigna radiata dan lain-lain. Satu hal perlu ditelaah lebih lanjut adalah bahwa gen penyandi sukrosa sintase sengon 40 berada satu subkluster dengan Arabidopsis thaliana yang bukan merupakan jenis pohon berkayu. Hal ini mungkin terjadi karena gen penyandi sukrosa sintase yang diperoleh belum dalam bentuk sekuen yang utuh atau cDNA komplit. Viscum album Citrus unshiu Gossypium hirsutum Eucalyptus di di Populus tremuloides A. thaliana Paraserianthes falcataria Medicago sativa Medicago truncatula Pisum sativum Vicia faba Glycine max Vigna radiata Gambar 12. Nilai kekerabatan sukrosa sintase berbagai tumbuhan berdasarkan deduksi asam amino 4.8. Pembahasan Umum Isolasi dan kloning gen penyandi sukrosa sintase dari sengon melalui teknik RT PCR yang telah dilakukan berhasil memperoleh 341 pb nukleotida. Fragmen cDNA yang diperoleh merupakan kandidat gen sukrosa sintase. Hal ini dapat dibuktikan berdasarkan uji tingkat homologi sekuen basa nukleotida dan asam amino fragmen sukrosa sintase yang diperoleh dibandingkan dengan gen penyandi sukrosa sintase dari organisme atau species yang lain menggunakan program BLAST. Penelitian ini merupakan studi awal untuk memperbaiki kualitas kayu sengon melalui peningkatan deposisi selulosa. Untuk itu masih perlu dilakukan penelitian lanjutan yang terkait dengan ”gene establishment” dan kloning gen 41 sukrosa sintase kembali ke tanaman sengon. Gen yang diperoleh dari sengon masih berupa cDNA parsial. Gene establishment bertujuan untuk memperoleh pustaka cDNA yang utuh. Pustaka cDNA yang utuh dapat diperoleh melalui tahapan-tahapan seperti, penapisan pustaka cDNA dengan menggunakan fragmen sukrosa sintase sengon sebagai pelacak (probe) dan sebagai dasar desain primer untuk metode “DNA walking” seperti metode RACE (Rapid Amplified cDNA End). Keunggulan metode RACE adalah amplifikasi cDNA dapat dilakukan pada ujung 3’ atau 5’ sehingga dapat diperoleh sekuen yang utuh. Selanjutnya, gen yang diperoleh diekspresikan ke prokariot untuk melihat apakah protein sukrosa sintase tersebut secara fungsional dapat meningkatkan deposisi selulosa. Kloning gen sukrosa sintase sengon yang telah diperoleh kembali sengon untuk peningkatan deposisi selulosa dapat dilakukan melalui dua pendekatan, yaitu kloning cDNA genomik dan kloning melalui perantaraan vektor plasmid. Kloning melalui cDNA genomik dapat dilakukan dengan cara penembakan partikel gen. Akan tetapi metode ini memiliki beberapa kelemahan, diantaranya adalah bahwa lokasi tempat gen akan menyisip bersifat random/acak sehingga tidak dapat diprediksi, jumlah “copy” besar kemungkinan banyak dan biayanya lebih mahal. Kloning melalui perantaraan vektor plasmid dapat dilakukan dengan cara transformasi ke Agrobacterium tumefaciens. Kloning melalui vektor plasmid relatif lebih menguntungkan dibanding kloning cDNA genomik karena DNA target dapat langsung menyisip ke DNA inti pada kromosom, jumlah “copy” lebih sedikit dan biayanya relatif lebih murah. 42 BAB V SIMPULAN DAN SARAN 5.1. Simpulan Isolasi RNA total sengon dan sintesis cDNA dengan kualitas yang baik melalui teknik RT PCR berhasil dilakukan. Berdasarkan hasil pengurutan basa nukleotida dan uji homologi dapat disimpulkan bahwa proses kloning gen penyandi sukrosa sintase berhasil dilakukan dan diperoleh fragmen gen dengan ukuran 341 pb. Fragmen ini memiliki kesamaan yang tinggi dengan sukrosa sintase pada Glycine max (79%), Citrus unshiu (78%), Vigna radiata (75%) dan Arabidopsis thaliana (73%) 5.2. Saran Perlu dilakukan penelitian lanjutan, yaitu “gene establishment” untuk memperoleh sekuen gen yang utuh yang meliputi tahap penapisan pustaka cDNA menggunakan fragmen sukrosa sintase sengon yang telah diperoleh sebagai pelacak dan sebagai dasar desain primer dengan metode RACE (Rapid Amplified cDNA End). Gen dengan sekuen utuh siap diklon dengan promoter dan gen penanda sehingga dapat diintroduksikan ke tanaman sengon melalui transformasi genetik, baik secara langsung menggunakan “particle gun” atau melalui Agrobacterium tumefaciens untuk memperoleh sengon yang kadar selulosanya tinggi. 43 DAFTAR PUSTAKA Amersham Biosciences. 2005. Protocol Ready To Go RT_PCR Beads. England. Amersham Biosciences. 2006. Sephaglas BrandPrep Kit Instructions. USA. Amor, Y., Haigler, C.H., Johnson, S., Wainscott, M., Delmer, D.P.1995. A membran associated form of sukrosa sintase and its potential role in synthesis of cellulosa and callose in plants. Proc Natl Acad Sci USA 92: 9353-9357. Applied Biosystem. 2002. Protocol Big Dye Terminator v3.1 Cycle Sequensing Kit. Ausubel, F.M., Brent, R., Kingston, R.E., Moore, D.D., Seidman, J.G., Smith, J.A., Struhl, K. 1990. Current Protocols in Molecular Biology. Volume 1. John Wiley and Sons. Blastn NCBI. 2007. http : //www.ncbi.nlm.nih.gov/blast Bohinski, R.C. 1987. Modern concepts in biochemistry. Prentice Hall International. Page : 398. Bowyer, J.L., Shmulsky, R., Haygreen, J.G. 2000. Forest products and wood science. Fourth edition. Iowa state press. Page: 299-314. Brown, T.A. 1987. Pengantar Kloning Gen. Yayasan Essentia Medica. Yogyakarta. Dale, J.W., Scantz, M. 2002. From Genes to Genomes: Concepts Application of DNA Technology. John Willwy & Sons. Delmer, D.P., Haigler, C.H. 2002. The regulation of metabolic flux to cellulose, a major sink for carbon in plants. Metabolic Enginering. Page :22-28. GibcoBRL. 2005. Protocol of Trizol Reagent. Life Technology. Glick, B.R., Pasternak, J.J. 1998. Molecular Biotechnology. Second Edition. ASM Press. Washington DC. Hidayat, J. 2002. Informasi Singkat Benih. No 23. Direktorat Perbenihan Tanaman Hutan. Hong, Z., Zhang, Z., Olson, J.M., Verma, D.P.S. 2001. A novel glucose transferase in part of the callose synthase complex. Plant Cell 13:769. Joshi, C.P., Bhandari, S., Ranjan, P., Kalluri, U.C., Liang, X., Fujino, T., Samuga, A. 2004. Research review: Genomics of cellulose biosynthesis in poplar. New Phytologist. Page : 53-54. Kartiwa, W. H. 2006. Variabilitas massa jenis kayu daun lebar tropis terhadap karakter serat, kimia dan pulp sulfat. J. Ilmu dan Teknologi Kayu Tropis. No 2 Volume 4. Halaman : 71-75. Kartiwa .2006. www.dephut.go.id. Konishi, T., Ohmiya, Y., Hayashi, T. 2001. Evidence that sukrosaloaded into the phloem of a poplar leaf is used directly by sukrosa sintase associated with 44 various β glucan synthesis in the stem. Plant Physiology 134. Page : 11461152. Lea, P.J., Leegood, R.C. 1994. Plant Biochemistry and Molecular Biology. John Wiley and Sons.Page : 89-109. Lin, R.J., Kim, D.H., Castanotto, D., Westaway, Rossi, J.J. 1996. RNA preparation from yeast cells. Di dalam : A Laboratory Guide to RNA. Isolation, Analysis and Synthesis. Krieg PA, New York, Willey Inc. Manchester, K.L. 1996. Use of UV method for measurement of protein and nucleic acid concentrations. BioTech 20. Page : 968-920. Mount, D. W. 2001. Bioinformatics : Sequence and Genome Analysis. New York. Cold Spring Harbor Laboratory Press. Neale, D.B. 2002. Discovering the genetics of wood leads to quality improvement. NRI. Oak Ridge National Laboratory Review (ORNL Review). 2007. The People’s Tree. Volume 40 No. 1. Pari, G. 1996. Analisis komponen kimia dari kayu sengon dan kayu karet pada beberapa macam umur. Buletin Penelitian Hasil Hutan 14. Halaman: 321327. Promega. 2005. Technical Manual pGemT and pGemT-Easy Vector System. USA. Read, S.M., Bacic, T. 2002. Prime time for cellulose. Science 295. Page : 59-60. Sambrook, J., Russel, D.W. 2001. Molecular Cloning. A Laboratory Manual, Third edition, Volume 1, CSHL Press, New York . Siagian, R.M., Roliadi, H., Suprapti, S., Komarayati, S. 2003. Studi peranan fungi pelapuk dalam proses biodelignifikasi kayu sengon. J. Ilmu dan Teknologi Kayu Tropis Vol. 1 No 1. Star, F., Star, K., Loope, L. 2003. Plants of Hawaii. Reports Plants of Hawaii www.protocol online . 2006. cDNA synthesis and Cloning. 45 Lampiran 1. Alur Penelitian Isolasi dan Kloning Gen Penyandi Sukrosa Sintase Pada Tanaman Sengon (Paraserianthes falcataria) Isolasi RNA total tanaman sengon RT-PCR Pemurnian Produk PCR Ligasi ke pGEMT easy Transformasi bakteri E. coli (DH5α) Uji Sisipan dalam Media Seleksi yang mengandung amphicilin Analisis Restriksi dan PCR dari E. coli Sekuensing Uji Homologi Terhadap Sukrosa Sintase 46 Lampiran 2. Deduksi asam amino yang menyandi sukrosa sintase sengon dibandingkan dengan asam amino sukrosa sintase dari berbagai tumbuhan Citrus unshiu KQQGLDITPQILIITRLLPDAVGTTCGQRLEKVYGTKYSDILRVPFRTEKGVVRKWISRF Gossypium hirsutum KQQGLNITPRILIITRLLPDAVGTTCGQRLEKVYGTEHSDILRVPFRTEKGIVRKWISRF Eucalyptus grandis KQQGLDITPRILIVTRLLPDAVGTTCNQRLEKVFGTEYSHILRVPFRTEKGMVRKWISRF Populus tremuloides KQQGLDIIPRILIITRLLPDAVGTTCGQRLEKVYGSEHCDILRVPFRDEKGMVRKRISRF Viscum album KQQGLDITPRILIVTRLLPDVVGTTCNQRLEKVFGTEHTHILRVPFRADKGIVRQWISRF Medicago sativa KKQGLDIIPRILIITRLLPDAVGTTCGQRLEKVYGTEHCHILRVPFRDEKRIVRKWISRF Medicago truncatula KKQGLDIIPRILIITRLLPDAVGTTCGQRLEKVYGTEHCHILRVPFRDTKGIVRKWISRF Visum sativum KKQGLDIVPRILIITRLLPDAVGTTCGQRLEKVYGTEHCHILRVPFRDQKGIVRKWISRF Vicia faba KKQGLDIVPRILIITRLLPDAVGTTCGQRLEKVYGTEHCHILRVPFRDQKGIVRKWISRF Glycine max KQQGLDIVPRILIITRLLPDAVGTTCGQRLEKVFGTEHSHILRVPFRTEKGIVRKWISRF Vigna radiata KQQGLDIVPRILIITRLLPDAVGTTCGQRLEKVFGTEHSHILRVPFRTENGIVRKWISRF A. thaliana KQQGLNIKPRILILTRLLPDAVGTTCGERLERVYDSEYCDILRVPFRTEKGIVRKWISRF P. falcataria ----------------------------------------VLRVKRR------------:*** * Citrus unshiu E-VWPYLETYTEDVAVEIAKELQGKPDLIIGNYSDGNIVASLLAHKLGVTQCTIAHALEK Gossypium hirsutum EKVWPYLETYTEDVAHEISKELHGTPDLIIGNXSDGNIVASLLAHKLGVTQCTIAHALEK Eucalyptus grandis E-VWPYLETYTEDVANEIAGELQGKPDLIIGNYSDGNIVASLLAHKLGVTQCTIAHALEK Populus tremuloides E-VWPYLETYTEDVAAEIAKELQGKPDLIIGNYSDGNVVASLLAHKLGVTECTIAHALEK Viscum album E-VWPYLENFTEDVALEIAGELQGKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK Medicago sativa E-VWPYLETYTEDVAHELAKELQSKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK Medicago truncatula E-VWPYLETYTEDVAHELAKELQGKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK Visum sativum E-VWPYLETYTEDVAHELAKELQGKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK Vicia faba E-VWPYLETYTEDVAHELAKELQGKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK Glycine max E-VWPYLETYTEDVAHELAKELQGKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK Vigna radiata E-VWPYLETYTEDVAHELAKELQGKPDLIVGNYSDGNIVASLLAHKLGVTQCTIAHALEK A. thaliana E-VWPYLETYTEDAAVELSKELNGKPDLIIGNYSDGNLVASLLAHKLGVTQCTIAHALEK P. falcataria ------------------------------------------------------------ Citrus unshiu TKYPDSDIYWKNLDDKYHFSCQFTADLIAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Gossypium hirsutum TKYPDSDIYWKKLEDKYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Eucalyptus grandis TKYPESDIYWKKFEEKYHFSCQFTADLIAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Populus tremuloides TKYPDSDIYWKKFDEKYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Viscum album TKYPDSDIYWKNLEEKYHFSCQFTADLIAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Medicago sativa TKYPESDIYWKKFEEKYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDKVGQYESHTAF Medicago truncatula TKYPESDIYWKKFEEKYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDKVGQYESHTAF Visum sativum TKYPESDIYWKKFEEKYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Vicia faba TKYPESDIYWKKFEEKYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Glycine max TKYPESDIYWKKLEERYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF Vigna radiata TKYPESDIYWKKLEERYHFSCQFTADLFAMNHTDFIITSTFQEIAGSKDTVGQYESHTAF A. thaliana TKYPDSDIYWKKLDDKYHFSCQFTADIFAMNHTDFIITSTFQEIAGSKETVGQYESHTAF P. falcataria ------------------------INLFKT----VILRCPKMGLPGAN-----LLCHTAF ::: .*: .. :.*:: .**** 47 Citrus unshiu TLPGLYRVVHGIDVFDPKFNIVSPGADMSIYFPYTEEKRRLKSFHPEIEELLYSDVENKE Gossypium hirsutum TLPGLYRVVHGIDVFDPKFNIVSPGADMEIYFPYTEEKRRLKHFHPEIEDLLYTKVENEE Eucalyptus grandis TLPGLYRVVHGIDVFDPKFNIVSPGADMSIYFSYTEEKLRLKSFHAEIEELLFSDVENKE Populus tremuloides TLPGLYRVVHGIDVFDPKFNIVSPGADESIYFPYTEQKLRLTSFHEEIEELLYSPVENDE Viscum album TLPGLYRVVHGIDVFDPKFNIVSPGADMSIYFPFTEEKRRLTALHPEIEELLFSDVENGE Medicago sativa TLPGLYRVVHGIDVFDPKFNIVSPGADQTIYFPYTETSRRLTSFYPEIEELLYSSVENEE Medicago truncatula TLPGLYRVVHGIDVFDPKFNIVSPGADQTIYFPYTETSRRLTSFYPEIEELLYSSVENEE Visum sativum TLPGLYRVVHGIDVFDPKFNIVSPGADQTIYFPYTETSRRLTSFYPEIEKLLYSTGGNEE Vicia faba TLPGLYRVVHGIDVFDPKFNIVSPGADQTIYFPYTETSRRLTSFYPEIEELLYSTVENEE Glycine max TLPGLYRVVHGIDVFDPKFNIVSPGADQTIYFPHTETSRRLTSFHPEIEELLYSSVENEE Vigna radiata TLPGLYRVVHGIDVFDPKFNIVSPGADQTIYFPHTETSRRLTSFHTEIEELLYSSVENEE A. thaliana TLPGLYRVVHGIDVFDPKFNIVSPGADMSIYFPYTEEKRRLTKFHSEIEELLYSDVENKE P. falcataria TLPVLTRVVHGIDVLDPKLNIVHTAMSCVFTSPSREVSR-------------YQGLGSRS *** * ********:***:*** .. . : . * . : . . Citrus unshiu HLCVLKDRNKPILFTMARLDRVKNLTGLVEWYGKNAKLRELVNLVVVGGDRRKESKDLEE Gossypium hirsutum HLCVLNDRNKPILFTMPRLDRVKNLTGLVEWCGKNPKLRELANLVVVGGDRRKESKDLEE Eucalyptus grandis HLCVLKDRNKPILFTMARLDRVKNLTGLVEWYGKNTRLRELVNLVVVGGDRRKESKDLEE Populus tremuloides HLCVLKDRNKPILFTMARLDRVKNLTGLVEWYGKNTKLREVLNLDVVGGDRRKESKDIEE Viscum album HLCVLKDRKKPIIFSMARLDRVKNITGLVELYGKNARLRELVNLVVVAGDRRKESKDLEE Medicago sativa HICVLKDRNKPIIFTMARLDRVKNITGLVEWYGKNAKLRELVNLVVVAGDRRKESKDLEE Medicago truncatula HICVLKDRNKPIIFTMARLDRVKNITGLVEWYGKNAKLRELVNLVVVAGDRRKESKDLEE Visum sativum HICVLKDRNKPIIFTMARLDRVKNITGLVEWYGKNAKLRELVNLVVVAGDRRKESKDLEE Vicia faba HICVLKDRSKPIIFTMARLDRVKNITGLVEWYGKNAKLRELVNLVVVAGDRRKESKDLEE Glycine max HICVLKDRSKPIIFTMARLDRVKNITGLVEWYGKNAKLRELVNLVVVAGDRRKESKDLEE Vigna radiata HICVLKDRSKPIIFTMARLDRVKNITGLVEWYGKNAKLRELVNLVVVAGDRRKESKDLEE A. thaliana HLCVLKDKKKPILFTMARLDRVKNLSGLVEWYGKNTRLRELANLVVVGGDRRKESKDNEE P. falcataria GRSFRKGKEKKIGSGMKSGTRQMFYS--KEWTGP-------------------------.. :.:.* * * * : * * 48