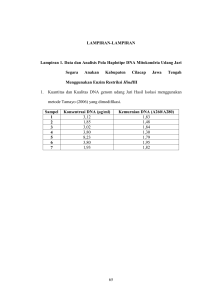
Perbandingan antara Metode SYBR Green dan Metode Hydrolysis
advertisement

UIN SYARIF HIDAYATULLAH JAKARTA Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time PCR SKRIPSI AFIFAH NURUL IZZAH NIM : 1110102000014 FAKULTAS KEDOKTERAN DAN ILMU KESEHATAN PROGRAM STUDI FARMASI JAKARTA OKTOBER 2014 i UIN SYARIF HIDAYATULLAH JAKARTA Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time Polymerase Chain Reaction (PCR) SKRIPSI Diajukan sebagai salah satu syarat untuk memperoleh gelar Sarjana Farmasi AFIFAH NURUL IZZAH NIM : 1110102000014 FAKULTAS KEDOKTERAN DAN ILMU KESEHATAN PROGRAM STUDI FARMASI JAKARTA OKTOBER 2014 HALAMAN PERNYATAAN ORISINALITAS Skripsi ini adalah hasil karya saya sendiri, dan semua sumber baik yang dikutip maupun dirujuk telah saya nyatakan dengan benar Nama : Afifah Nurul Izzah NIM : 1110102000014 Tanda Tangan : Tanggal : 9 Oktober 2014 ii UIN Syarif Hidayatullah Jakarta HALAMAN PERSETUJUAN PEMBIMBING Nama : Afifah Nurul Izzah NIM : 1110102000014 Program Studi : Strata-1 Farmasi Judul : Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time Polymerase Chain Reaction (PCR) Disetujui Oleh: Pembimbing 1 Pembimbing 2 Ofa Suzanti Betha, M.Si., Apt NIP: 19750104200912201 Zilhadia, M.Si., Apt NIP: 197308222008012007 Mengetahui, Ketua Program Studi Farmasi Fakultas Kedokteran dan Ilmu Kesehatan UIN Syarif Hidayatullah Jakarta Drs. Umar Mansur, M.Sc., Apt. iii UIN Syarif Hidayatullah Jakarta HALAMAN PENGESAHAN SKRIPSI Skripsi ini diajukan oleh Nama NIM Program Studi Judul : : : : Afifah Nurul Izzah 1110102000014 Farmasi Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time Polymerase Chain Reaction (PCR) Telah berhasil dipertahankan dihadapan Dewan Penguji dan diterima sebagai bagian persyaratan yang diperlukan untuk memperoleh gelar Sarjana Farmasi pada Program Studi Farmasi Fakultas Kedokteran dan Ilmu Kesehatan Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta DEWAN PENGUJI Pembimbing I : Ofa Suzanti Betha, M.Si., Apt ( ) Pembimbing II : Zilhadia, M.Si., Apt ( ) Penguji I : Yuni Anggraeni, M.Farm., Apt ( ) Penguji II : Lina Elfita, M.Si., Apt ( ) Ditetapkan di Tanggal : Ciputat : 9 Oktober 2014 iv UIN Syarif Hidayatullah Jakarta ABSTRAK Nama Program Studi Judul Skripsi : Afifah Nurul Izzah : Farmasi : Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time Polymerase Chain Reaction (PCR) Pemanfaatan gelatin secara luas menimbulkan kontroversi dan kekhawatiran bagi masyarakat muslim karena pada umumnya gelatin terbuat dari kulit babi dan sapi. Salah satu teknik analisis yang dapat membedakan gelatin sapi dan gelatin babi adalah Real Time PCR. Real Time PCR merupakan metode analisis berbasis DNA yang handal, efektif, dan terpecaya. Dalam analisa kualitatif dan kuantitatif, Real Time PCR membutuhkan pewarna fluoresens. Pewarna fluoresens yang umum digunakan pada Real Time PCR, yaitu SYBR Green dan Hydrolysis Probe. Telah dilakukan perbandingan antara metode SYBR Green dan Hydrolysis Probe dalam analisis DNA gelatin menggunakan Real Time PCR. DNA pada gelatin diekstraksi dan diisolasi menggunakan kit komersial. Isolat DNA gelatin sapi dan DNA gelatin babi didapatkan sebanyak 19,38 ng/μl dan 13,63 ng/μl dengan kemurnian 1,566 dan 1,573. Isolat DNA dilakukan analisis dengan metode SYBR Green menggunakan suhu annealing 65oC untuk primer sapi dan suhu annealing 60oC untuk primer babi. Isolat DNA dianalisis dengan metode Hydrolysis Probe menggunakan suhu annealing 60oC untuk primer babi dan primer sapi. Hasil analisis dari kedua metode menunjukkan bahwa metode Hydrolysis Probe mampu mengidentifikasi DNA pada gelatin secara spesifik dibandingkan menggunakan metode SYBR Green. Kata Kunci: Real Time Polymerase Chain Reaction, Gelatin, SYBR Green, Hydrolysis Probe v UIN Syarif Hidayatullah Jakarta ABSTRACT Name Major Title : Afifah Nurul Izzah : Pharmacy : Comparison of SYBR Green and Hydrolysis Probe methods in Analysis of Porcine Gelatin DNA and Bovine Gelatin DNA Using Real Time Polymerase Chain Reaction (PCR) The wide usage of gelatin in various products led to continous controvercy among Muslim consumers because most of them are derived from porcine and bovine skin. One of the analytical techniques that can differentiate bovine gelatin and porcine gelatin is Real Time PCR. Real Time PCR is a DNA-based technology analysis which is a robust, effective, and reliable. In qualitative and quantitative analysis, there are two kinds methods popular of floresence dye. They are SYBR Green and Hydrolysis Probe. In this study, we have perfomed a comparison between SYBR Green method and Hydrolysis Probe method in analysis Porcine gelatin DNA and Bovine gelatin DNA using Real Time PCR. The DNA is extracted and isolated by commercial kit. Porcine gelatine DNA and Bovine gelatin DNA obtained were 19.38 ng/μl and 13.63 ng/μl with purity were 1,566 and 1,573. Then, DNA isolates were analyzed by SYBR Green methods, with annealing temperature was 65oC using bovine primer and 60oC using porcine primer. While DNA is analyzed by Hydrolysis Probe methods, with annealing temperature was 60oC using both porcine primer annd bovine primer. The result showed that Hydrolysis Probe is able to identify DNA of porcine and bovine gelatin with high specificity than SYBR Green. Keyword: Real Time Polymerase Chain Reaction, Gelatin, SYBR Green, Hydrolysis Probe. vi UIN Syarif Hidayatullah Jakarta KATA PENGANTAR Segala puji dan syukur kehadirat Allah SWT yang senantiasa mencurahkan segala rahmat-Nya kepada kita semua, khususnya penulis dalam menyelesaikan skripsi yang berjudul “Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time PCR”. Shalawat dan salam senantiasa terlimpah kepada junjungan kita Nabi Muhammad SAW, teladan bagi umat manusia dalam menjalani kehidupan. Skripsi ini disusun untuk memenuhi salah satu syarat menempuh ujian akhir guna mendapatkan gelar Sarjana Farmasi (S. Far) pada Program Studi Farmasi, Fakultas Kedokteran dan Ilmu Kesehatan, Universitas Islam Negeri Syarif Hidayatullah Jakarta. Selesainya penelitian dan penyusunan skripsi ini tidak lepas dari bantuan berbagai pihak, maka dalam kesempatan ini perkenankanlah penulis menyampaikan ucapan terimakasih yang tulus dan sebesar-besarnya, khususnya kepada: 1. Bapak Prof. Dr. (hc). Dr. MK.Tadjudin, Sp. And selaku Dekan Fakultas Kedokteran dan Ilmu Kesehatan Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta. 2. Ibu Ofa Suzanti Betha, M.Si., Apt. sebagai Pembimbing I dan Ibu Zilhadia, M.Si., Apt. sebagai Pembimbing II yang sangat baik telah memberikan ilmu, nasehat, waktu, tenaga, pikiran, materi dan dukungan selama penelitian dan penulisan skripsi. 3. Bapak Drs.Umar Mansur, M.Sc., Apt. selaku Ketua Program Studi Farmasi Fakultas Kedokteran dan Ilmu Kesehatan Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta. 4. Kedua orang tua, ayahanda tersayang Ali Mugiono dan ibunda tercinta Fahrida yang selalu ikhlas memberikan dukungan moral, material, nasehat-nasehat, serta lantunan doa yang tiada pernah putus di setiap hembusan nafas beliau hingga penulis dapat menyelesaikan studi di jurusan Farmasi FKIK UIN Syarif Hidayatullah Jakarta. Juga kepada kakakku tersayang Aini Zahra dan adikku tercinta Abdurrahman Marahimin yang selalu memberikan dukungan dan semangat 5. Bapak dan Ibu staf pengajar, serta karyawan yang telah memberikan ilmu pengetahuan, bimbingan dan bantuan selama penulis menempuh pendidikan di farmasi, FKIK UIN Jakarta 6. Teman-teman Andalusia yang telah menjadi kepingan memori yang berharga. Kebersamaan kita di dalam suka dan duka akan selalu terkenang didalam hati vii UIN Syarif Hidayatullah Jakarta 7. Kak Rahmadi, Kak Liken, Kak Eris, Kak Lisna, Kak Rani, Kak Tiwi, Kak Lilis, dan Kak Ai yang sangat banyak membantu penulis melakukan penelitian di laboratorium. 8. Teman-teman seperjuangan tim PCR: Yanti dan Kak Sulaiman yang selalu meluangkan waktunya untuk bekerja sama, berdiskusi, memberikan masukan, membantu penulis dalam melakukan peneltian, serta memberikan dukungan doa dan semangat kepada penulis dalam penyelesaian skripsi ini. Terimakasih atas kebersamaan yang indah ini. 9. Tim Roche Indonesia: Pak Deka, Pak Yos, dan Mbak Helen yang telah memberikan masukan dan bantuan selama penulis melakukan penelitian. 10. Sahabat tersayang “ngocol”: Dias, Zakiya, Ipho, Vicka, Diah, Amel, Dita, dan Desi yang senantiasa mewarnai kehidupan selama dikampus. Terima kasih atas dukungan, kasih sayang, perhatian, doa dan persahabatan yang indah ini. 11. Farida, Yusna, Fahrur, Yuni, Deisy, Desti, Hanny, Liana, Delvina, Mayta, Athiyah, Fathia, Dwikky, Hadi, Fikry, yang menginspirasi. Terimakasih atas dukungan dan semangatnya. 12. Dan kepada semua pihak yang telah membantu penulis selama ini yang tidak dapat disebutkan namanya satu persatu. Semoga semua bantuan yang telah diberikan mendapatkan balasan dari Allah SWT. Penulis menyadari bahwa penulisan skripsi ini masih jauh dari kesempurnaan. Oleh karena itu, saran dan kritik yang bersifat membangun akan penulis nantikan. Dan semoga skripsi ini bisa bermanfaat bagi pengembangan ilmu pengetahuan. Jakarta, 14 Oktober 2014 Penulis viii UIN Syarif Hidayatullah Jakarta HALAMAN PERNYATAAN PERSETUJUAN PUBLIKASI TUGAS AKHIR UNTUK KEPENTINGAN AKADEMIS Sebagai sivitas akademika Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta, saya yang bertanda tangan di bawah ini: Nama : Afifah Nurul Izzah NIM : 1110102000014 Program Studi : Strata-1 Farmasi Fakultas : Kedokteran dan Ilmu Kesehatan Jenis Karya : Skripsi demi pengembangan ilmu pengetahuan, saya menyetujui skripsi/karya ilmiah saya, dengan judul : Perbandingan antara Metode SYBR Green dan Metode Hydrolysis Probe dalam Analisis DNA Gelatin Sapi dan DNA Gelatin Babi dengan Menggunakan Real Time PCR untuk dipublikasikan atau ditampilkan di internet atau media lain yaitu Digital Library Perpustakaan Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta untuk kepentingan akademik sebatas sesuai dengan Undang-Undang Hak Cipta. Demikian pernyataan publikasi karya ilmiah ini saya buat dengan sebenarnya. Dibuat di : Jakarta Pada tanggal : Oktober 2014 Yang menyatakan, Afifah Nurul Izzah ix UIN Syarif Hidayatullah Jakarta DAFTAR ISI HALAMAN JUDUL................................................................................................. HALAMAN PERNYATAAN ORISINALITAS..................................................... LEMBAR PERSETUJUAN PEMBIMBING......................................................... HALAMAN PENGESAHAN SKRIPSI.................................................................. ABSTRAK.................................................................................................................. ABSTRACT............................................................................................................... KATA PENGANTAR............................................................................................... HALAMAN PERNYATAAN PERSETUJUAN PUBLIKASI.............................. DAFTAR ISI.............................................................................................................. DAFTAR GAMBAR................................................................................................. DAFTAR TABEL...................................................................................................... DAFTAR LAMPIRAN............................................................................................. DAFTAR SINGKATAN........................................................................................... DAFTAR ISTILAH................................................................................................... BAB 1 PENDAHULUAN.......................................................................................... 1.1 Latar Belakang......................................................................................... 1.2 Rumusan Masalah.................................................................................... 1.4 Tujuan Penelitian..................................................................................... 1.5 Manfaat Penelitian................................................................................... BAB 2 TINJAUAN PUSTAKA................................................................................ 2.1 Gelatin...................................................................................................... 2.1.1 Sifat Fisika Kimia Gelatin............................................................. 2.1.2 Struktur Kimia Gelatin.................................................................. 2.1.3 Aplikasi dan Pemanfaatan gelatin................................................. 2.2 Asam Nukleat.......................................................................................... 2.2.1 DNA............................................................................................... 2.2.3.1 Struktur DNA................................................................... 2.2.3.2 Sifat Fisika DNA.............................................................. 2.2.2 DNA Mitokondria.......................................................................... 2.3 Ekstraksi dan Isolasi DNA....................................................................... 2.4 Elektroforesis Gel Agarosa...................................................................... 2.5 PCR.......................................................................................................... 2.5.1 Pengertian PCR.............................................................................. 2.5.2 Tahapan PCR................................................................................. 2.5.3 Komponen PCR............................................................................. 2.6 Real-Time PCR........................................................................................ 2.6.1 Pewarna Fluoresens....................................................................... x i ii iii iv v vi vii ix x xii xiii xiv xv xvi 1 1 3 3 3 4 4 5 7 7 8 9 9 11 12 14 16 18 18 19 22 22 23 UIN Syarif Hidayatullah Jakarta BAB 3 Metodologi Penelitian................................................................................... 3.1 Tempat dan Waktu Penelitian.................................................................. 3.1.1 Tempat........................................................................................... 3.1.2 Waktu............................................................................................. 3.2 Alat dan Bahan........................................................................................ 3.2.1 Alat................................................................................................ 3.2.2 Bahan............................................................................................. 3.3 Prosedur Penelitian.................................................................................. 3.3.1 Ekstraksi dan Isolasi DNA............................................................ 3.3.2 Analisis Isolat DNA....................................................................... 3.3.2.1 Analisis Isolat DNA dengan Elektroforesis Agarosa....... 3.3.2.2 Analisis Isolat DNA dengan Spektrofotometri UV.......... 3.3.3 Uji Spesifisitas Primer dan Probe dengan Database NCBI.......... 3.3.4. Amplifikasi DNA Menggunakan Real Time PCR........................ 3.3.5.1 Amplifikasi DNA dengan Metode SYBR Green............... 3.3.5.2 Amplifikasi DNA dengan Metode Hydrolysis Probe....... BAB 4 HASIL DAN PEMBAHASAN..................................................................... 4.1 Hasil Analisis Isolat DNA....................................................................... 4.1.1 Hasil Analisis Isolat DNA dengan Elektroforesis Agarosa........... 4.1.2 Hasil Analisis Isolat DNA dengan Spektrofotometri UV............. 4.2. Hasil Uji Spesifisitas Primer dan Probe dengan Database NCBI.......... 4.3 Hasil Amplifikasi DNA Gelatin Sapi dan Babi Menggunakan Real Time Polymerase Chain Reaction (PCR)................................................ 4.3.1 Hasil Amplifikasi DNA Gelatin Sapi dan Babi Menggunakan Real Time PCR dengan Metode SYBR Green................................ 4.3.2 Hasil Ampifikasi DNA Gelatin Sapi dan Babi Menggunakan Real Time PCR dengan Metode Hydrolysis Probe........................ BAB 5 KESIMPULAN DAN SARAN..................................................................... 5.1 Kesimpulan.............................................................................................. 5.2 Saran........................................................................................................ DAFTAR PUSTAKA................................................................................................ LAMPIRAN............................................................................................................... xi 26 26 26 26 26 26 26 27 27 29 29 30 31 31 31 34 36 36 36 37 38 40 40 46 50 50 50 51 57 UIN Syarif Hidayatullah Jakarta DAFTAR GAMBAR Gambar 2.1 Gambar 2.2 Gambar 2.3 Gambar 2.4 Gambar 2.5 Gambar 2.6 Gambar 2.7 Gambar 2.8 Gambar 2.9 Gambar 2.10 Gambar 2.11 Gambar 2.12 Gambar 2.13 Gambar 2.14 Gambar 4.1 Gambar 4.2 Gambar 4.3 Gambar 4.4 Gambar 4.5 Gambar 4.6 Gambar 4.7 Gambar 4.8 Gambar 4.9 Metode Ekstraksi Gelatin dari Jaringan yang Mengandung Kolagen........................................................................................... Struktur dan Asam Amino Penyusun Kolagen dan Gelatin............ Komponen Asam Amino Penyusun................................................ Struktur Nukleotida......................................................................... Ikatan Fosfodiester pada DNA dan RNA....................................... Struktur DNA.................................................................................. Struktur Mitokondria...................................................................... Struktur DNA Mitokondria............................................................. Presipitasi DNA Setelah Penambahan Etanol Absolut................... Interkalasi Etidium Bromida pada DNA......................................... Tahapan Proses PCR....................................................................... Pelipatgandaan Molekul DNA Target............................................ Bentuk Kurva Real-Time PCR........................................................ Mekanisme Hydrolysis Probe dan SYBR Green............................. Hasil Elektroforesis Isolat DNA..................................................... Hasil Uji Spesifisitas Primer Babi pada Database NCBI............... Hasil Uji Spesifisitas Primer Sapi pada Database NCBI................ Kurva Amplifikasi dengan Metode SYBR Green Menggunakan Primer Sapi...................................................................................... Melting Peaks Hasil Amplifikasi dengan Metode SYBR Green Menggunakan Primer Sapi.............................................................. Kurva Amplifikasi dengan Metode SYBR Green Menggunakan Primer Babi..................................................................................... Melting Peaks Hasil Amplifikasi dengan Metode SYBR Green Menggunakan Primer Babi............................................................. Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer-Probe Sapi................................................... Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer-Probe Babi................................................... xii 5 6 7 9 10 11 12 13 15 18 19 21 23 25 37 39 39 41 42 44 45 47 48 UIN Syarif Hidayatullah Jakarta DAFTAR TABEL Tabel 1 Tabel 2 Tabel 3 Tabel 4 Tabel 5 Tabel 6 Tabel 7 Tabel 8 Kisaran Umum Konsentrasi Agarosa................................................... Urutan Basa Primer-Probe.................................................................... Pengaturan Program Amplifikasi dengan Metode SYBR Green.......... Pengaturan Program Amplifikasi dengan Metode Hydrolysis Probe.. Konsentrasi dan kemurnian DNA hasil isolasi..................................... Campuran Reaksi SYBR Green Mastermix.......................................... Campuran Reaksi Hydrolysis Probe Mastermix.................................. Spesifikasi Kit Ekstraksi Komersial..................................................... xiii 17 27 32 34 38 60 60 62 UIN Syarif Hidayatullah Jakarta DAFTAR LAMPIRAN Lampiran 1 Lampiran 2 Lampiran 3 Lampiran 4 Lampiran 5 Lampiran 6 Lampiran 7 Lampiran 8 Lampiran 9 Lampiran 10 Lampiran 11 Lampiran 12 Lampiran 13 Lampiran 14 Lampiran 15 Lampiran 16 Kerangka Penelitian................................................................... Perhitungan Pembuatan Larutan Primer dan Probe................... Perhitungan Tm (Melting Temperature) Primer........................ Campuran Reaksi Mastermix untuk Amplifikasi DNA............. Hasil Optimasi Suhu Annealing dengan Metode Gradien PCR. Spesifikasi Kit Ekstraksi dan Isolasi DNA................................ Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 60oC.......................................................................... Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 62oC.......................................................................... Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 64oC.......................................................................... Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 65oC.......................................................................... Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Babi dengan Waktu Annealing 10 detik dan Waktu Extension 7 detik...................... Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Babi dengan Waktu Annealing 20 detik dan Waktu Extension 30 detik.................... Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Babi dengan Waktu Annealing 20 detik dan Waktu Extension 25 detik.................... Hasil Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer Sapi......................................................... Hasil Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer Babi......................................................... Gambar Alat yang Digunakan dalam Penelitian........................ xiv 57 58 60 61 62 63 64 65 66 67 68 69 70 71 71 72 UIN Syarif Hidayatullah Jakarta DAFTAR SINGKATAN BHQ-1 BLAST CP cyt b dATP dCTP dGTP dNTP dTTP DNA ELISA FAM FTIR LCMS mtDNA NCBI NTC PCR pb qPCR RE RNA SDS TAE Tm Ta : Black Hole Quencher-1 : Basic Local Aligment Search Tool : Crossing Point : Cytochrome b : Deoxyadenosine Triphosphate : Deoxycytidine Triphosphate : Deoxyguanosine Triphosphate : Deoxyribonucleaside Triphosphate : Deoxythymidine Triphosphate : Deoxyribonucleic Acid : Enzyme-linked Immunosorbent Assay : Fluorescein Amidite : Forier Transform Infrared Spectroscopy : Liquid Chromatography Mass-Spectrophotometry : mitochondria DNA : National Center for Biotechnology Information : No Template Control : Polymerase Chain Reaction : Pasang Basa : Quantitative Polymerase Chain Reaction : Retikulum Endoplasma : Ribonucleic Acid : Sodium Dodesil Sulfat : Tris-Acetate-EDTA : Temperature Melting : Temperature Annealing xv UIN Syarif Hidayatullah Jakarta DAFTAR ISTILAH BLAST Blastn CP Garis Treshold Formasi Hairpin Melting curve Mis-priming NCBI NTC Primer-Dimer Query Tm : Basic Local Aligment Search Tool merupakan program untuk menganalisis kesejajaran sekuen query (DNA atau protein) dengan sekuen DNA atau protein pada database NCBI. : Nucleotide Basic Local Aligment Search Tool merupakan salah satu variasi dari program BLAST untuk menganalisis kesejajaran nukleotida query dengan nukleotida pada database di NCBI. : Crossing Point merupakan siklus yang menunjukkan sinyal fluoresensi dari akumulasi amplikon telah melewati garis threshold. : Garis penanda siklus awal dari reaksi PCR yaitu saat sinyal fluoresensi berada pada tingkat terendah ( siklus 3-15). : Terbentuknya struktur loop/hairpin pada primer yang disebabkan oleh interaksi intramolekular yang dapat memicu terbentuknya amplifikasi yang nonspesifik. : Analisis data pada Real-time PCR digunakan untuk menguji spesifisitas amplikon yanng terbentuk. : Penempelan primer di luar sekuen target sehingga membentuk produk amplifikasi yang nonspesifik. : National Center for Biotechnology Information merupakan suatu institusi milik United States National Library of Medicine yang berperan sebagai sumber informasi perkembangan biologi molekular. Situs NCBI berisi database meliputi gene bank mengenai sekuens DNA atau protein serta memuat publikasi ilmiah. : No Template Control merupakan kontrol negatif karena well tidak dimasukkan DNA. : Berikatannya suatu primer dengan primer sejenis atau dengan primer lainnya sehingga membentuk produk amplifikasi yang nonspesifik. : Sekuen yang dimasukkan ke dalam program BLAST untuk diketahui kesejajarannya. : Temperature melting atau suhu melting adalah suhu saat 50% bagian DNA telah terbuka menjadi untai tunggal. xvi UIN Syarif Hidayatullah Jakarta 1 BAB 1 PENDAHULUAN 1.1 LATAR BELAKANG Gelatin adalah polipeptida larut air yang merupakan hasil hidrolisis parsial kolagen dari kulit, tulang dan tulang rawan hewan (Zhang et al., 2008). Gelatin memiliki sifat yang unik dan berbagai fungsi yaitu sebagai zat pembentuk gel, zat pengental, zat pembentuk film, zat pengemulsi, dan zat pensuspensi (Rowe, Sheskey & Quinn, 2009). Oleh karena itu gelatin digunakan secara luas dalam berbagai sektor industri, misalnya sektor industri farmasi, makanan, fotografi, kosmetik, dan produk kedokteran. Dalam industri makanan, gelatin dapat ditemukan dalam produk ice cream, jelly, dan marshmallow. Pada industri farmasi, gelatin digunakan dalam tablet, cangkang kapsul keras dan lunak, tablet salut gula, enkapsulasi vitamin, dan pensubtitusi plasma darah (Nhari, Ismail & Che Man, 2012). Sumber gelatin yang beredar di pasaran berasal dari babi, sapi, tulang ikan, kerang, rumput laut, dan lain-lain (Hinterwaldner, 1997; R.T. Jones, 2004). Produksi gelatin di dunia pada tahun 2007 dilaporkan bahwa 46% berasal dari kulit babi; 29,4% dari kulit sapi; 23,1% dari campuran tulang babi dan sapi; dan 1,5% dari tulang ikan, kerang, dan lain-lain (Guillen et al., 2009). Pembuatan gelatin yang bersumber dari babi dan sapi lebih banyak diminati karena gelatin yang dihasilkan memberikan kualitas yang lebih baik dibandingkan dengan sumber lainnya seperti ikan (Hinterwaldner, 1997). Gelatin umumnya masih diimpor dari negara-negara nonmuslim sehingga menimbulkan kontroversi kehalalannya. Pemanfaatan gelatin dalam berbagai bidang industri dan peningkatan kesadaran masyarakat muslim terhadap penggunaan produk halal, menimbulkan konsekwensi perlunya perlindungan konsumen dengan pemberian jaminan kehalalan sumber gelatin (Jamaludin et al., 2012). Hal ini membutuhkan metode identifikasi gelatin sapi dan gelatin babi yang handal, tepercaya, efektif, dan efisien. 1 UIN Syarif Hidayatullah Jakarta 2 Keberadaan gelatin babi dan sapi pada produk pangan, kosmetik, dan obat-obatan sangat sukar untuk diidentifikasi karena memiliki sifat fisik dan kimia yang sangat mirip (Nemati et al., 2004). Oleh karena itu, perlu diupayakan metode yang akurat untuk identifikasi gelatin sapi dan gelatin babi. Metode identifikasi gelatin sapi dan gelatin babi telah banyak dilakukan dan dikembangkan. Di antaranya dengan teknik presipitasi kimia (Hikada dan Liu, 2003), FTIR (Hashim et al., 2010), LCMS (Zhang et al., 2008), ELISA (Venien dan Levieux, 2005), dan analisis berbasis DNA dengan Real Time PCR (Cai et al., 2011; Demirhan et al., 2011). DNA merupakan molekul yang memiliki kestabilan tinggi, sehingga analisis berbasis DNA dengan Real Time PCR dapat dilakukan pada produk olahan. Selain itu, metode ini dapat mendeteksi campuran gelatin babi dan gelatin sapi dengan level kontaminasi 1% (Cai et al., 2011; Demirhan et al., 2011). Dengan metode ini, kita dapat mengamati secara langsung hasil amplifikasi atau perbanyakan DNA fragmen. Metode ini juga dapat menentukan konsentrasi DNA yang terdapat pada sampel dengan mengukur peningkatan pewarna fluoresens yang berpendar ketika terikat dengan untai ganda DNA (Arya et al., 2005). Dengan demikian, analisis kehalalan gelatin dengan Real-Time PCR merupakan metode yang sederhana, handal dan terpercaya (Cai et al., 2011). Dalam mendeteksi dan identifikasi DNA pada sampel, Real Time PCR membutuhkan pewarna fluoresens. Pewarna fluoresens yang umum digunakan dalam Real-Time PCR adalah SYBR Green dan Hydrolysis Probe. Pewarna fluoresens SYBR Green akan berpendar ketika terinterkalasi dengan untai ganda DNA. Sedangkan fluoresens dari Hydrolysis Probe akan berpendar ketika Probe, yang merupakan dua pewarna fluoresens (terdiri atas Reporter dan Quencher), secara fisik terpisah melalui hidrolisis oleh aktivitas nuclease (Arya et al., 2005). Dibandingkan Hydrolysis Probe, metode SYBR Green lebih ekonomis dan mudah dalam penanganan. SYBR Green tidak memerlukan Probe yang membutuhkan banyak biaya untuk mensintesisnya. Namun demikian, SYBR Green sangat tergantung dari spesifisitas primer karena dapat menyebabkan terjadinya mis-priming dan primer-dimer UIN Syarif Hidayatullah Jakarta 3 sehingga menyebabkan terbentuknya produk amplifikasi yang nonspesifik. Sedangkan Hydrolysis Probe lebih mampu mencegah terjadinya produk amplifikasi yang nonspesifik (Arya et al., 2005). Pada penelitian ini akan dilakukan perbandingan antara metode SYBR Green dan metode Hydrolysis Probe dalam analisis DNA gelatin sapi dan DNA gelatin babi dengan menggunakan Real Time PCR. 1.2 RUMUSAN MASALAH a. Belum diketahuinya efektivitas metode SYBR Green dan Hydrolysis Probe dalam menganalisis DNA gelatin sapi dan gelatin babi menggunakan Real Time PCR. b. Belum diketahuinya apakah metode SYBR Green dan metode Hydrolysis Probe secara spesifik dapat mengamplifikasi DNA gelatin sapi atau gelatin babi. 1.3 TUJUAN PENELITIAN Mengetahui perbandingan hasil analisis DNA gelatin sapi dan DNA gelatin babi dengan metode SYBR Green dan Hydrolisis Probe. 1.4 MANFAAT PENELITIAN Hasil dari penelitian ini diharapkan dapat memberikan informasi perbandingan metode SYBR Green dan metode Hydrolysis Probe dalam analisis DNA gelatin sapi dan DNA gelatin babi, khususnya pada produk makanan, obat-obatan, ataupun kosmetik yang mengandung gelatin. UIN Syarif Hidayatullah Jakarta 4 BAB 2 TINJAUAN PUSTAKA 2.1 Gelatin Kata gelatin berawal dari bahasa latin “gelatus” yang berarti kaku atau beku. Gelatin adalah polipeptida hasil hidrolisis parsial kolagen yang diperoleh dari jaringan ikat, kulit, atau tulang rawan sapi dan babi. Proses hidrolisis ini dapat dikatalisis dengan penambahan asam kuat atau basa kuat (Rabadiya, 2013). Ketika kolagen diperlakukan dengan asam atau basa dan diikuti dengan panas, struktur fibrosa kolagen dipecah ireversibel menghasilkan gelatin. Gelatin dihasilkan melalui cross-linking (ikatan silang) diantara rantai polipetida pada kolagen dengan disertai sejumlah perusakan pada rantai ikatan peptida (Zhou dan Regenstein, 2004). Selain berasal dari hewan mamalia, gelatin juga dapat diperoleh dari limbah tulang ikan. Pembuatan gelatin merupakan upaya untuk mendayagunakan limbah tulang yang biasanya tidak terpakai dan dibuang dirumah pemotongan hewan (Balti et al., 2010). Berdasarkan proses pembuatannya, gelatin dapat dikategorikan dalam dua prinsip dasar (Jannah, 2008), yaitu: a. Gelatin Tipe A (Acid) Gelatin ini dihasilkan dengan proses asam dari bahan baku kolagen. Tipe A umumnya diperoleh dari kulit babi, tetapi ada juga beberapa pabrik yang menggunakan bahan dasar tulang babi. Kulit dari babi muda tidak memerlukan penanganan alkalis yang intensif, karena jaringan ikatnya tidak terlalu kuat. Oleh karena itu, pada proses ini kulit muda cukup direndam dengan asam klorida (HCl) encer selama beberapa hari, kemudian dinetralkan dan dicuci beberapa kali dengan aquadest agar garam yang terbentuk hilang. b. Gelatin Tipe B (Base) Gelatin tipe tipe ini dihasilkan melalui proses basa atau alkali. Bahan dasar biasanya berasal dari kulit tua atau keras maupun tulang 4 UIN Syarif Hidayatullah Jakarta 5 ruminasia. Proses pembuatannya yaitu bahan mula-mula direndam dalam larutan kalsium hidroksida, sehingga ikatan jaringan kolagen akan mengembang dan terurai. Kemudian bahan dinetralkan dengan asam dan dicuci dengan aquadest untuk menghilangkan garam yang terbentuk. Gambar 2.1 Metode ekstrasi gelatin dari jaringan yang mengandung kolagen (Sumber: Ikada, 2002) 2.1.1 Sifat Fisika Kimia Gelatin Fraksi protein pada gelatin hampir seluruhnya terdiri atas berbagai macam asam amino yang bergabung melalui ikatan amida dan membentuk polimer yang linear. Gelatin memiliki berat molekul yang bervariasi yaitu 20 kDa sampai 200 kDa. Kadar protein pada gelatin tinggi yaitu pada gelatin kering (dengan kadar air 8 – 12%) mengandung protein sekitar 84 86% (Carr et al.,1995). Gelatin komersial secara luas diproduksi di berbagai belahan dunia. Di eropa, gelatin untuk pangan tersedia dalam bentuk lembaran tipis (flake), sedangkan di Amerika Serikat gelatin dapat diperoleh dalam bentuk granul atau serbuk. Gelatin tidak memiliki rasa dan bau. Warna serbuk granul dan flakes pada gelatin yaitu putih agak kuning pucat. Pada bentuk lembaran, gelatin berwarna kuning pucat transparan (Rowe, Sheskey & Quinn, 2009). UIN Syarif Hidayatullah Jakarta 6 Gelatin praktis tidak larut dalam pelarut organik seperti aseton, kloroform, etanol (95%), eter dan metanol. Zat ini larut dalam gliserin, larutan asam dan basa. Namun pada larutan asam kuat atau basa kuat, gelatin akan mengalami presipitasi. Gelatin larut dalam air hangat dan apabila didinginkan dibawah suhu 30oC, larutan koloid ini akan membetuk gel dengan sifat tiksotropik dan reversibel menjadi cair kembali apabila dipanaskan. pH larutan atau gel gelatin akan berbeda tergantung tipenya yaitu tipe A pH 3,8 – 5,5; sedangkan tipe B pH 5,0 – 7,5. Gambar 2.2. Struktur dan Asam Amino Penyusun Kolagen dan Gelatin (Sumber: http://www.gelatin.in) Bloom (kekuatan gel) tergantung pada konsentrasi gelatin di dalam air, berat molekul gelatin, dan pH gel. Bloom akan meningkat apabila konsentrasi gelatin tinggi, berat molekul gelatin tinggi, dan pH gel yang mendekati netral. Gelatin memiliki kekuatan Bloom yang bervariasi, yaitu 50 hingga 300. Industri gelatin umumnya mencampur bahan baku untuk mendapatkan kekuatan gel yang diinginkan (Rabadiya, 2013). 2.1.2 Struktur Kimia Gelatin Struktur gelatin terdiri dari 300 sampai 4.000 rantai asam amino yang dihubungkan oleh ikatan peptida. Gelatin mengandung sejumlah 18 asam UIN Syarif Hidayatullah Jakarta 7 amino spesifik yang berbeda dan bekerja sama berurutan untuk membentuk rantai polipeptida dengan 1000 asam amino setiap rantai. Sebanyak 3 rantai polipeptida terbentuk bekerja sama sebagai spiral sisi kiri untuk memberi struktur sekunder. Dalam struktur tersier, spiral menggulung dan melipat sendiri pada sisi kanan (triplehelix). Ini membentuk molekul berbentuk tangkai yang disebut protofibril. Rantai asam amino dominan yang terdapat dalam gelatin adalah glysin (26-34%), prolin (10-18%) dan hidroksiprolin (7-15%). Jenis asam amino lain terdapat dalam gelatin, yaitu alanin (8-11%), arginin (8-9%), asam aspartat (6-7%), dan asam glutamat (10-12%). Meskipun demikian, gelatin bukan merupakan protein yang lengkap. Hal ini karena gelatin tidak mengandung asam amino triptofan dan hanya sedikit mengandung asam amino isoleusin, treonin, metionin, sistein, dan sistin (Rabadiya, 2013). Gambar 2.3. Komponen Asam Amino Penyusun Gelatin (Sumber: http://www.pbgelatins.com) 2.1.3 Aplikasi dan pemanfaatan gelatin Gelatin merupakan bahan pangan yang telah lama digunakan secara luas pada produk pangan dan farmasi. Gelatin tidak memiliki rasa dan bau, akan tetapi gelatin memiliki sifat gel yang unik, sehingga dapat digunakan sebagai penstabil, pengikat, dan pengemulsi dalam industri farmasi. Sebagai bahan pangan, gelatin memiliki sifat yang unik sebagai UIN Syarif Hidayatullah Jakarta 8 penstabil busa, yaitu dibutuhkan pada saat pembuatan permen. Bahan pangan yang menggunakan gelatin antara lain cokelat, susu, permen, jelly, roti, marshmallow, pada produk daging sebagai pengikat air, dan lain-lain. Sedangkan penggunaan gelatin pada industri farmasi dan kedokteran meliputi cangkang kapsul, salut tablet, zat pengemulsi dan penstabil dalam pembuatan krim, mikroenkapsulasi, dan plasma pensubtitusi darah. Tidak hanya pada industri pangan dan farmasi, gelatin juga dapat bermanfaat pada bidang fotografi yaitu sebagai pelapis zat warna film. Dari data SKW biosystem, penggunaan gelatin dalam industri nonpangan sejumlah 100.000 ton, pada industri pembuatan film foto sebanyak 27.000 ton, untuk kapsul lunak sebanyak 22.600 ton, untuk produksi cangkang capsul keras sebanyak 20.200 ton, serta dalam dunia farmasi dan teknis sebanyak 12.000 ton dan 6.000 ton. Penggunaan gelatin dalam industri pangan adalah sebesar 154.000 ton, dimana penggunaan terbesar adalah industri konfeksioneri yaitu sebesar 68.000 ton, dan produk jelli sebanyak 36.000 ton. Pada industri daging dan susu memiliki jumlah penggunaan gelatin yang sama yaitu 16.000 ton dan pada makanan fungsional (food supplement) memeliki kontribusi penggunaan gelatin yang sama yaitu sebesar 4.000 ton (LPPOM MUI, 2008). 2.2. Asam Nukleat Asam nukleat dan protein merupakan senyawa polimer utama yang terdapat pada sel. Asam nukleat berfungsi menyimpan dan mentransmisikan informasi genetik dalam sel (Koolman & Roehm, 2005). Sel mempunyai dua jenis molekul asam nukleat yaitu DNA (asam deoksiribonukleat) dan RNA (asam ribonukleat). DNA menyimpan informasi genetik yang spesifik untuk setiap individu dan spesies tertentu, yang akan diwariskan ke generasi berikutnya (Gaffar, 2007). DNA ditemukan di dalam nukleus pada sel eukariotik, dan ditemukan di sitoplasma atau nukleoid pada sel prokariotik. Molekul RNA disintesis dari DNA template dan berperan dalam pembentukan protein didalam sitoplasma (Stansfield et al., 2003). UIN Syarif Hidayatullah Jakarta 9 2.2.1 DNA 2.2.1.1 Struktur DNA DNA dan RNA merupakan polimer linier (polinukleotida) yang tersusun dari subunit nukleotida. Nukleotida merupakan ester dari asam fosfat dan nukleosida. Komponen penyusun nukleotida terdiri dari tiga jenis molekul, yaitu gula pentosa (deoksiribosa pada DNA atau ribosa pada RNA), basa nitrogen heterosiklik, dan gugus fosfat yang bermuatan negatif (Gaffar, 2007). Gambar 2.4. Struktur Nukleotida (Sumber: Gaffar, 2007) Basa yang ditemukan pada nukleotida adalah basa purin (adenin = A, guanin = G) dan basa pirimidin (cytosin = C, tymin = T, dan urasil = U). Monomer nukleotida mempunyai gugus hidroksil pada posisi karbon 3’, gugus fosfat pada posisi karbon 5’ dan basa pada posisi karbon 1’ molekul gula. Nukleotida satu dengan yang lainnya berikatan membentuk polinukleotida melalui ikatan fosfodiester antara gugus 5’fosfat dengan gugus 3’hidroksil. Sehingga tulang punggung molekul DNA dan RNA terdiri dari gugus fosfat dan pentosa secara bergantian (Stansfield et al., 2003). Struktur DNA mirip dengan struktur RNA. Perbedaan diantara keduanya terdapat pada jenis gula dan basa pada monomernya serta jumlah untai penyusunnya. DNA tidak terdapat gugus hidroksil pada posisi karbon 2’ dari molekul gula (2-deoksiribosa) sementara pada RNA molekul gulanya adalah ribosa. Basa nitrogen yang terdapat pada DNA adalah adenin, guanin, sitosin dan timin, sedangkan pada RNA jenis UIN Syarif Hidayatullah Jakarta 10 basanya adalah adenin, sitosin, guanin dan urasil. RNA merupakan polinukleotida yang membentuk satu rantai/untai sedangkan DNA merupakan polinukleotida yang membentuk 2 untai (heliks ganda) yang memutar ke kanan. Kedua rantai polinukleotida memutar pada sumbu yang sama dan bergabung satu dengan yang lainnya melalui ikatan hidrogen antara basa-basanya (Gaffar, 2007). Gambar 2.5. Ikatan Fosfodiester pada DNA dan RNA (Gaffar, 2007) UIN Syarif Hidayatullah Jakarta 11 Pada DNA, basa guanin berpasangan dengan basa sitosin membentuk tiga ikatan hidrogen, sedangkan basa adenin berpasangan dengan basa tymin membentuk dua ikatan hidrogen. Sehingga dalam molekul DNA jumlah basa G akan selalu sama dengan jumlah basa C, sedangkan jumlah basa A sama dengan jumlah basa T. Kemudian jumlah basa purin akan sama dengan jumlah basa pirimidin (Purves et al., 2004). Gambar 2.6. Struktur DNA (Sumber: Purves et al., 2004) Untai ganda DNA saling berkomplementasi melalui basa penyusunnya dengan arah antiparalel (berlawanan 5’→ 3’ dan 3’→5’), ujung yang mengandung gugus fosfat bebas disebut ujung 5’ sedangkan pada ujung lainnya yang mengandung gugus hidroksil bebas disebut ujung 3’. Kedua untai tersebut saling melilit satu sama lain membentuk struktur heliks ganda. Gugus fosfat dan gula yang tersusun bergantian menjadi tulang punggung (backbone) molekul DNA sementara pada bagian dalam terdapat basa yang melekat pada molekul gula (Gaffar, 2007). Struktur helix ganda membentuk pilinan berjarak 3.4 nm dengan sepuluh pasang basa setiap pilinan dan memiliki lebar sekitar 2 nm (Stansfield, 2003). 2.2.1.2 Sifat Fisika DNA Untai heliks DNA dapat memisah menjadi struktur untai tunggal dengan adanya pemanasan dengan suhu tinggi (> 90oC), peristiwa ini UIN Syarif Hidayatullah Jakarta 12 sering dikenal dengan denaturasi. Denaturasi DNA bersifat reversible. Proses pembentukan kembali struktur untai ganda dari keadaan terdenaturasi disebut renaturasi. Proses renaturasi dapat berjalan jika suhu mendekati suhu subdenaturasi (mendekati 60oC). DNA menyerap sinar UV pada panjang gelombang 260 nm (Yuwono, 2009). 2.2.2 DNA Mitokondria Mitokondria merupakan organel sel yang berfungsi sebagai penghasil energi dalam bentuk ATP yang akan dipergunakan untuk aktivitas seluruh sel-sel tubuh manusia. Mitokondria memiliki diameter sebesar 1-2 μm (Passarge, 2007). Sel eukariotik rata-rata terdiri dari 103-104 salinan mitokondria yaitu mencapai 25% volume sel. Mitokondria dikelilingi oleh dua membran yaitu membran dalam dan membran luar. Membran bagian dalam membentuk lipatan-lipatan yang disebut kristae dimana terdapat enzim-enzim oksidase. Membran dalam juga memiliki permukaan yang besar yang mengelilingi ruang matriks. Matriks ini mengandung DNA, RNA, ribosom, dan berbagai enzim yang berperan dalam oksidasi zat-zat makanan. DNA yang terdapat pada mitokondria disebut DNA mitokondria atau disingkat menjadi mtDNA (Koolman dan Roehm, 2005). Gambar 2.7. Sruktur Mitokondria (Koolman dan Roehm, 2005). Umumnya, mitokondria terdiri dari 2-10 molekul DNA. mtDNA merupakan DNA rantai ganda yang berbentuk sirkuler dengan ukuran genom mitokondria hewan berkisar 14000-39000 pasang basa. Ukuran UIN Syarif Hidayatullah Jakarta 13 mtDNA relatif sangat kecil dibandingkan dengan ukuran DNA inti. DNA mitokondria hewan secara umum memiliki jumlah dan jenis gen yang sama yaitu 13 daerah yang mengkode protein (URF1, URF2, URF3, URF4, URF5, URF6, URFA6L, URF4L, Cytochrome Oxidase unit I, Cytochrome Oxidase unit II, Cytochrome Oxidase unit III, Cytochrome b dan ATPase 6); 2 gen pengkode rRNA yaitu 12S rRNA dan 16S rRNA; dan 22 gen pengkode tRNA (Sharma et al., 2005). Gambar 2.8. Struktur DNA Mitokondria (www.bmb.leeds.ac.uk) Karakteristik mtDNA dapat dijadikan alat yang signifikan untuk keperluan analisis. mtDNA mempunyai jumlah copy yang tinggi, meskipun di dalam sel yang tidak mengandung inti. Jumlah copy per sel yaitu sekitar 1000-10000 sehingga mtDNA dapat digunakan untuk analisis sampel dengan jumlah DNA yang sangat terbatas, atau DNA yang mudah terdegradasi, apabila analisis DNA inti tidak dapat dilakukan. mtDNA manusia diturunkan secara maternal sehingga setiap individu pada garis keturunan ibu yang sama akan mempunyai tipe mtDNA yang identik. Ukuran DNA mitokondria relatif kecil (14-39 kb) sehingga dapat dipelajari sebagai satu kesatuan yang utuh. mtDNA mempunyai laju polimorfisme yang tinggi dengan laju evolusinya sekitar 5-10 kali lebih cepat dari DNA inti (Hartati et al., 2004). UIN Syarif Hidayatullah Jakarta 14 2.3 Ekstraksi dan Isolasi DNA Prinsip isolasi DNA adalah memisahkan DNA dari komponenkomponen sel. Isolasi DNA dari organisme eukariotik dilakukan melalui proses penghancuran membran sel (lisis), pemisahan DNA dari protein sel, dan purifikasi DNA (Muladno, 2010). Berikut adalah uraian tahapan isolasi DNA: 1. Penghancuran Membran Sel (lisis) Untuk mendapatkan DNA, membran terlebih dahulu dilisiskan dengan senyawa kimia seperti lisozim, EDTA, dan SDS. Lisozim merupakan enzim yang dapat memakan dinding sel. Penggunaan Lisozim biasanya untuk ekstraksi dan isolasi DNA dari sel tumbuhan. EDTA merupakan zat yang dapat merusak membran sel dengan cara mengikat ion Mg2+ yang berfungsi untuk mempertahankan integritas sel maupun mempertahankan aktivitas enzim nuklease yang merusak asam nukleat. SDS (sodium dodesil sulfat) merupakan detergent yang dapat merusak integritas membran sel dengan cara mengikat lipid yang terdapat pada membran sel. Larutan pelisis sel umumnya terdiri atas satu atau lebih detergent seperti SDS, NP-40, atau Triton X-100. Sel debris yang ditimbulkan akibat cairan pelisis dilakukan setrifugasi sehingga hanya tertinggal molekul nukleotida. 2. Pemisahan DNA dari Protein Sel Protein pada sel dihancurkan dengan bantuan enzim proteinase K. Enzim ini dapat memecahkan protein histon sehingga DNA terurai. Untuk menghilangkan protein dari larutan, digunakan fenol (untuk mengikat protein dan sebagian kecil RNA) dan kloroform (untuk membersihkan protein dan polisakarida dari larutan). Umumnya ditambahkan dengan perbandingan 1:1. Kemudian dilakukan sentifugasi kembali untuk mengendapkan molekul yang berat, dan dipisahkan DNA yang berada pada supernatant. 3. Purifikasi DNA DNA yang telah dibersihkan dari protein masih tercampur dengan RNA. Untuk membersihkan molekul RNA dari larutan, enzim RNAse UIN Syarif Hidayatullah Jakarta 15 digunakan untuk mendegradasi molekul RNA. Dengan hilangnya protein dan RNA, maka DNA dapat diisolasi secara utuh. Molekul DNA yang telah diisolasi tersebut kemudian dimurnikan dengan cara presipitasi menggunakan etanol absolut dan larutan garam. Dengan adanya larutan garam (kation monovalen seperti Na+), pada suhu -20oC etanol absolut dapat mengendapkan DNA dengan baik sehingga mudah dipisahkan dengan cara sentrifugasi. Gambar 2.9. Presipitasi DNA setelah Penambahan Etanol Absolut (Sumber: www.learn.genetics.utah.edu) Pada umumnya isolasi DNA dengan metode konvensional yang dijelaskan sebelumnya cukup melelahkan karena membutuhkan waktu hingga beberapa jam bahkan beberapa hari. Selain itu, berkembang pesatnya kebutuhan di bidang diagnostik molelular dan filogeni molekular yang menuntut kecepatan, prosedur yang sederhana, hasil yang akurat dalam ekstraksi DNA dari berbagai jenis sampel, menciptakan pengembangan teknologi baru untuk pengektraksian DNA yang mudah dan lebih cepat dari sebelumnya. Salah satu pengembangan teknik purifikasi DNA adalah dengan bantuan spin column yang mengandung silicon dioxide sebagai filter yang dapat mengabsorpsi asam nukleat (Chee Tan dan Chin Yiap, 2009). Prinsip ekstraksi dan isolasi DNA dengan metode ini adalah berdasarkan tingginya afinitas muatan negatif dari rantai utama DNA terhadap partikel silika yang bermuatan positif. DNA dapat terikat dengan kuat pada silika dibawah UIN Syarif Hidayatullah Jakarta 16 kondisi garam chaotropic seperti Guanidine-HCl. Garam ini dapat memecah ikatan hidrogen antara ion oksigen pada DNA dengan ion hidrogen pada air. DNA yang terabsorpsi oleh silika dapat dipisahkan dari protein dan sel debris dengan pencucian. DNA murni kemudian dapat dielusi dari silika dengan buffer Tris-EDTA atau Aquabidest (Gjerse et al., 2009). 2.4 Elektroforesis Gel Agarosa Elektroforesis adalah suatu cara untuk memisahkan fraksi suatu campuran berdasarkkan pergerakan partikel koloid yang bermuatan dibawah pengaruh medan listrik. Cara elektroforesis telah digunakan untuk analisa virus, asam nukleat, enzim, protein, serta molekul-molekul organik dengan berat molekul rendah seperti asam amino (Westermeier, 2004). Salah satu gel yang dapat digunakan pada elektroforesis adalah gel agarosa. Agarosa digunakan untuk memisahkan, mengidentifikasi, dan memurnikan fragmen-fragmen DNA dengan ukuran molekul lebih besar dari 100 pb dan dijalankan secara horizontal. Molekul DNA termasuk senyawa bermuatan negatif. Sifat ini menjadikan molekul DNA yang ditempatkan pada medan listrik akan bermigrasi menuju kutub positif Mobilitas fragmen DNA pada gel elektroforesis sangat dipengaruhi oleh beberapa faktor (Sambrook et al., 2001), yaitu: 1. Ukuran Molekul DNA Migrasi DNA terutama ditentukan oleh ukuran panjang DNA. Fragmen DNA yang berukuran kecil akan bermigrasi lebih cepat dibandingkan dengan fragmen DNA yang berukuran lebih besar. Sehingga elektroforesis mampu memisahkan fragmen berdasarkan ukuran panjangnya. 2. Konsentrasi Agarosa Migrasi molekul DNA pada gel berkonsentrasi lebih rendah lebih cepat daripada migrasi molekul DNA yang sama pada gel berkonsentrasi tinggi. UIN Syarif Hidayatullah Jakarta 17 Tabel 1. Kisaran umum konsentrasi agarosa (Muladno, 2010) Konsentrasi Efisiensi Pemisahan Agarosa (%) DNA (kb) 0,3 5-60 0,6 1-20 0,7 0,8-10 0,9 0,5-7 1,2 0,4-6 1,5 0,2-3 2,0 0,1-2 3. Konformasi DNA Konformasi atau bentuk rangkaian molekul DNA berukuran sama akan bermigrasi dengan kecepatan yang berbeda. DNA dalam bentuk sirkular akan lebih cepat bermigrasi dibandingkan dengan DNA bentuk linier. 4. Voltase yang Digunakan Kecepatan migrasi DNA sebanding dengan tingginya voltase yang digunakan. Akan tetapi, apabila penggunaan voltase terlalu tinggi, mobilitas molekul DNA meningkat secara tajam. Hal ini mengakibatkan efektifitas pemisahan molekul DNA menurun dengan meningkatnya voltase yang digunakan. Penggunaan voltase yang ideal untuk mendapatkan separasi molekul DNA berukuran lebih besar 2 kb adalah tidak lebih dari 5 Volt per cm. 5. Etidium Bromida Keberadaan etidium bromida di dalam gel dapat mengakibatkan pengurangan tingkat kecepatan migrasi molekul DNA linear sebesar 15%. 6. Komposisi Larutan Buffer Apabila tidak ada kekuatan ion di dalam larutan, maka aliran listrik akan sangat minimal dan migrasi DNA sangat lambat. Sementara larutan buffer berkekuatan ion tinggi akan meningkatkan panas, sehingga aliran listrik menjadi sangat maksimal. Ada kemungkinan gel akan meleleh dan DNA dapat mengalami denaturasi. UIN Syarif Hidayatullah Jakarta 18 Visualisasi fragment DNA dilakukan dengan penambahan larutan etidium bromida yang akan masuk (interkalasi) di antara ikatan hidrogen pada DNA, sehingga pita fragmen DNA akan terlihat di bawah lampu UV Pita DNA yang berpendar pada gel agarosa menunjukkan hasil positif bahwa terdapat DNA pada setiap lajur. Larutan etidium bromida sangat berbahaya dan bersifat karsinogen, oleh karena itu semua larutan yang mengandung etidium bromida harus didekontaminasi sebelum dibuang. Selain etidium bromida, pewarnaan dapat dilakukan dengan larutan SYBR safe sebagai penggantinya (Muladno, 2010; Sambrook dan Russel, 2001; Gaffar, 2007). Gambar 2.10. Interkalasi Etidium Bromida pada DNA (Sumber: www.sciencemag.org) 2.5 PCR 2.5.1 Pengertian PCR PCR (Polymerase Chain Reaction) merupakan suatu metode amplifikasi DNA secara in vitro pada daerah spesifik yang dibatasi oleh dua buah primer oligonukleotida dengan bantuan enzim polymerase, dimana potongan DNA tertentu dapat dilipat gandakan (Zyskind dan Bernstain, 1992). Metode ini paling banyak dipelajari dan digunakan secara luas. Dalam waktu sembilan tahun sejak pertama kali dikemukakan oleh ilmuan dari Cetus Corporation, Kary Mullis, PCR telah berkembang UIN Syarif Hidayatullah Jakarta 19 menjadi teknik utama dalam laboratorium biologi molekuler, antara lain untuk transkripsi in vitro dari PCR template, PCR rekombinan, DNAse I footprinting, sequencing dengan bantuan phage promoters, dan sebagainya (Putra, 1999). Teknik ini memungkinkan adanya amplifikasi antara dua region DNA yang diketahui, hanya di dalam tabung reaksi, tanpa perlu memasukkannya ke dalam sel (in vivo). Primer yang digunakan sebagai pembatas daerah yang diamplifikasi merupakan DNA untai tunggal yang urutannya komplemen dengan DNA template-nya. Amplifikasi ini dapat menghasilkan lebih dari satu kali DNA asli. 2.5.2 Tahapan PCR Menurut Sambrook et al., (2001), tahapan yang terjadi dalam proses amplifikasi DNA pada PCR yaitu pemisahan (denaturasi) rantai DNA template, penempelan (annealing) pasangan primer pada DNA target dan pemanjangan (extension) primer atau reaksi polimerisasi yang dikatalisis oleh DNA polimerase. Gambar 2.12. Tahapan Proses PCR (Sumber: www.bioserv.fiu.edu) 1. Denaturasi DNA template Pada tahap ini, DNA untai ganda akan membuka menjadi dua untai tunggal. Hal ini disebabkan karena suhu denaturasi yang UIN Syarif Hidayatullah Jakarta 20 tinggi menyebabkan putusnya ikatan hidrogen diantara basa-basa yang komplemen. Denaturasi biasanya dilakukan antara suhu 9095oC selama 3 menit untuk meyakinkan bahwa molekul DNA target yang ingin dilipat gandakan jumlahnya benar-benar telah terdenaturasi menjadi untai tunggal. Untuk denaturasi berikutnya, waktu yang diperlukan hanya 30 detik pada suhu 95oC atau 15 detik pada suhu 97oC. Suhu denaturasi dipengaruhi oleh sekuen DNA target. Jika DNA target kaya akan G-C maka diperlukan suhu yang lebih tinggi, hal ini dikarenakan ikatan hidrogen pada G-C lebih banyak dibandingkan ikatan A-T. Selain itu, suhu denaturasi juga tidak boleh terlalu tinggi dan waktu denaturasi yang terlalu lama, karena dapat mengakibatkan hilangnya atau berkurangnya aktivitas enzim Taq polymerase. 2. Penempelan Primer (annealing) Primer akan menuju daerah yang spesifik, dimana daerah tersebut memiliki komplemen dengan primernya. Pada proses annealing ini, ikatan hidrogen akan terbentuk. Selanjutnya, enzim Taq DNA polymerase akan berikatan sehingga ikatan hidrogen tersebut akan menjadi sangat kuat dan tidak akan putus kembali. Proses ini biasanya dilakukan pada suhu 50-60oC. Spesifisitas PCR sangat tergantung pada suhu melting (Tm) primer, yaitu suhu dimana separuh jumlah primer menempel pada template. Temperatur penempelan yang digunakan biasanya 5oC di bawah Tm, dimana formula untuk menghitung Tm = 4oC (G+C) + 2oC (A+T). Semakin panjang ukuran primer, menempel tadi semakin tinggi temperaturnya. 3. Reaksi polimerisasi (extension) Primer yang telah akan mengalami perpanjangan pada sisi 3' nya dengan penambahan dNTP yang komplemen dengan template oleh DNA polimerase. Umumnya reaksi polimerisasi (extension) atau perpanjangan rantai, terjadi UIN Syarif Hidayatullah Jakarta 21 pada suhu 72oC karena merupakan suhu optimum Taq polymerase. Kecepatan penyusunan nukleotida oleh enzim tersebut pada suhu 72oC diperkirakan antara 35 sampai 100 nukleotida per detik, bergantung pada buffer, pH, konsentrasi garam, dan molekul DNA target. Dengan demikian, untuk produk PCR sepanjang 2000 pasang basa, waktu 1 menit sudah lebih dari cukup untuk tahap pemanjangan primer ini. Ketiga tahapan tersebut terjadi dalam satu siklus. Produk DNA pada siklus amplifikasi pertama akan menjadi cetakan pada siklus berikutnya. Produk yang dihasilkan bersifat eksponensial (2n), dengan n adalah jumlah siklus yang dilakukan (Keller dan Manak, 1989). Ini mengakibatkan PCR sangat efektif karena dapat menghasilkan DNA dalam jumlah mikrogram dari jumlah DNA awal dalam pikogram (Zyskind dan Bernstain, 1992). Gambar 2.13. Pelipatgandaan Molekul DNA Target (Sumber: www.bioserv.fiu.edu) 2.5.3 Komponen PCR Beberapa komponen penting yang dibutuhkan dalam PCR adalah DNA template, primer, enzim Taq DNA Polymerase, deoxyribonucleaside triphosphate (dNTP’s), dan buffer PCR. DNA template merupakan DNA yang akan diamplifikasi, dapat berupa untai tunggal atau untai ganda. Primer yang digunakkan sebaiknya berukuran 20-26 pasang basa, dengan UIN Syarif Hidayatullah Jakarta 22 komposisi 50-60% C+G, sekuens DNA kedua primer tidak saling berkomplemen yang akan menyebabkan terjadinya penempelan antar primer (primer-dimer), dan sekuens DNA dalam satu primer tidak saling berkomplemen yang akan mengakibatkan terbentuknya struktur sekunder dan mengurangi efisiensi PCR. Deoxyribonucleaside triphosphate (dNTP’s) merupakan bahan dasar pembentuk DNA yang terdiri dari dATP, dTTP, dGTP, dan dCTP. Taq DNA Polymerase berfungsi sebagai katalisator terjadinya pemanjangan primer. Buffer PCR yang biasanya terdiri atas bahan-bahan kimia untuk mengkondisikan reaksi agar berjalan optimum dan menstabilkan enzim DNA polymerase. Ion Mg2+ yang biasanya terdapat dalam buffer berfungsi sebagai kofaktor enzim. Keberadaan ion ini sangat penting karena ion Mg2+ bebas akan mengikat DNA template, primer dan membentuk kompleks terlarut dengan dNTP untuk membuat subtrat yang akan dikenali oleh enzim Taq Polymerase (Zyskind dan Bernstain, 1992). 2.6 Real-Time PCR Real-Time PCR adalah suatu metoda analisa yang dikembangkan dari reaksi PCR. Dalam ilmu biologi molekular, Real-Time PCR (juga dikenal sebagai quantitative real time polymerase chain reaction (Q-PCR atau qPCR) atau kinetic polymerase chain reaction) adalah suatu teknik pengerjaan PCR di laboratorium untuk mengamplifikasi sekaligus mengkuantifikasi jumlah target molekul DNA hasil amplifikasi tersebut. Instrumen Real-Time PCR mendeteksi amplikon dengan mengukur peningkatan pewarna (dye) fluorescent yang berpendar ketika terikat dengan untai ganda DNA. Karena sifat inilah maka pertumbuhan fragment DNA hasil amplifikasi dapat diikuti secara seketika, semakin banyak DNA yang terbentuk semakin `tinggi pula intensitas fluorescent yang dihasilkan. Alat ini dapat mendeteksi secara akurat konsentrasi DNA hingga hitungan pikogram atau setara dengan sel tunggal karena sensitifitas dye yang sangat tinggi. Hasil peningkatan fluorescent digambarkan melalui kurva UIN Syarif Hidayatullah Jakarta 23 amplifikasi yang menunjukkan tiga fasa yaitu fasa awal, fasa eksponensial atau puncak dan fasa plateau atau stabil (Vaerman, 2004). Gambar 2.14. Bentuk Kurva Real-Time PCR (Sumber: bio-rad.com) 2.6.1. Pewarna Fluoresensi Real-time PCR menggunakan pewarna fluoresensi dan probe dengan fluoresensi. Probe berupa oligonukleotida yang tersusun dari sekuen spesifik dan akan berpasangan dengan sekuen DNA cetakan yang akan diamplifikasi. Macam-macam fluoresensi yang umumnya digunakan antara lain SYBR Green, Hydrolysis probes, Hybridization probes, dan Molecular beacons. Fluoresensi akan berpendar hijau saat berikatan dengan DNA cetakan (Kubista et al., 2006). Pewarna yang umum digunakan adalah SYBR Green dan Hydrolysis Probe. a. Hydrolysis Probe Hydrolisis probe menggunakan oligonukleotida spesifik yang berkomplemen dengan DNA target disebut Probe. Probe dilabeli oleh dua molekul, yaitu reporter pada ujung 5’ probe yang merupakan pewarna fluoresensi dan quencer pada ujung 3’ probe yang merupakan molekul penerima sinyal fluoresensi. Hydrolysis probe memiliki prinsip kerja, yaitu saat probe belum berkomplemen dengan DNA target, molekul reporter akan UIN Syarif Hidayatullah Jakarta 24 mengeksitasikan sinyal fluoresensi ke molekul quencer karena jarak antara kedua molekul berdekatan. Probe akan berkomplemen dengan DNA target saat mencapai suhu annealing, lalu mekanisme eksitasi sinyal fluoresensi dari reporter ke quencer terhenti karena jarak kedua molekul berjauhan. DNA polimerase akan mengelongasi DNA target sampai DNA polimerase dan probe berdekatan maka 5’nuklease yang terdapat pada DNA polimerase akan menghidrolisis molekul reporter sehingga emisi sinyal fluoresensi dapat tertangkap oleh detektor pada Real Time PCR (Shipley, 2007). Hydrolysis probe biasa digunakan dalam multiplex pada qRT PCR yang menggunakan DNA target dan pasangan primer lebih dari satu dalam satu reaksi karena probe akan berikatan secara spesifik dengan beberapa DNA target yang berbeda (BioRad, 2006). b. SYBR Green Prinsip dari SYBR Green adalah pewarna fluoresensi akan mengikat bagian minor dari untai ganda DNA sehingga menghasilkan pendaran warna hijau pada akhir fase extension selama proses Real Time PCR berlangsung (Shipley, 2007). Intensitas fluoresensi yang semakin tinggi meenunjukkan peningkatan jumlah untai ganda DNA yang terdeteksi oleh detektor. Intensitas fluoresensi yang akan dihitung dalam analisis kualitatif maupun kuantitatif pada qRT PCR. SYBR Green umumnya digunakan dalam singleplex pada Real Time PCR yang menggunakan satu DNA target dan sepasang primer. SYBR Green lebih sering digunakan dibandingkan Hydrolisis probe karena memiliki beberapa keunggulan, yaitu dapat digunakan untuk beberapa pengujian karena mampu bekerja pada semua jenis primer, teknik pengujian lebih sederhana karena tidak perlu merancang probe, dan harga pewarna fluoresensi yang relatif lebih rendah (Shipley, 2007). SYBR Green memiliki kelemahan, yaitu SYBR Green dapat mengikat DNA untai ganda UIN Syarif Hidayatullah Jakarta 25 pada bagian tidak spesifik sehingga primer-dimer juga akan diikat oleh SYBR Green. Namun, produk tidak spesifik dapat dianalisis keberadaannya dalam analisis melting curve (Valasek & Repa ,2005; Kubista et al., 2006). Gambar 2.16. Mekanisme Hydrolysis Probe dan SYBR Green (Sumber: Thermoscientificbio.com) UIN Syarif Hidayatullah Jakarta 26 BAB 3 METODOLOGI PENELITIAN 3.1 Tempat dan Waktu Penelitian 3.1.1 Tempat Penelitian ini dilaksanakan di Laboratorium Analisis Obat & Pangan Halal; Laboratorium Penelitian 2; dan Laboratorium Biokimia/ Patologi Klinis. Fakultas Kedokteran dan Ilmu Kesehatan, Universitas Islam Negeri Syarif Hidayatullah Jakarta. 3.1.2 Waktu Waktu pelaksanaan penelitian dilakukan pada bulan Maret 2014 hingga September 2014. 3.2 Alat dan Bahan 3.2.1 Alat Real Time PCR (Light Cycler® 480 - Roche), Multiwell Plate 96 (Roche), Sealing Foil (Roche), Mikropipet 0.5-10 μl (Biorad), Mikropipet 2-20 μl (Biorad), Mikropipet 20-200 μl (Biorad), Mikropipet 100-1000 μl (Biorad), Microtips volume 10 μl; 200 μl; dan 1000 μl (Genfollower), Spektrofotometri UV DNA (BioDrop), Gel Documentation (AE-6933 ATTO), satu set alat Elektroforesis Agarosa (Biorad), Digital Waterbath (SB-100 Eyela), Vortex, Sentrifugator (5417R – Eppendorf), Filter Tube dan Collection Tube (Kit High Pure PCR Template Preparation – Roche), plastic wrap, alumunium foil, Microsentrifuge tube volume 1,5 ml (Biogenix), pisau steril, kaca arloji, erlenmeyer 50 ml, dan spatula. 3.2.2 Bahan Daging sapi segar, daging babi segar, gelatin sapi (Sigma Aldrich), gelatin babi (Sigma Aldrich), Tris Base (SBS Genetech), asam asetat glasial (Merck), EDTA (Merck), satu set reagen isolasi DNA meliputi Tissue Lysis Buffer; Binding Buffer; Proteinase K; Inhibitor Removal Buffer; Washing Buffer; dan Elution Buffer (Kit High Pure PCR Template 26 UIN Syarif Hidayatullah Jakarta 27 Preparation - Roche), Aquabidest (IKA Pharmindo), Etanol Absolut (Merck), Isopropanol (Merck), Etidium Bromida 10 mg/ml (Bio Basic Inc.), Agarosa (Fermentas), loading dye (Promega), SYBR Green I Master (LC 480 - Roche), Probe Master (LC 480 - Roche), Aquadest (PCR Grade - Roche) dan Primer-probe (Alpha DNA). Tabel 3. Urutan Basa Primer dan Probe (Tanabe et al., 2007) Nama Primer Babi Forward Reverse Probe Sapi Forward Reverse Probe Runutan Basa 5'-CTTGCAAATCCTAACAGGCCTG-3' 5'-CGTTTGCATGTAGATAGCGAATAAC-3' 5'-(FAM)-ACAGCTTTCTCATCAGTTAC-(BHQ1)-3' 5'-CCCGATTCTTCGCTTTCCAT-3' 5'-CTACGTCTGAGGAAATTCCTGTTG-3' 5'-(FAM)-CATCATAGCAATTGCC-(BHQ1)-3' 3.3 Prosedur Penelitian 3.3.1 Ekstraksi dan Isolasi DNA Proses ekstraksi dan isolasi DNA dilakukan dengan menggunakan kit komersial High Pure PCR Template Preparation (Roche). 1. Preparasi Sampel a. Daging Segar (Rochea, 2012; Erwanto et al., 2012) Sebanyak 50 mg masing-masing daging sapi segar dan daging babi segar dicincang halus dengan pisau steril. Masing masing daging tersebut dimasukkan ke dalam microsentrifuge tube. Kemudian ke dalam tube tersebut masing-masing ditambahkan 200 μl Tissue Lysis Buffer dan 40 μl larutan Proteinase K. Campuran divortex selama 1 menit dan diinkubasi pada suhu 57oC selama 20 jam dalam digital waterbath. Larutan daging yang dihasilkan selanjutnya dilakukan proses ekstraksi dan isolasi DNA. b. Gelatin (Rochea, 2012; Erwanto et al., 2012) Sebanyak 50 mg masing-masing gelatin sapi dan gelatin babi ditimbang dan dimasukkan ke dalam microsentrifuge tube. Kemudian ke dalam tube tersebut masing-masing ditambahkan 100 μl Aquabidest, 200 μl Tissue Lysis Buffer dan 40 μl larutan Proteinase K. UIN Syarif Hidayatullah Jakarta 28 Campuran divortex selama 1 menit dan diinkubasi pada suhu 57oC selama 20 jam dalam digital waterbath. Larutan daging yang dihasilkan selanjutnya dilakukan proses ekstraksi dan isolasi DNA. 2. Proses Ekstraksi dan Isolasi DNA (Rochea, 2012 dengan modifikasi) Larutan daging sapi, daging babi, gelatin sapi dan gelatin babi yang telah dinkubasi sebelumnya selama 20 jam, masing-masing ditambahkan 200 μl larutan Binding Buffer. Campuran divortex segera selama 10 detik dan diinkubasi kembali pada suhu ± 70oC selama 10 menit dalam digital waterbath. Ke dalam masing-masing tube tesebut kemudian ditambahkan 150 μl isopropanol absolut dan dihomogenkan dengan vortex selama 10 detik. Campuran dipipet dan dimasukkan ke dalam Filter Tube yang telah dipasangkan Collection Tube. Kemudian tube ditutup dan disentrifugasi pada kecepatan 8000 rpm selama 1 menit. Filter tube dilepaskan dari collection tube dan cairan yang melewati filter dibuang bersamaan collection tube. Filter tube dipasangkan kembali dengan collection tube yang baru. Kemudian 500 μl Inhibitor Removal Buffer ditambahkan melalui penyangga atas Filter Tube dan disentrifugasi kembali dengan kecepatan 8000 rpm selama 1 menit. Filter tube dilepaskan dari collection tube dan cairan yang melewati filter dibuang bersamaan collection tube. Filter tube dipasangkan kembali dengan collection tube yang baru. Kemudian 500 μl Washing Buffer ditambahkan dan disentrifugasi kembali pada kecepatan 8000 rpm selama 1 menit. Pencucian dengan Washing Buffer dilakukan sekali lagi. Setelah itu, filter tube dilepaskan dari collection tube dan cairan yang melewati filter dibuang. Filter tube dipasang kembali dengan collection tube dan disetrifugasi kembali selama 10 detik pada kecepatan maksimum agar semua Washing Buffer tidak tertinggal pada filter. 3. Elusi DNA (Rochea, 2012 dengan modifikasi) Filter tube dan Collection tube yang telah disentrifugasi pada kecepatan maksimum kemudian Filter tube dipisahkan dengan collection UIN Syarif Hidayatullah Jakarta 29 tube. Collection tube dibuang dan Filter tube tersebut kemudian dipasangkan pada Microsentrifuge tube steril. Ke dalam filter yang mengandung DNA daging sapi dan daging babi, masing-masing ditambahkan 200 μl Elution Buffer hangat (70oC). Sedangkan ke dalam filter yang mengandung DNA gelatin sapi dan DNA gelatin babi, masing-masing ditambahkan 150 μl Elution Buffer hangat (70oC). Filter dan Microsentrifuge tube yang telah ditambahkan Elution Buffer kemudian disetrifugasi selama 1 menit pada kecepatan 8000 rpm. Filter tube dilepaskan dari Microsentrifuge tube. Pada Microsentrifuge tube telah mengandung isolat DNA. Isolat DNA yang didapatkan dianalisis keberadaannya dengan elektroforesis agarosa dan dianalisis konsentrasi dan kemurnian DNA dengan spektrofotmetri UV. 3.3.2 Analisis Isolat DNA 3.3.2.1 Analisis Isolat DNA dengan Elektroforesis Agarosa a. Pembuatan Buffer Elektroforesis TAE 10x, 1 liter (Biorad, 2012) Sebanyak 48,4 g Tris Base dilarutkan dalam 800 mL aquabidest. Ke dalam larutan tersebut ditambahkan 11,4 mL asam asetat glasial dan 20 ml 0,5 M EDTA pH 8. Larutan dihomogenkan dan dimasukkan ke dalam labu ukur 1000 ml. Kemudian ke dalam labu ukur tersebut ditambahkan aquabidest sampai tanda batas. b. Pembuatan Buffer Elektroforesis TAE 1x, 1 liter (Biorad, 2012) Sebanyak 100 ml TAE 10x dimasukkan ke dalam labu ukur 1000 ml dan ditambahkan aquabidest sampai tanda batas. Larutan tersebut kemudian dihomogenkan. c. Pembuatan Gel Agarosa 1% (Sambrook et al., 2001) Sebanyak 0,5 g agarosa dilarutkan dengan 50 mL TAE 1x di dalam erlenmeyer. Erlenmeyer ditutup dengan alumunium foil, kemudian dipanaskan di atas hot plate sampai larutan menjadi bening dan mendidih. Larutan agarosa didiamkan hingga suhu 60oC dan ditambahkan 2,5 μL etidium bromida 10 mg/ml, kemudian larutan diaduk hingga homogen. Larutan dituangkan pada gel caster dan comb UIN Syarif Hidayatullah Jakarta 30 disisipkan untuk membuat well (sumur). Larutan agarosa dibiarkan hingga mengeras. Setelah gel mengeras dan membentuk agar, gel diletakkan pada chamber electrophoresis dengan posisi well pada muatan negatif (warna hitam). Kemudian buffer TAE 1x dituang hingga gel terendam dalam chamber electrophoresis namun tidak melebihi garis batas maksimum buffer. d. Loading Sampel (Sambrook et al., 2001 dengan modifikasi) Plastic wrap direkatkan pada wadah mendatar sebagai tempat melakukan pencampuran sampel yang akan dimasukkan ke dalam well pada gel. Masing-masing sebanyak 5 μL isolat DNA dipipet dan ditaruh di atas plastic wrap. Kemudian masing-masingnya ditambahkan 1 μL loading dye dan dihomogenkan dengan menaik turunkan mikropipet. Masing-masing campuran tersebut dipipet dan dimasukkan ke dalam well agarosa yang telah dibuat sebelumnya dengan urutan dari kiri ke kanan adalah; DNA daging sapi; DNA daging babi; DNA gelatin sapi; DNA gelatin babi. e. Running Sampel (Sambrook et al., 2001) Chamber elektroforesis yang telah mengandung gel agarosa, DNA, dan buffer TAE 1X kemudian ditutup rapat dan kabel chamber dihubungkan dengan power supply. Kabel merah (muatan positif) dipasangkan pada lubang merah dan kabel hitam (muatan negatif) dipasangkan pada lubang hitam. Running sampel dilakukan selama 45 menit dengan voltase 90 Volt 400 mA. f. Dokumentasi Gel Agarosa (Atto, 2009) Gel agarosa yang telah mengandung DNA terelektroforesis kemudian diletakkan pada Gel Doc untuk visulasi isolat DNA. Tombol ON ditekan pada Saver, Printer dan Transluminator. Stand by LED dipilih untuk melihat posisi Gel. Panjang gelombang dipilih, kemudian exposure Time, Focus dan aperture cahaya diatur. Expose UV. Freeze ditekan dan Save. Data gambar yang akan digunakan sebagai dokumentasi dicetak dengan menekan tombol Print. UIN Syarif Hidayatullah Jakarta 31 3.3.2.2 Analisis DNA Hasil Isolasi dengan Spektrofotometri UV Pada sistem layar, panel “Nucleic Acid” dipilih untuk penentuan konsentrasi dan kemurnian DNA. Sample port dibersihkan dengan tisu steril. Sebanyak 2 μL Elution Buffer ditaruh di atas Sample port dan dianalisis sebagai blangko. Sample port kembali dibersihkan dengan tisu steril. Identitas sampel dimasukkan pada kolom Sampel ID dan sebanyak 2 μL DNA sampel tersebut ditaruh di atas Sample port. Kemudian tombol ”Measure” diklik. DNA dianalis pada panjang gelombang 260 nm dan 280 nm. Hasil instrumentasi akan didapatkan data konsentrasi DNA dalam ng/μl dan kemurnian DNA dengan perbandingan rasio A280 dan A260. (Biodrop, 2012) 3.3.3 Uji Spesifisitas Primer dan Probe dengan Database NCBI Uji spesifisitas primer dan probe dilakukan dengan melakukan BLAST melalui database NCBI. Pada halaman “BLAST”, dipilih menu “nucleotide blast”. Kemudian pada kolom “Enter Query Sequence” dimasukkan urutan basa primer yang akan diuji. Tombol “BLAST” kemudian diklik. Data hasil pengujian berupa daftar spesies yang memiliki kemiripan 99%-100% dengan urutan basa primer yang diuji (NCBI). 3.3.4 Amplifikasi DNA Menggunakan Real-Time PCR 3.3.4.1. Amplifikasi DNA dengan Metode SYBR Green a. Pembuatan Larutan Primer 10 μM dari Larutan Induk Primer 100 μM Sebanyak 10 μL larutan induk primer 100 μM dimasukkan ke dalam Microsentrifuge tube volume 1,5 ml. Kemudian ke dalam tube tesebut ditambahkan 90 μL aquadest. Larutan tersebut dihomogenkan dengan menaik turunkan pegas pada mikropipet. b. Pembuatan SYBR Green Mastermix (Rochec, 2005) Mastermix dibuat dengan volume total 20 μL yang terdiri dari 5 μL DNA yang diuji; 3 μL Aquabidest; 1 μL primer forward 10 μM; 1 μL primer reverse 10 μM; dan 10 μL LightCycler® 480 SYBR Green I UIN Syarif Hidayatullah Jakarta 32 master (enzim Taq DNA Polymerase, SYBR Green 1, dNTP mix, dan 6,4 mM MgCl2). c. Loading Sampel dan SYBR Green Master Mix ke dalam Multiwell Plate (Rochec, 2005) Sebanyak 5 μL DNA yang akan diuji dan 15 μL Mastermix dimasukkan ke dalam multiwell plate pada well yang diinginkan. Campuran dihomogenkan dengan menaik turunkan pegas mikropipet secara perlahan. Multiwell plate kemudian ditutup dengan sealing foil. d. Pengaturan Program Proses Amplifikasi (Rochec, 2005 dengan modifikasi) Pengaturan program LightCycler® 480 Real-Time PCR yang akan digunakan untuk proses amplifikasi, dilakukan dengan percobaan sebagai berikut: Tabel 4. Pengaturan program amplifikasi dengan metode SYBR Green Perc. 1 Perc. 2 Perc. 3 Analisis dengan Primer Sapi Jumlah Siklus Suhu o Pre Incubation 1 Siklus 95 C Amplification 45 siklus 95oC 60oC 72oC Melting Curve 1 Siklus Analysis 95oC 60oC 97oC/5oC Cooling 1 Siklus 40oC Jumlah Siklus Suhu o Pre Incubation 1 Siklus 95 C Amplification 45 siklus 95oC 62oC 72oC Melting Curve 1 Siklus Analysis 95oC 60oC 97oC/5oC Cooling 1 Siklus 40oC Jumlah Siklus Suhu Waktu 10 menit 10 detik 20 detik 30 detik 5 detik 1 menit 10 detik Waktu 10 menit 10 detik 20 detik 30 detik 5 detik 1 menit 10 detik Waktu UIN Syarif Hidayatullah Jakarta 33 Pre Incubation Amplification Melting Curve Analysis Perc. 4 Perc. 1 Perc. 2 1 Siklus 45 siklus 95oC 10 menit 95oC 64oC 72oC 10 detik 20 detik 30 detik 1 Siklus 95oC 60oC 97oC/5oC Cooling 1 Siklus 40oC Jumlah Siklus Suhu o Pre Incubation 1 Siklus 95 C Amplification 45 siklus 95oC 65oC 72oC Melting Curve 1 Siklus Analysis 95oC 60oC 97oC/5oC Cooling 1 Siklus 40oC Analisis dengan Primer Babi Jumlah Siklus Suhu Pre Incubation 1 Siklus 95oC Amplification 45 siklus 95oC 60oC 72oC Melting Curve 1 Siklus Analysis 95oC 60oC 97oC/5oC Cooling 1 Siklus 40oC Jumlah Siklus Suhu o Pre Incubation 1 Siklus 95 C Amplification 45 siklus 95oC 60oC 72oC Melting Curve 1 Siklus Analysis 95oC 5 detik 1 menit 10 detik Waktu 10 menit 10 detik 20 detik 30 detik 5 detik 1 menit 10 detik Waktu 10 menit 10 detik 10 detik 7 detik 5 detik 1 menit 10 detik Waktu 10 menit 10 detik 20 detik 30 detik 5 detik UIN Syarif Hidayatullah Jakarta 34 60oC 97oC/5oC Cooling 1 Siklus 40oC Perc. 3 Jumlah Siklus Suhu o Pre Incubation 1 Siklus 95 C Amplification 45 siklus 95oC 60oC 72oC Melting Curve 1 Siklus Analysis 95oC 60oC 97oC/5oC Cooling 1 Siklus 40oC Keterangan: = Modifikasi yang dilakukan pada proses Real Time PCR 1 menit 10 detik Waktu 10 menit 10 detik 20 detik 25 detik 5 detik 1 menit 10 detik amplifikasi 3.3.4.2. Amplifikasi DNA dengan Metode Hydrolysis Probe a. Pembuatan Primer dan Probe 10 μM dari Larutan Induk 100 μM Sebanyak 10 μL larutan induk primer 100 μM dan probe 100 μM masing-masing dimasukkan ke dalam Microsentrifuge tube volume 1,5 ml. Kemudian ke dalam tube tesebut masing-masing ditambahkan 90 μL aquadest. Larutan tersebut masing-masing dihomogenkan dengan menaik turunkan pegas pada mikropipet. b. Pembuatan Hydrolysis Probe Mastermix (Roched, 2008) Mastermix dibuat dengan volume total 20 μL yang terdiri dari 5 μL DNA yang diuji; 1,4 μL Aquabidest; 1,6 μL primer forward 10 μM; 1,6 μL primer reverse 10 μM; 0,4 μL probe 10 μM; dan 10 μL LightCycler® 480 Probe master (enzim Taq DNA Polymerase, dNTP mix, dan 6,4 mM MgCl2). c. Loading Sampel dan Hydrolisis Probe Mastermix ke dalam Multiwell Plate (Roched, 2008) Sebanyak 5 μL DNA yang akan diuji dan 15 μL Mastermix dimasukkan ke dalam multiwell plate pada well yang diinginkan. Campuran dihomogenkan dengan menaik turunkan pegas mikropipet secara perlahan. Multiwell plate kemudian ditutup dengan sealing foil. UIN Syarif Hidayatullah Jakarta 35 d. Pengaturan Program Proses Amplifikasi (Roched, 2008) Proses pengaturan program LightCycler® 480 Real-Time PCR yang akan digunakan untuk proses amplifikasi dilakukan sebagai berikut: Tabel 5. Pengaturan program amplifikasi dengan metode Hydrolysis Probe Analisis dengan Primer-probe Sapi Pre Incubation Amplification Cooling Pre Incubation Amplification Cooling Jumlah Siklus 1 Siklus 60 siklus Suhu o 95 C 95oC 60oC 1 Siklus 40oC Analisis dengan Primer-probe Babi Jumlah Siklus 1 Siklus 60 siklus 1 Siklus Suhu Waktu 10 menit 10 detik 1 menit 10 detik 95 C Waktu 10 menit 95oC 60oC 40oC 10 detik 1 menit 10 detik o UIN Syarif Hidayatullah Jakarta 36 BAB 4 HASIL DAN PEMBAHASAN Pada penelitian ini dilakukan perbandingan metode SYBR Green dan metode Hydrolysis Probe dalam analisis DNA gelatin sapi dan DNA gelatin babi dengan menggunakan Real Time PCR. Perbandingan metode SYBR Green dan metode Hydrolysis Probe dilihat melalui hasil kurva amplifikasi apakah metode tersebut dapat mengamplifikasi DNA gelatin sapi dan DNA gelatin babi secara spesifik. 4.1. Hasil Analisis Isolat DNA Isolat DNA didapatkan dari proses ekstraksi dan isolasi DNA dengan menggunakan kit komersial High Pure PCR Template Preparation. Prinsip kit ini yaitu DNA diabsorpsi oleh Silikon Dioksida (SiO2) sehingga DNA mudah dipisahkan dari protein dan sel debris hasil pelisisisan sel dengan cara sentrifugasi (Rocheb, 2012). Reagen yang ditambahkan pada proses ekstraksi dan isolasi DNA meliputi: Tissue lysis buffer sebagai pelisis membran sel, proteinase K sebagai enzim pemecah makromolekul protein menjadi molekul lebih kecil, Binding buffer sebagai chaotropic agent agar DNA dapat terabsorbsi kuat dengan silikon dioksida yang terdapat pada filter, Inhibitor Removal Buffer sebagai buffer yang dapat menghilangkan zat pengotor yang mengganggu proses PCR, Washing buffer sebagai pencuci garam chaotropic dan pengotor lainya, dan Elution Buffer sebagai pengelusi DNA dari silikon dioksida (Rocheb, 2012). Isolat DNA yang didapatkan dianalisis dengan Elektroforesis Agarosa dan Spektrofotometer UV. 4.1.1. Hasil Analisis Isolat DNA dengan Elektroforesis Agarosa Isolat DNA divisualisasi keberadaannya dengan elektroforesis gel agarosa 1% seperti gambar sebagai berikut: 36 UIN Syarif Hidayatullah Jakarta 37 Gambar 4.1 Hasil elektroforesis isolat DNA *Keterangan: (1) Daging Babi; (2) Daging Sapi; (3) Gelatin Babi; (4) Gelatin Sapi Melalui elektroforesis agarosa, DNA dapat tervisualisasi karena larutan Etidium Bromida yang ditambahkan ter-interkalasi dengan untai ganda DNA, sehingga pita fragmen DNA dapat terlihat di bawah lampu UV (Gaffar, 2007). Pada Gambar 4.1 menunjukkan pita yang jelas pada isolat DNA sapi dibandingkan pada isolat DNA babi. Kedua pita tersebut merupakan pita yang smear. Hasil pita yang smear pada gel elektroforesis dapat disebabkan tidak utuhnya DNA yang terisolasi dimana fragmenfragmen DNA dengan ukuran yang berbeda tertahan pada gel sesuai dengan ukuran pasang basa DNA (Lewis, 2001). Terjadinya fragmentasi DNA daging dikarenakan DNA yang terisolasi mengalami degradasi saat proses pencincangan atau selama proses isolasi berlangsung. Hasil elektroforesis pada isolat DNA gelatin babi dan DNA gelatin sapi menunjukkan tidak adanya pita. Tidak adanya pita pada gel agarosa, bukanlah berarti DNA gelatin tidak berhasil diisolasi. Hal ini dapat dikarenakan jumlah DNA hasil isolasi pada gelatin terlalu kecil dan banyak yang terdegradasi akibat pengolahann sehingga tidak tervisualisasi pada gel agarosa. Batas minimal konsentrasi DNA yang dapat terdeteksi pada elektroforesis agarosa dengan pewarna etidium bromida adalah 25 ng (Lewis, 2001). UIN Syarif Hidayatullah Jakarta 38 4.1.2 Hasil Analisis Isolat DNA dengan Spektrofotometri UV Konsentrasi dan kemurnian isolat DNA dianalisis dengan menggunakan spektrofotometri UV. Hasil analisis terlihat pada tabel 6. Tabel 5. Konsentrasi dan kemurnian DNA hasil ekstraksi dan isolasi Kemurnian No. Sampel Konsentrasi (A280/A260) 1. Daging Sapi 85,99 ng/μl 1,804 2. Daging Babi 80,08 ng/μl 1,824 3. Gelatin Sapi 19,38 ng/μl 1,566 4. Gelatin Babi 13,63 ng/μl 1,573 Analisis konsentrasi dan kemurnian isolat DNA menggunakan spektrofotometri UV dilakukan pada panjang gelombang 260 nm dan 280 nm. Konsentrasi DNA yang didapatkan pada daging berkisar antara 80 ng/μl – 85 ng/μl dengan kemurnian kisaran 1,8. Sedangkan konsentrasi DNA yang didapatkan pada gelatin berkisar antara 13 ng/μl – 19 ng/μl dengan kemurnian kisaran 1,5. DNA dapat dikatakan murni dari protein apabila nilai rasio A260/A280 yaitu berkisar antara 1,8 sampai 2,0 (Sambrook et al., 2001). Konsentrasi isolat DNA daging yang didapatkan jauh lebih besar dibandingkan DNA gelatin. Hal ini dikarenakan pada daging memiliki jumlah sel yang banyak, sedangkan gelatin merupakan produk hasil olahan dari kolagen sehingga isolat DNA gelatin yang didapatkan sedikit. Nilai kemurnian hasil isolasi pada gelatin dengan nilai dibawah 1,8 menunjukkan bahwa masih bayak pengotor protein pada hasil isolat DNA gelatin (Sambrook et al., 2001). Berdasarkan penelitian yang telah dilakukan Glasel (1995), nilai kemurnian 1,5 memiliki perbandingan persentase teoritis protein dan asam nukleat yaitu sekitar 80% dan 20%. Hal ini menunjukkan bahwa kit komersial ekstraksi dan isolasi DNA yang digunakan belum mampu memisahkan DNA gelatin dari pengotor protein dengan baik. 4.2 Hasil Uji Spesifisitas Primer dan Probe dengan Database NCBI Runutan basa primer dan probe yang diperoleh dari penelitian Tanabe S. et al., (2007), dianalisa secara in silico dengan cara BLAST berdasarkan UIN Syarif Hidayatullah Jakarta 39 informasi database NCBI melalui internet. Tujuan dari proses ini adalah untuk mengetahui apakah primer dan probe yang digunakan merupakan primer-probe spesifik yang hanya mengamplifikasi satu jenis spesies. Gambar 4.2 Hasil uji spesifitas primer babi dengan BLAST melalui database NCBI *Keterangan: = primer forward babi; = Probe babi; = Primer reverse babi. Hasil BLAST dari database NCBI pada primer forward babi, primer reverse babi, dan probe babi menunjukkan bahwa primer–probe yang digunakan spesifik untuk DNA babi dengan nilai maksimum identitas 100%. DNA target yang akan diamplifikasi yaitu pada DNA mitokondria-sitokrom b dengan panjang amplifikasi 131 pasang basa (Gambar 4.2). Gambar 4.3 Hasil Uji Spesifitas Primer Sapi dengan BLAST Melalui Database NCBI *Keterangan: = primer forward sapi; = Probe sapi; = Primer reverse sapi. UIN Syarif Hidayatullah Jakarta 40 Hasil BLAST dari database NCBI pada primer-probe sapi juga menunjukkan bahwa primer-probe sapi yang digunakan spesifik untuk DNA sapi digunakan spesifik untuk DNA sapi dengan nilai maksimum identitas 100%. DNA target yang akan diamplifikasi yaitu pada DNA mitokondriasitokrom b dengan panjang amplifikasi 120 pasang basa (Gambar 4.3). 4.3 Hasil Amplifikasi DNA Gelatin Sapi dan Gelatin Babi Menggunakan Real Time PCR Amplifikasi DNA gelatin sapi dan DNA gelatin babi menggunkan Real Time PCR dilakukan dengan dua metode, yaitu metode SYBR Green dan metode Hydrolysis Probe. Hasil amplifikasi dari kedua metode dibandingkan satu sama lain dengan melihat spesifisitas dalam mengamplifikasi DNA gelatin sapi dan DNA gelatin babi. 4.3.1 Hasil Amplifikasi DNA Gelatin Sapi dan Gelatin Babi Menggunakan Real Time PCR dengan Metode SYBR Green Konsentrasi DNA daging sapi; DNA daging babi; DNA gelatin sapi; dan DNA gelatin babi yang digunakan dalam proses amplifikasi masingmasing adalah 8,59 ng/μL; 8,0 ng/μL; 19,38 ng/μL; dan 13,63 ng/μL. Konsentrasi DNA daging yang digunakan dilakukan pengenceran sebanyak sepuluh kali dari isolat DNA agar mendapatkan kurva amplifikasi yang optimal, yaitu kurva yang didapatkan tidak terlalu cepat mencapai fasa plateau pada awal siklus (Roched, 2008). Analisis kurva amplifikasi dilihat melalui kenaikan kurva dan nilai CP (Crossing Point) pada kurva amplifikasi. Spesifisitas hasil amplifikasi dapat dilihat melalui nilai Tm (Melting Temperature) pada melt curve. CP adalah jumlah siklus dimana sampel mulai terbaca di atas Arbitrary Fluorescence Level (AFL) yang menunjukkan awal mulainya fase pertumbuhan eksponensial. Oleh karena itu, semakin rendah nilai CP semakin tinggi jumlah DNA target. Tm adalah suhu dimana 50% bagian dari DNA telah terbuka menjadi untai tunggal. Nilai Tm tergantung dari jumlah basa A, G, T, dan C. Primer yang spesifik akan menghasilkan satu puncak Tm pada DNA target (Sambrook et al., 2001). UIN Syarif Hidayatullah Jakarta 41 a. Hasil Amplifikasi DNA Metode SYBR Green dengan Primer Sapi Amplifikasi DNA dilakukan dengan beberapa percobaan dengan cara memodifikasi program proses amplifikasi yang bertujuan untuk mendapatkan kondisi optimal dalam amplifikasi. Pada penelitian ini dilakukan modifikasi suhu annealing yaitu pada suhu 60oC, 62oC, 64oC, dan 65oC. Dari percobaan tersebut didapatkan hasil amplifikasi DNA yang terbaik yaitu pada suhu 65oC yang dapat dilihat pada gambar 4.4. Gambar 4.4. Kurva Amplifikasi dengan Metode SYBR Green Menggunakan Primer Sapi *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; dan CP = Crossing Point Pada gambar 4.4, nilai CP hasil amplifikasi DNA daging sapi dan DNA gelatin sapi masing-masing adalah 14,21 dan 20,01. Secara teoritis, seharusnya nilai CP pada DNA gelatin sapi lebih kecil dibandingkan DNA daging sapi karena konsentrasi DNA gelatin sapi yang digunakan lebih tinggi dibanding DNA daging sapi. Hal ini dapat dikarenakan DNA pada gelatin memiliki nilai kemurnian yang rendah yaitu 1,5 dimana terlalu banyak pengotor protein pada isolat DNA gelatin. Nilai kemurnia 1,5 memiliki perbandingan persentase protein dan asam nukleat yaitu 80% dan 20% (Glasel, 1995). Oleh karena itu jumlah DNA yang teramplifikasi pada DNA gelatin sapi lebih sedikit dibandingkan pada DNA daging sapi. Selain itu, pada gelatin banyak DNA yang terfragmentasi. Fragmentasi DNA dapat terjadi dikarenakan gelatin merupakan produk olahan dari kolagen. Semakin banyak jumlah DNA UIN Syarif Hidayatullah Jakarta 42 yang terfragmentasi, semakin kecil situs DNA target yang teramplifikasi (Edward et al., 1996). Berdasarkan hasil kurva amplifikasi yang terdapat pada gambar 4.4, DNA daging babi, DNA gelatin babi, dan NTC tidak mengalami kenaikan kurva amplifikasi. Hal ini menunjukkan bahwa metode SYBR Green dengan primer sapi dapat mengamplifikasi DNA sapi secara spesifik. Spesifisitas proses amplifikasi menggunakan SYBR Green dapat dianalisa melalui Melting peaks. Proses amplifikasi yang spesifik akan menghasilkan satu jenis puncak dengan nilai Tm yang sama. Pada gambar 4.5, DNA daging sapi dan DNA gelatin sapi memiliki nilai Tm yang sama yaitu pada suhu 78oC. Nilai Tm tersebut merupakan Tm DNA target amplifikasi. Namun, pada DNA daging sapi, muncul puncak Tm lainnya yaitu 81,78 yang menunjukkann telah terbentuknya produk amplifikasi yang nonspesifik. Pada daging babi, gelatin babi, dan NTC tidak muncul puncak Tm yang menunjukkan bahwa tidak terjadi mis-priming ataupun primer-dimer. Primer-dimer adalah terbentuknya struktur sekunder yang disebabkan karena menempelnya sesama primer sejenis ataupun primer yang tidak sejenis seperti antara primer forward dengan komplemen primer reverse. Sedangkan mis-priming adalah penempelan primer diluar sekuen DNA target (Ponchel, 2007). Gambar 4.5. Melting Peaks Hasil Amplifikasi dengan Metode SYBR Green Menggunakan Primer Sapi *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control UIN Syarif Hidayatullah Jakarta 43 Pada penelitian ini, proses amplifikasi dilakukan modifikasi suhu annealing karena terdapat perbedaan jauh nilai Tm teoritis pada primer forward sapi dan Tm pada primer reverse sapi sesuai dengan perhitungan pada lampiran 4, yakni 60oC dan 70oC. Nilai Tm primer akan mempengaruhi suhu annealing. Apabila suhu annealing yang terlalu rendah dan tidak tepat, dapat menyebabkan primer menempel dengan DNA secara tidak spesifik sehingga dapat menyebabkan kesalahan analisis. Menurut Scmittgen (2007) pasangan primer yang baik adalah kedua primer memiliki perbedaan nilai Tm tidak lebih dari 2oC. Berdasarkan Rahmawati (2012), optimasi suhu annealing dengan metode gradien PCR pada primer sapi yang sama dengan penelitian ini didapatkan hasil suhu optimal 60oC (Lampiran 5). Namun, setelah dilakukan amplifikasi dengan suhu annealing 60oC, terjadi amplifikasi pada DNA nontarget seperti DNA yang mengandung babi maupun NTC (No Template Control). Hal ini diduga terjadi karena suhu annealing yang digunakan terlalu rendah bagi primer reverse yang memiliki Tm 70oC, sehingga terjadi mis-priming atau primer-dimer. Oleh karena itu optimasi dilakukan dengan meninggikan suhu annealing. Setelah dilakukan amplifikasi pada suhu annealing 62oC, 64oC, dan 65oC, didapatkan suhu yang paling baik untuk amplifikasi DNA dengan metode SYBR Green pada primer sapi ini adalah 65oC. Pada suhu ini, hanya DNA yang mengandung sapi saja yang dapat teramplifikasi (Gambar 4.4). Sedangkan menggunakan suhu annealing 60oC, 62oC, dan 64oC terjadi amplifikasi pada semua DNA baik DNA yang mengandung sapi, mengandung babi, ataupun pada NTC (No Template Control) yang tidak mengandung DNA (Lampiran 7-9). b. Hasil Amplifikasi DNA Metode SYBR Green dengan Primer Babi Pada penelitian ini, amplifikasi DNA dilakukan dengan modifikasi waktu annealing dan extension dilakukan pada 10 detik (annealing) & 7 detik (extension); 20 detik & 25 detik; dan 20 detik & 30 detik. Hasil amplifikasi DNA yang paling baik yaitu dengan modifikasi waktu UIN Syarif Hidayatullah Jakarta 44 annealing 20 detik dan extension 25 detik. Hasil amplifikasi terlihat pada gambar 4.6. Gambar 4.6. Kurva Amplifikasi dengan Metode SYBR Green Menggunakan Primer Babi *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; dan CP = Crossing Point Pada gambar 4.6, nilai CP hasil amplifikasi DNA daging babi dan DNA gelatin babi masing-masing adalah 11,86 dan 43,71. Secara teoritis, seharusnya nilai CP pada DNA gelatin babi lebih kecil dibandingkan DNA daging babi, karena konsentrasi DNA gelatin babi yang digunakan lebih tinggi dibanding DNA daging babi. Hasil ini sesuai dengan yang terjadi pada amplifikasi metode SYBR Green dengan primer sapi yang telah diuraikan sebelumnya, yaitu DNA gelatin yang digunakan memiliki kemurnian yang rendah dan banyak yang terfragmentasi. Sehingga DNA gelatin babi pada hasil kurva amplifikasi memiliki CP yang lebih besar daripada DNA daging babi. Berdasarkan gambar 4.6, terjadi kenaikan kurva amplifikasi DNA daging sapi, DNA gelatin sapi, dan NTC pada siklus di atas 50 yang dimana seharusnya ketiga DNA tersebut tidak terjadi amplifikasi. Hal ini menunjukkan bahwa terjadi amplifikasi nonspesifik yang dapat disebabkan karena mis-priming atau primer-dimer seperti self dimer, cross dimer, atau pembentukan formasi hairpin. Terjadinya amplifikasi nonspesifik dianalisis melalui nilai Tm pada Melting Peaks seperti pada gambar 4.7. Pada kurva tersebut menunjukkan bahwa pada DNA daging UIN Syarif Hidayatullah Jakarta 45 sapi, gelatin sapi, NTC muncul puncak Tm yang seharusnya tidak ada. Puncak pada ketiga DNA tersebut merupakan hasil amplifikasi nonspesifik (Ponchel, 2007). Dari kurva amplifikasi dan Melting Peaks menunjukkan bahwa analisis DNA gelatin babi dan DNA gelatin sapi dengan metode Sybr Green pada primer babi yang di desain oleh Tanabe et. al., 2007 tidak dapat digunakan untuk analisis lebih lanjut pada sampel. Gambar 4.7 Melting Curve Sybr Green dengan menggunakan primer babi *Keterangan :DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control Pada primer babi tidak dilakukan modifikasi suhu annealing karena nilai Tm teoritis pada primer forward babi dan primer reverse babi memiliki nilai yang sama, yakni 66oC (lampiran 3). Berbeda dengan yang terjadi pada primer sapi yang memiliki perbedaan nilai Tm yang jauh dan memungkinkan terjadinya mis-priming atau primer-dimer karena tidak sesuainya suhu annealing. Oleh karena itu optimasi dilakukan hanya dengan modifikasi waktu annealing dan extension. Modifikasi waktu annealing dan extension diawali dengan waktu terendah yaitu 10 detik & 7 detik. Kurva yang dihasilkan pada modifikasi ini menunjukkan bahwa amplifikasi hanya terjadi pada DNA daging babi saja dan tidak mampu mengamplifikasi DNA gelatin babi (lampiran 11). Hal ini terjadi diduga karena waktu annealing dan extension yang terlalu cepat, sehingga proses amplifikasi tidak optimal. Selanjutnya dilakukan UIN Syarif Hidayatullah Jakarta 46 percobaan waktu annealing dan extension dengan durasi paling maksimal yaitu pada 20 detik & 30 detik, diharapkan mendapatkan hasil amplifikasi yang maksimal, sebagaimana yang berhasil dilakukan pada primer sapi. Namun hasil percobaan menunjukkan terjadi amplifikasi pada DNA dagang sapi, DNA gelatin sapi, dan NTC yang seharusnya tidak terjadi amplifikasi (lampiran 12). Terbentuknya produk amplifikasi yang nonspesifik diduga terjadinya amplifikasi dikarenakan terbentuknya primer-dimer atau mis-priming. Oleh karena itu diputuskan untuk menurunkan waktu extension menjadi 25 detik untuk percobaan selanjutnya. Pada percobaan dengan waktu annealing 20 detik dan extension 25 detik, didapatkan hasil kurva amplifikasi yang lebih baik dibandingkan dengan waktu 20 detik & 30 detik. Pada percobaan ini, di siklus 45, DNA gelatin babi teramplifikasi tanpa diikuti teramplifikasinya DNA daging sapi, DNA gelatin sapi, dan NTC. Namun, hasil amplifikasi DNA gelatin babi belum terbentuk kurva Plateau yang menunjukkan bahwa proses amplifikasi belum selesai. Oleh karena itu, proses amplifikasi dilanjutnya sampai didapatkan kurva yang plateau. Pada siklus 60, DNA gelatin sapi sudah membentuk kurva amplifikasi yang plateau. Akan tetapi penambahan siklus ini, juga menyebabkan teramplifikasinya DNA yang tidak mengandung babi, yaitu DNA daging sapi, DNA gelatin sapi, dan NTC yang sebelumnya tidak teramplifikasi (gambar 4.6). Peningkatan kurva amplifikasi pada DNA daging sapi, DNA gelatin sapi, dan NTC disebabkan karena terjadinya amplifikasi yang nonspesifik seperti mispriming atau primer-dimer. 4.3.2 Hasil Amplifikasi DNA Gelatin Sapi dan Gelatin Babi Menggunakan Real Time PCR dengan Metode Hydrolysis Probe a. Hasil Amplifikasi DNA Gelatin dengan Metode Hydrolysis Probe Menggunakan Primer Sapi Proses amplifikasi DNA dengan metode Hydrolysis Probe dilakukan dalam tiga tahap, yaitu tahap inisial denaturasi, tahap amplifikasi, dan tahap pendinginan. Konsentrasi DNA daging sapi; DNA UIN Syarif Hidayatullah Jakarta 47 daging babi; DNA gelatin sapi; dan DNA gelatin babi yang digunakan dalam proses amplifikasi masing-masing adalah 8,59 ng/μL; 8,0 ng/μL; 19,38 ng/μL; dan 13,63 ng/μL. Metode analisis menggunakan Hydrolysis Probe tidak dilakukan optimasi suhu annealing terlebih dahulu. Suhu annealing yang digunakan dipilih berdasarkan hasil optimasi yang telah dilakukan Rahmawati (2012), yaitu 60oC. Hasil amplifikasi DNA gelatin sapi dan DNA gelatin babi dengan primer sapi dapat dilihat melalui gambar 4.8. dibawah ini. Gambar 4.8 Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer-Probe Sapi *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; dan CP = Crossing Point Pada gambar 4.8, daging sapi memberikan nilai CP lebih awal yaitu pada 16,49 meskipun konsentrasi DNA daging sapi yang digunakan hanya 8,59 ng/μL. Sedangkan gelatin sapi yang memiliki konsentrasi lebih tinggi yaitu 19,38 ng/μL memberikan nilai CP lebih lama yaitu pada 29,41. Secara teoritis, seharusnya nilai CP pada DNA gelatin sapi lebih kecil dibandingkan DNA daging sapi. Hal ini sejalan dengan kurva yang dihasilkan metode SYBR Green yaitu disebabkan karena kemurnian DNA gelatin yang rendah dan DNA gelatin yang banyak terfragmentasi (Glasel, 1995; Edward et al., 1996). Spesifisitas amplifikasi dengan menggunakan Hydrolysis Probe tidak dapat dianalisa melalui Melting Peaks, karena perbedaan prinsip pewarnaan fluoresens antara metode SYBR Green dengan Hydrolysis UIN Syarif Hidayatullah Jakarta 48 Probe. Spesifisitas metode Hydrolysis Probe dilihat berdasarkan kenaikan kurva amplifikasi pada kontrol negatif. Pada gambar 4.8, DNA daging babi, DNA gelatin babi, dan NTC yang merupakan kontrol negatif, tidak terjadi kenaikan kurva amplifikasi. Hal ini menunjukkan bahwa primer-probe sapi dapat mengamplifikasi DNA sapi secara spesifik. Oleh karena hal tersebut, metode ini dapat digunakan untuk analisa lebih lanjut pada sampel yang mengandung gelatin. b. Hasil Amplifikasi DNA Gelatin dengan Metode Hydrolysis Probe Menggunakan Primer Babi Konsentrasi DNA daging sapi; DNA daging babi; DNA gelatin sapi; dan DNA gelatin babi yang digunakan dalam proses amplifikasi masing-masing adalah 8,59 ng/μL; 8,0 ng/μL; 19,38 ng/μL; dan 13,63 ng/μL. Suhu annealing yang digunakan dipilih berdasarkan hasil optimasi yang telah dilakukan Rahmawati (2012), yaitu 60oC (Lampiran 5). Hasil amplifikasi DNA gelatin sapi dan DNA gelatin babi dengan metode Hydrolysis Probe menggunakan primer babi dapat dilihat melalui gambar 4.9 dibawah ini. Gambar 4.9. Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer-Probe Babi *Keterangan: DS= Daging sapi; GS= Gelatin Sapi; GB= Gelatin Babi; DB= Daging Babi; NTC = No Template Control; dan CP = Crossing Point Pada gambar 4.9, daging babi memberikan nilai CP lebih awal yaitu pada 16,93, diikuti dengan gelatin babi dengan nilai CP 43,67. UIN Syarif Hidayatullah Jakarta 49 Secara teoritis, seharusnya nilai CP pada DNA gelatin babi lebih kecil dibandingkan DNA daging babi dimana konsentrasi DNA gelatin yang digunakan dalam proses amplifikasi lebih tinggi dibandingkan DNA daging. Hal ini dapat disebabkan kemurnian DNA gelatin yang rendah dan DNA gelatin yang banyak terfragmentasi (Glasel, 1995; Edward et al., 1996). Hasil kurva amplifikasi pada gambar 4.9 menunjukkan metode analisa ini cukup spesifik untuk deteksi DNA babi yang ditandai dengan tidak terjadinya amplifikasi pada DNA daging sapi, gelatin sapi, dan NTC. Oleh karena hal tersebut, metode ini dapat digunakan untuk analisa lebih lanjut pada sampel yang mengandung gelatin. UIN Syarif Hidayatullah Jakarta 50 BAB 5 KESIMPULAN DAN SARAN 5.1 Kesimpulan Dari hasil penelitian yang telah dilakukan, dapat diambil kesimpulan sebagai berikut: a. Analisis DNA gelatin sapi dan DNA gelatin babi dengan metode SYBR Green menghasilkan amplifikasi yang nonspesifik, sehingga metode ini kurang baik digunakan sebagai metode analisis DNA gelatin sapi dan DNA gelatin babi khususnya pada produk yang mengandung gelatin. b. Analisis DNA gelatin sapi dan DNA gelatin babi dengan metode Hydrolysis Probe dapat mengamplifikasi DNA gelatin sapi dan DNA gelatin babi secara spesifik. Sehingga metode ini dapat digunakan sebagai metode analisis DNA gelatin sapi dan DNA gelatin babi khususnya pada produk yang mengandung gelatin. 5.2 Saran a. Diharapkan melakukan optimasi metode ekstraksi DNA pada gelatin agar mendapatkan DNA dengan nilai kemurnian 1,8 sampai 2,0. b. Diharapkan untuk melakukan optimasi lebih lanjut dalam analisis DNA gelatin sapi dan DNA gelatin babi untuk mendapatkan kondisi optimal dalam mendeteksi, sehingga dapat dijadikan sebagai metode untuk pengujian kehalalan pada produk yang mengandung gelatin. 50 UIN Syarif Hidayatullah Jakarta 51 DAFTAR PUSTAKA Aida, A. A., Che Man, Y. B., Raha, A. R., & Son, R. 2007. Detection of pig derivatives in food products for halal authentication by polymerase chain reaction–restriction fragment length polymorphism. Journal of the Science of Food and Agriculture. Vol. 87. Hal: 569–572. Ansel, Howard C. 2005. Pengantar Bentuk Sediaan Farmasi. Edisi keempat. Jakarta: UI-Press. Arya, M., Iqbal S. Shergill, Magali Wiliamson, Lyndon Gommersall, Neehar Arya, dan Hitendra RH Patel. 2005. Basic Principle of Real-Time Quantitative PCR. Future Drugs Review Article. Vol. 5 No. 2, Hal: 209– 219 Atto. 2008. ATTO Printgraph AE-6933 Operation Manual. Tokyo: ATTO Corporation. Azira, Nur T., Amin, I. dan Che Man, Y. B. 2012. Differentiation of bovine and porcine gelatins in processed products via Sodium Dodecyl SulphatePolyacrylamide Gel Electrophoresis (SDS-PAGE) and principal component analysis (PCA) techniques. International Food Research Journal. Vol. 19 (3). Hal: 1175-1180 Balti, Rafik., J. Mourad, A. Sila, N. Souissi, N. Nedjar-Arroume, D. Guillochon, M. Nasri. 2010. Extraction and Functional Properties f Gelatin From The Skin of Cuttlefish (Sepia Officinalis) usiing Smooth Hound Crude Acid Protease-Aided Process. Artikel pada PressFood Hydrocolloids. BioDrop. 2012. Quick Start Guide. http://www.biodrop.co.uk. Diakses pada tanggal 30 September 2014 pukul 20.30. Bio-Rad. 2012. Protocols: Nucleic Acid Electrophoresis. Buletin 6230 Rev A. Bio-Rad Laboratories, Inc. Cai, H., X. Gu, M.S. Scanlan, D.H. Ramatlapeng dan C.R. Lively. 2012. Realtime PCR assays for detection and quantitation of porcine and bovine DNA in gelatin mixtures and gelatin capsules. Journal of Food Composition and Analysis. Vol. 25. Hal: 83-87. Campbell, Nail A., Reece, Jane B., Mitchell, Lawrence G. 2002. Biologi. Edisi Kelima Jilid 1, alih bahasa Rahayu Lestari. Jakarta: Erlangga Carr, J. M., K. Sufferling, dan J. Poppe. 1995. Hydrocolloids and their use in the confectionery Industry. Journal of Food Technology. Vol. 32 (7). Hal: 41-42 UIN Syarif Hidayatullah Jakarta 52 Chee Tan, Siun, dan Beow Chin Yiap. 2009. DNA, RNA, and Protein Extraction: The Past and The Present. Journal of Biomedicine and Biotechnology. Volume 2009, Article ID 574398. Demirhan, Y., P. Ulca dan H.Z. Senyuva. 2012. Detection of porcine DNA in gelatine and gelatine-containing processed food products-Halal/Kosher authentication, Meat Science. Vol. 90. Hal: 686-689. Edward, M. G., Ann Bickel dan Paul Weihs. 1996. Effect of highly fragmented DNA on PCR. Nucleic Acids Research, Vol. 24, No. 24. Oxford University Press. Erwanto, Y., Abidin, M.Z., Sismindari, dan Rohman, A. 2012. Pig Species Identification in Meatballs Using Polymerase Chain ReactionRestriction Fragment Length Polymorphism for Halal Authentication. International Food Research Journal 19 (3): 901-906. Gaffar, Shabrani, M.Si. 2007. Buku Ajar Bioteknologi Molekul. Bandung: FMIPA-Universitas Padjajaran Gjerse, D. T., L. Hoang, and D. Hornby. 2009. RNA Purification and Analysis: Sample Preparation, Extraction, Chromatography. Edisi ke-1. Germany: Wiley VCH,Weinheim. Guillen, Gómez M.C., Pérez-Mateos, M., Gómez-Estaca, J., López-Caballero, E., Giménez, B., & Montero, P. (2009). Fish gelatin: a renewable material for the development of active biodegradable films. Trends in Food Science and Technology. Vol. 20. Hal: 3-16. Hartati, Yeni W., Maksum, Iman P. 2010. Amplifikasi 0,4 kb Daerah D-Loop DNA Mitokondria dari Sel Epitel Rongga Mulut untuk Keperluan Forensik. Bandung: FMIPA-Universitas Padjadjaran. Hashim, D.M., Y.B.C. Man, R. Norakasha, M. Shuhaimi, Y. Salmah dan Z.A. Syahariza. 2010. Potential use of Fourier transform infrared spectroscopy for differentiation of bovine and porcine gelatin. Food Chemistry. Vol. 118. Hal: 856-860. Hidaka, S. dan S.Y. Liu. 2003. Effects of gelatins on calcium phosphate precipitation: A possible application for distinguishing bovine bone gelatin from porcine skin gelatin. Journal of Food Composition and Analysis. Vol. 16. Hal: 477-483. Hinterwaldner, R. 1977. Technology of gelatin manufacture. The Science and Technology of Gelatin. London: Academic Press. Hal: 315–361. Horton, H. Robert, Laurence A. Moran, K. Gray Scrimgeour, Marc D. Perry, J. David Rawn. 2006. Principles of Biochemistry. Edisi Keempat. USA: Pearson Education, Inc. UIN Syarif Hidayatullah Jakarta 53 Ikada, Y. 2002. Biological Materials. Integrated Biomaterials Science. USA: Academic /Plenium Publishers. Jamaludin, M. A., Mohd. Anuar Ramli, Dzulkifly Mat Hashim. 2012. Fiqh Istihalah: Integration of Science and Islamic Law. Revelation and Science. Vol. 02, No.02 Jannah, A. 2008. Gelatin: Tinjauan Kehalalan dan Alternatif Produksi. Malang: UIN-Malang Press Jones, B.E., 2004. The history of The Medicinal Capsule. Dalam: Podczeck, F., Jones, B.E. (Eds.). Pharmaceutical Capsules. USA: Pharmaceutical Press. Hal: 1–22. Koolman, Jan dan Klaus-Heinrich Roehm. 2005. Color Atlas of Biochemistry. Edisi Kelima. Germany: Georg Thieme Verlag Lelana, Neo Endra, Sutarno dan Nita Etikawati. 2003. Identifikasi Polimorfisme pada Fragmen ND-5 DNA Mitokondria Sapi Benggala dan Madura dengan Teknik PCR-RFLP. Biodiversitas. Vol. 4 (1). Surakarta: FMIPAUniversitas Sebelas Maret Lewis, Matt. 2001. Agarose Gel Electrophoresis (Basic Method). http://www.methodbook.net/. Diakses pada tanggal 1 Oktober 2014 pukul 14.15. LPPOM MUI. 2008. Halal Menentramkan Umat. Jurnal Halal No.72. Morrison TB, Weis JJ, dan Wittwer CT. 1998. Quantification of Low-copy Transcripts Bay continuous SYBR Green I Monitoring During Amplification. BioTechniques 24. Hal: 954–958. Muladno. 2010. Teknologi Rekayasa Genetika. Edisi Kedua. Bogor: IPB Press Nemati, M., M.R. Oveisi, H. Abdollahi dan O. Sabzevari. 2004. Differentiation of bovine and porcine gelatins using principal component analysis. Journal of Pharmaceutical and Biomedical Analysis. Vol. 34. Hal: 485-492. Nhari, R.M.H.R., A. Ismail and Y.B. Che Man. 2012. Analytical methods for gelatin differentiation from bovine and porcine origins and food products. Journal of Food Science. Vol. 71 Hal: 42-46. Passarge, Ebenhard. 2007. Color Atlas of Genetics. Edisi Ketiga. New York: Thieme Purves, William K., David Sadava., Gordan H. Orians., & Craig Heller. 2004. Life: The Science of Biology. Edisi Ketujuh. United States of America: Sinauer Associates and W. H. Freeman UIN Syarif Hidayatullah Jakarta 54 Putra, Suhartono. 1999. Biologi Molekuler Kedokteran, editor: Suhartono Taat Putra. Surabaya : Airlangga University Press. Rabadiya, Bhavisha dan Paresh Rabadiya. 2013. Capsule Shell Material From Gelatin To Non Animal Origin Material. International Journal of Pharmaceutical Research and Bio Science (IJPRBS). Vol. 2(3). Hal: 4271 Rachmawati, Putri. 2012. Analisis Cemaran Daging Babi pada Produk Dendeng Sapi yang Beredar di Wilayah Ciputat dengan Metode Amplifikasi DNA Menggunakan Real-Time PCR. SKRIPSI. Program Studi Farmasi. Fakultas Kedokteran dan Ilmu Kesehatan. Universitas Islam Negeri Syarif Hidayatullah Jakarta. Raven, Peter H., George B. Johnson, Kenneth A. Mason, Jonathan B. Losos, Susan R. Singer. 2008. Biology. Edisi Kesembilan. New York: McGrawHill Companies, Inc. Republika. 2013. Ini Pernyataan Menkes Soal Obat Halal yang Disesalkan MUI via www.republika.co.id. Diakses pada tanggal 28 Desember 2013 Republika. 2014. MUI: Menkes Langgar Hak Asasi Konsumen via http://www.republika.co.id. Diakses pada tanggal 28 Desember 2013 Rochea. 2012. High Pure PCR Template Preparation Kit. http://www.rocheapplied-science.com. Diakses pada 13 Juni 2014 pukul 17.05 Rocheb. 2012. High Pure Technology and Silica Adsorption Kits. http://lifescience.roche.com. Diakses pada 22 September 2014 pukul 04.17 Rochec. 2005. LightCycler® 480 DNA SYBR Green I Master. www.rocheappliedscience.com. Diakses pada 2 September 2014 pukul 10.30 Roched. 2008. LightCycler® 480 Probes Master. www.rocheapplied-science.com. Diakses pada 2 September 2014 pukul 12.30 Rochee. 2008. The LightCycler® 480 Instrument Operator’s Manual. www.rocheapplied-science.com. Diakses pada 10 Juni 2014 pukul 15.05 Rowe, Raymond C., Paul J. Sheskey, dan Marian E Quinn. 2009. Handbook of Pharmaceutical Excipients. Edisi Keenam. London: Pharmaceutical Press. Riaz, M.N. dan M.M. Chaudry. 2004. Halal Food production. USA: CRC Press. Sahilah, A. M., Mohd. Fadly, A. S. Norrakiah, A. Aminah, Wan Aida, W. M. Ma’aruf, A. G dan Mohd. Khan, A. 2012. Halal market surveillance of soft and hard gel capsules in pharmaceutical products using PCR and UIN Syarif Hidayatullah Jakarta 55 southern-hybridization on. International Food Research Journal 19(1): 371-375. Sambrook, J., E.F. Fritsch dan T. Manuatis. 2001. Molecular Cloning, A Laboratory Manual. Edisi ketiga. New York: Cold Spring Harbour Lab. Press. Schrieber, R., & Gareis, H. 2007. Gelatine Handbook: Theory dan Industrial Practice. Jerman: Wiley VCH Verlag GmbH dan Co. KGaA Sharma, Himani et al,. 2005. Mutations in the mitochondrial DNA D-loop region are frequent in cervical cancer. Cancer Cell International 2005, 5:34. Smith, Cindy J. dan A. Mark Osborn. 2008. Advantages and Limitations of Quantitative PCR (Q-PCR)-based Approaches in Microbial Ecology. Federation of European Microbiological Societies UK: Blackwell Publishing Ltd. Stanley, J.P. 1976. Capsule: The Theory and Practice of Industrial Pharmacy. Diedit oleh L. Lachman, H.A. Lieberman, dan J.L. Kanig. Philadelphia, Lea & Febiger. Hlm 404 - 420. Stansfield, William D., Jaime S.Colomé., Raúl J.Cano. 2003. Shaum’s Easy Outlines: Molecular and Cell Biology. USA: McGraw-Hill Companies, Inc. Vaerman, J.L., P. Saussoy, I. Ingargiola. 2004. Evaluation of Real-Time PCR Data. Belgium : Cliniques Saint Luc, Bruxelles Venien, A. dan D. Levieux. 2005. Differentiation of gelatins using polyclonal antibodies raised against tyrosylated bovine and porcine gelatins. J. Immunoassay Immunochem., 26, 215-229. Widyaninggar, A., Triwahyudi., Triyana, K., dan Ab.Rohman. 2012. Differentiation between porcine and bovine gelatin in commercial capsule shells based on amino acid profiles and principal component analysis. Indonesian J.Pharm Vol. 23 No. 2: 96-101 ISSN-p : 0126-1037 Westermeier, 2004. Electrophoresis in Practice: A Guide to Theory and Practice. New-Jersey: John Wiley & Sons inc. Wink, M. 2006. An Introduction to Molecular Biotechnology: Molecular Fundamentals, Methods and Application in Modern Biotechnology, Germany: Wiley-VCH,Weinheim. Yuwono, Triwibowo. 2009. Biologi Molekular. Jakarta : Erlangga Zhang, G.F., T. Liu, Q. Wang, J.D. Lei, G.H. Ma dan Z.G. Su. 2008. Identification of marker peptides in digested gelatins by high UIN Syarif Hidayatullah Jakarta 56 performance liquid chromatography/ mass spectrometry. Chin. J. Anal. Chem., 36, 1499-1504. Zhou, P. dan Regenstein, J. M. 2005. Effects of Alkaline and Acid Pretreatments on Alaska Pollock Skin Gelatin Extraction. Journal of Food Science, 70(6): C392–C396. Zyskind, J. W. dan S. I. Bernstain. 1992. Recombinant DNA Manual. Academic Press, Inc. California UIN Syarif Hidayatullah Jakarta 57 Lampiran 1. Kerangka Penelitian Daging Segar Daging Sapi Gelatin Standard Daging Babi Gelatin Sapi Gelatin Babi Ekstraksi dan isolasi DNA Analisa DNA hasil isolasi dengan Elektroforesis Agarosa dan Spektrofotometri tidak DNA terisolasi? ya Isolat DNA Amplifikasi dengan Real Time PCR Metode SYBR Green Metode Hydrolysis Probe Analisa Hasil Kesimpulan UIN Syarif Hidayatullah Jakarta 58 Lampiran 2. Perhitungan pembuatan larutan primer dan probe 1. Membuat larutan primer dan probe 10 μM dari larutan induk 100 μM V1 . M1 = V2 . M2 X . 100 μM = 100 μL . 10 μM X = 1000 100 = 10 μL Maka, 10 μL diambil dari masing-masing primer 100 μM dan di add 90 μL PCR water grade 2. Mengetahui volume primer dan probe yang diambil dari larutan primer dan probe 10 μM dalam pembuatan master mix a) Rekomendasi konsentrasi untuk primer pada SYBR Green mastermix adalah 0.3-1 μM (Roche), dipilih konsentrasi 0.5 μM untuk tiap primer. V1 . M1 = V2 . M2 X . 10 μM = 20 μL . 0,5 μM X = 10 10 = 1 μL Maka, diambil 1 μL dari larutan primer konsentrasi 10 μM b) Rekomendasi konsentrasi untuk primer pada Hydrolysis Probe mastermix adalah 0.3-1 μM (Roche), dipilih konsentrasi 0.8 μM untuk tiap primer. V1 . M1 = V2 . M2 X . 10 μM = 20 μL . 0,8 μM X = 16 10 = 1,6 μL Maka, diambil 1,6 μL dari larutan primer konsentrasi 10 μM UIN Syarif Hidayatullah Jakarta 59 c) Rekomendasi konsentrasi untuk primer pada Hydrolysis Probe adalah 0.05-0.2 μM (Roche), dipilih konsentrasi 0.2 μM untuk tiap primer. V1 . M1 = V2 . M2 X . 10 μM = 20 μL . 0,2 μM X=4 10 = 0,4 μL Maka, diambil 0,4 μL dari larutan primer konsentrasi 10 μM UIN Syarif Hidayatullah Jakarta 60 Lampiran 3. Perhitungan Tm (Melting Temperature) Primer Rumus Tm = 2oC (A+T) + 4oC (G+C) Primer Sapi Primer Babi Primer sapi forward Primer babi forward Tm = 2oC (2+8) + 4oC (2+8) = 60oC Primer sapi reverse Tm = 2oC (5+8) + 4oC (6+5) Tm = 2oC (6+5) + 4oC (4+7) = 66oC Primer babi reverse Tm = 2oC (8+7) + 4oC (6+3) = 70oC = 66oC Tm rata-rata Primer sapi: Tm rata-rata Primer babi: Tm = (60 + 70) / 2 = 65oC Ta* = 60oC Tm = (66 + 66) / 2 = 66oC Ta* = 61oC *) Suhu annealing yang digunakan biasanya 5oC di bawah Tm (Muladno, 2010) UIN Syarif Hidayatullah Jakarta 61 Lampiran 4. Campuran Reaksi Mastermix untuk Amplifikasi DNA Tabel 7. Campuran reaksi SYBR Green mastermix Konsentrasi Larutan Induk Jumlah yang digunakan Primer Forward 0,5 μM 10 μM 1 μL Primer Reverse 0,5 μM 10 μM 1 μL LightCycler® 480 SYBR Green I - - 10 μL ddH2O - - 3 μL DNA template - - 5 μL 20 μL Total volume reaksi Tabel 8. Campuran reaksi Hydrolysis Probe mastermix Konsentrasi Larutan Induk Jumlah yang digunakan Primer Forward 0,8 μM 10 μM 1,6 μL Primer Reverse 0,8 μM 10 μM 1,6 μL Probe 0,2 μM 10 μM 0,4 μL LightCycler® 480 Probe Master - - 10 μL ddH2O - - 1,4 μL DNA template - - 5 μL Total volume reaksi 20 μL UIN Syarif Hidayatullah Jakarta 62 Lampiran 5. Hasil Optimasi Suhu Annealing dengan Metode Gradien PCR Gambar 1. Elektroforesis produk PCR hasil optimasi suhu annealing primer sapi pada DNA daging sapi dengan metode gradien PCR (Sumber: Rahmawati, 2012) Gambar 2. Elektroforesis produk PCR hasil optimasi suhu annealing primer babi pada DNA daging babi dengan metode gradien PCR (Sumber: Rahmawati, 2012) UIN Syarif Hidayatullah Jakarta 63 Lampiran 6. Spesifikasi kit ekstraksi dan isolasi DNA: High Pure PCR Template Preparation Kit (Roche) Tabel 9. Spesifikasi kit ekstraksi (Sumber: Rochea, 2012) No. Warna Tutup Label Vial Vial 1 Putih Kandungan Tissue Lysis Buffer 4 M urea, 200 mM Tris, 20 mM NaCl, 200 nM EDTA. pH 7.4. 2 Hijau Binding Buffer 6 M guannidine-HCl, 10 mM urea, 10 mM Tris-HCl, 20 % Triton X-100 (v/v). pH 4.4 3 Merah muda Proteinase K Proteinase K (rekombinan, PCR grade) Hitam 5M guanidine-HCl, 20 mM Inhibitor Removal Buffer Tris-HCl, 36% etanol absolut (v/v). pH 6.6 4a 4 Biru 5 Tidak Berwarna Elution Buffer (Bening) - - Washing Buffer - - 20 mM NaCl, 2 mM TrisHCl, 80% etanol absolut (v/v). pH 7.5 10 mM Tris-HCl, pH 8.5 High Pure Filter Tube Tube berbahan polypropylene dengan filter yang terbuat dari kapas fiber glass. Dapat menampung 700 μl volume sampel. Collection Tube Tube berbahan polypropylene. Dapat menampung 2 ml volume sampel. UIN Syarif Hidayatullah Jakarta 64 Lampiran 7. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 60oC *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 65 Lampiran 8. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 62oC *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 66 Lampiran 9. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 64oC *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 67 Lampiran 10. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Sapi dengan Suhu Annealing 65oC *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 68 Lampiran 11. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Babi dengan Waktu Annealing 10 detik dan Waktu Extension 7 detik *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 69 Lampiran 12. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Babi dengan Waktu Annealing 20 detik dan Waktu Extension 30 detik *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 70 Lampiran 13. Hasil Kurva Amplifikasi dan Melting Peaks dengan Metode SYBR Green Menggunakan Primer Babi dengan Waktu Annealing 20 detik dan Waktu Extension 25 detik *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point; Tm = Melting Temperature UIN Syarif Hidayatullah Jakarta 71 Lampiran 14. Hasil Kurva Amplifikasi dengan Metode Hydrolysis Probe Menggunakan Primer Sapi *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point Lampiran 15. Hasil Kurva Amplifikasi dengan metode Hydrolysis Probe Menggunakan Primer Sapi *Keterangan : DS = Daging sapi; GS = Gelatin Sapi; GB = Gelatin Babi; DB = Daging Babi; NTC = No Template Control; CP = Crossing Point UIN Syarif Hidayatullah Jakarta 72 Lampiran 16. Gambar alat alat yang digunakan dalam penelitian Gambar 1. Mikropipet (1) Vol 1000 ul; (2) Vol 200 ul; (3) Vol 20 ul; (4) Vol 10 ul Gambar 2. Mikrotips Gambar 3. Microsentrifuge tube Gambar 4. Satu set alat elektroforesis (1) Chamber; (2) Gel Caster; (3) Comb; (4) Power Supply Gambar 5. Gel Documentation Gambar 6. Microsentrifugator UIN Syarif Hidayatullah Jakarta 73 Gamabar 7. Vortex Gambar 8. Water Bath Gambar 9. Kit Ekstraksi Gambar 10. Filter tube dan Collection tube Gambar 11. Spektrofotometri DNA Gambar 12. Multiwell Plates Gambar 13. Sealing Foil Gambar 14. Real Time PCR UIN Syarif Hidayatullah Jakarta