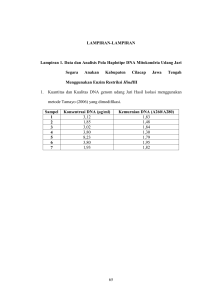
SKRIPSI KAJIAN METODE DETEKSI BAKTERI PATOGEN
advertisement

SKRIPSI KAJIAN METODE DETEKSI BAKTERI PATOGEN PENYEBAB PENYAKIT ASAL PANGAN DI PUSAT RISET OBAT DAN MAKANAN BADAN POM RI Oleh : TRI OCTORA ANGELIA F24051927 2009 DEPARTEMEN ILMU DAN TEKNOLOGI PANGAN FAKULTAS TEKNOLOGI PERTANIAN INSTITUT PERTANIAN BOGOR BOGOR KAJIAN METODE DETEKSI BAKTERI PATOGEN PENYEBAB PENYAKIT ASAL PANGAN DI PUSAT RISET OBAT DAN MAKANAN BADAN POM RI SKRIPSI Sebagai salah satu syarat untuk memperoleh gelar SARJANA TEKNOLOGI PERTANIAN Pada Departemen Ilmu dan Teknologi Pangan Fakultas Teknologi Pertanian Institut Pertanian Bogor Oleh : TRI OCTORA ANGELIA F24051927 2009 DEPARTEMEN ILMU DAN TEKNOLOGI PANGAN FAKULTAS TEKNOLOGI PERTANIAN INSTITUT PERTANIAN BOGOR BOGOR Tri Octora Angelia. F24051927. Kajian Metode Deteksi Bakteri Patogen Penyebab Penyakit Asal Pangan di Pusat Riset Obat dan Makanan Badan POM RI. Di bawah bimbingan Ratih Dewanti-Hariyadi dan Winiati P. Rahayu. RINGKASAN Mikroorganisme sering mencemari pangan dan deteksinya dapat dilakukan dengan metode konvensional dan metode cepat. Metode konvensional memerlukan waktu yang lama karena berbasiskan morfologi dan sifat biokimiawi, dan kadangkadang memerlukan konfirmasi berdasarkan uji serologi. Metode cepat yang berbasiskan Polymerase Chain Reaction (PCR) dapat mengidentifikasi dengan cara memperbanyak DNA target pada patogen. Perbanyakan DNA tersebut dapat berlangsung dengan adanya fragmen oligonukleotida yang komplementer terhadap DNA target tersebut (primer), enzim DNA polimerase, deoksinukleotida, Mg2+, buffer dan thermal cycler. Deteksi patogen dalam pangan juga dilakukan oleh Pusat Riset Obat dan Makanan, Badan POM RI. Penelitian ini bertujuan untuk mempelajari uji serologi untuk konfirmasi Salmonella spp., metode deteksi berbasiskan DNA terhadap B. cereus dalam pangan, dan menetapkan limit deteksi B. cereus dalam nasi goreng. Konfirmasi Salmonella spp. dilakukan dengan uji serologi antigen O dengan Slide Agglutination Test (SAT). Reaksi positif SAT adalah terbentuknya aglutinasi karena antigen dan antibodi saling mengkompleks. Deteksi B. cereus dengan amplifikasi gen penyandi enterotoksin T menggunakan real-time PCR. Isolat DNA yang digunakan sebagai DNA target dalam amplifikasi diperoleh dari tiga metode isolasi, yaitu metode pendidihan, metode dengan pelarut fenol:kloroform, dan metode dengan kit komersial. Isolat DNA yang sudah diamplifikasi, kemudian ditetapkan limit deteksinya berdasarkan threshold cycle (Ct) amplikon. Uji serologi terhadap sepuluh isolat Salmonella spp. menunjukkan bahwa masing-masing isolat memiliki antigen O dan motilitas yang berbeda-beda. Hal ini memungkinkan spesies Salmonella spp. tersebut diklasifikasikan ke dalam subspesies yang lebih spesifik. Isolasi DNA dengan menggunakan metode ekstraksi fenol:kloroform menghasilkan isolat DNA yang lebih murni. Hal tersebut ditunjukkan oleh puncak kurva peleburan produk amplifikasi DNA B. cereus terpusat pada suhu 81.5°C. Limit deteksi B. cereus dalam nasi goreng adalah 1.3 CFU/ml. Optimasi suhu annealing, konsentrasi primer, dan konsentrasi Mg2+ perlu dilakukan untuk memperoleh hasil optimal dalam proses amplifikasi PCR. INSTITUT PERTANIAN BOGOR FAKULTAS TEKNOLOGI PERTANIAN KAJIAN METODE DETEKSI BAKTERI PATOGEN PENYEBAB PENYAKIT ASAL PANGAN DI PUSAT RISET OBAT DAN MAKANAN BADAN POM RI SKRIPSI Sebagai salah satu syarat untuk memperoleh gelar SARJANA TEKNOLOGI PERTANIAN Pada Departemen Ilmu dan Teknologi Pangan Fakultas Teknologi Pertanian Institut Pertanian Bogor Oleh: TRI OCTORA ANGELIA F24051927 Dilahirkan di Medan, 6 Oktober 1987 Tanggal lulus : 28 Agustus 2009 Menyetujui, Bogor, 7 September 2009 Dr. Ir. Ratih Dewanti-Hariyadi, MSc Dosen Pembimbing I Prof. Dr. Winiati P. Rahayu Dosen Pembimbing II / Lapang Mengetahui, Dr. Ir. Nurheni Sri Palupi, Msi A.n. Ketua Departemen Ilmu dan Teknologi Pangan Sekretaris RIWAYAT HIDUP Penulis dilahirkan pada tanggal 6 Oktober 1987 di Medan dari pasangan Hisar P. Samosir dan Tiodor Agustina Saragi. Kedua orangtua memberi penulis seorang kakak lelaki, dr. Andre Somba Gugun Samosir dan seorang kakak perempuan Andika Putri Listiawati, STP. Pendidikan penulis dimulai dari TK Anging Mamiri, dilanjutkan ke SD Negeri Setia Mulya 1, SLTP Negeri 1 Cimahi, dan SMA Negeri 2 Cimahi. Kemudian penulis melanjutkan studi di Institut Pertanian Bogor Fakultas Teknologi Pertanian dengan mayor Ilmu dan Teknologi Pangan dan minor Perkembangan Anak. Selama menempuh pendidikan di Institut Pertanian Bogor, penulis mengimbangi studi dengan kegiatan ekstrakurikuler, antara lain: menyuluh para pedagang pangan dan warga sekitar kampus IPB mengenai keamanan pangan, mengepalai Departemen Peduli Pangan Indonesia dalam Himpunan Profesi HIMITEPA, mengurus komisariat Himpunan Mahasiswa Peduli Pangan (HMPPI), mengikuti Paduan Suara Fakultas, dan mengambil peran dalam sejumlah kegiatan Persekutuan Mahasiswa Kristen. Kegemaran penulis mempelajari hal-hal yang baru dan menciptakan hubungan sosial dengan orang lain dapat dipenuhi melalui kegiatakegiatan tersebut. KATA PENGANTAR Puji dan syukur penulis panjatkan kepada Tuhan yang Maha Pengasih. Kebaikan-Nya dan kekuasaan-Nya telah memberi penulis kemampuan untuk menyelesaikan skripsi ini. Penulis juga ingin menyampaikan ungkapan terima kasih yang tulus kepada: 1. Mama dan Papa tersayang, Abang Andre sebagai panutanku, Kakak Andika Putri sebagai pengaduanku. 2. Ibu Dr. Ir. Ratih Dewanti-Hariyadi, M.Sc sebagai pembimbing yang senantiasa menuntun dan mengarahkan penulis selama studi hingga memperoleh kelulusan ini. 3. Ibu Prof. Dr. Winiati P. Rahayu sebagai pembimbing yang memberi kesempatan magang bagi penulis di Pusat Riset Obat dan Makanan Badan POM RI dan memberi saran-saran untuk penelitian dan penyelesaian skripsi ini. 4. Ibu Siti Nurjanah, STP, M.Si sebagai penguji yang memberi masukan bagi penulis demi kelayakan skripsi ini. 5. Seluruh Dosen dan Staf Departemen Ilmu dan Teknologi Pangan (ITP) atas bantuan dan bimbingan yang diberikan kepada penulis selama menempuh pendidikan di Departemen ITP. 6. Saudara-saudara yang selalu mendukungku, Uda Ridwan beserta Inanguda, Abang Jon, Eda Ika beserta Nate dan Naomi, Nastry, dan Dian yang terkasih. Terima kasih atas doa kalian. 7. Golden Generation ITP 42 yang telah berjuang bersama-sama dalam menempuh studi di ITP. Yanka, Tiyu, Icha, Fahmi, Kamlit, Hesti, Galih, Dina, Ester, Tere senang dapat memiliki pengalaman dan mempelajari hal-hal baru bersama kalian. 8. Ntet, Nonk, Mpe, Ditol, Dini yang terus bersamaku dari awal berada di asrama IPB hingga studi kita untuk strata-1 ini berakhir. 9. Ibu Ida, Ibu Suci, Ibu Novi, Ibu Ning, Ibu Khusnul, Ibu Wiwi serta para staf Pusat Riset Obat dan Makanan Badan POM yang menjadi rekan sekaligus pembimbing teknis. i 10. Mike, Tiwi, Upik, dan Fauzan yang telah bersama-sama dengan penulis melaksanakan magang di Pusat Riset Obat dan Makanan, Badan POM. 11. Seluruh pihak yang telah membantu penulis hingga sampai di titik akhir Strata-1 ini. Penulis menyadari kekurangan ataupun kesalahan tak lekang dari skripsi ini. Oleh karena itu, penulis sangat menghargai dan mengharapkan kritik dan saran bagi skripsi ini. Semoga skripsi ini bermanfaat bagi kemajuan pendidikan dan perkembangan ilmu pengetahuan. Bogor, 7 September 2009 Penulis ii DAFTAR ISI Halaman KATA PENGANTAR ...................................................................................... i DAFTAR ISI ..................................................................................................... iii DAFTAR TABEL ............................................................................................ v DAFTAR GAMBAR ........................................................................................ vi DAFTAR LAMPIRAN .................................................................................... viii I. PENDAHULUAN ..................................................................................... 1 A. LATAR BELAKANG ............................................................................ 1 B. TUJUAN ................................................................................................ 4 C. MANFAAT ............................................................................................ 4 II. TINJAUAN PUSTAKA ............................................................................ 5 A. KEADAAN UMUM INSTANSI MAGANG ........................................ 5 1. Sejarah dan Perkembangan Instansi .................................................. 5 2. Lokasi dan Tata Letak Instansi .......................................................... 6 3. Visi dan Misi Instansi ........................................................................ 6 4. Struktur Organisasi Instansi ............................................................... 6 B. KERACUNAN PANGAN ..................................................................... 8 C. Bacillus cereus ....................................................................................... 9 D. Salmonella spp. ...................................................................................... 11 E. METODE DETEKSI KONVENSIONAL ............................................. 14 F. METODE DETEKSI CEPAT (RAPID METHOD) BAKTERI PATOGEN DENGAN PCR ................................................................... 17 1. Tahap Pra-Amplifikasi Real-Time PCR ............................................ 19 2. Pembacaan Produk Hasil Real-Time PCR ......................................... 23 III. METODOLOGI PENELITIAN .............................................................. 27 A. TEMPAT DAN WAKTU MAGANG .................................................... 27 B. LINGKUP KEGIATAN PENELITIAN ................................................ 27 C. BAHAN DAN ALAT ............................................................................ 29 iii 1. Identifikasi Serovar Salmonella spp. ............................................... 29 2. Metode Deteksi B. cereus Berbasiskan DNA ................................. 30 D. METODE PENELITIAN ....................................................................... 31 1. Identifikasi Serovar Salmonella spp. ............................................... 31 2. Metode Deteksi B. cereus Berbasiskan DNA ................................. 32 IV. HASIL DAN PEMBAHASAN ................................................................. 41 A. Identifikasi Serovar Salmonella spp. ...................................................... 41 B. Metode Deteksi B. cereus Berbasiskan DNA ......................................... 45 1. Tahap Persiapan Sampel .................................................................. 45 2. Tahap Isolasi DNA .......................................................................... 48 3. Tahap Amplifikasi ........................................................................... 57 4. Evaluasi Kinerja Real-Time PCR ..................................................... 59 V. KESIMPULAN DAN SARAN ................................................................. 69 A. KESIMPULAN ...................................................................................... 69 B. SARAN .................................................................................................. 69 DAFTAR PUSTAKA ....................................................................................... 70 LAMPIRAN ...................................................................................................... 74 iv DAFTAR TABEL Halaman Tabel 1. Pembagian subgenus Salmonella berdasarkan karakteristik biokimia ............................................................................................ 12 Tabel 2. Distribusi serovar dalam genus Salmonella pada tahun 2007 ........... 13 Tabel 3. Data uji serologi antigen O dan motilitas........................................... 41 Tabel 4 Data kurva standar dari metode isolasi pendidihan, ekstraksi dengan fenol:kloroform, dan ekstraksi dengan kit komersial DNA .............. 61 Tabel 5. Limit deteksi kultur murni B. cereus dan B. cereus dalam nasi goreng ........................................................................................ 62 v DAFTAR GAMBAR Halaman Gambar 1a. Antisera ...................................................................................... 16 Gambar 1b. Visualisasi aglutinasi .................................................................. 16 Gambar 2. Grafik sigmoidal proses amplifikasi dengan real-time PCR (Edwards et al., 2004). ................................................................ Gambar 3. 23 Kurva standar amplifikasi kultur murni dari enam tingkat pengenceran dengan real-time PCR (Edwards et al., 2004) ...... 24 Gambar 4. Melt curve yang memiliki sebuah puncak Tm ........................... 25 Gambar 5. Tahapan kerja pada identifikasi serovar Salmonella spp. .......... 27 Gambar 6. Tahapan kerja pada uji amplifikasi DNA kultur murni B. cereus ...................................................................................... Gambar 7. Gambar 8. 28 Tahapan kerja pada uji amplifikasi DNA B. cereus dalam nasi goreng .................................................................................. 29 Koloni tipikal B. cereus pada MYPA + Polymyxin B ............... 46 Gambar 9a. Fase aqueous (atas), Interfase (tengah), dan Fase organik (bawah) dalam isolasi fenol:kloroform yang memiliki lapisan interfase tebal ............................................................................. 53 Gambar 9b. Fase aqueous (atas), Interfase (tengah), dan Fase organik (bawah) dalam isolasi fenol:kloroform yang memiliki lapisan interfase sangat tipis ................................................................... 53 Gambar 10a. Grafik amplifikasi DNA B. cereus hasil isolasi metode pendidihan ..................................................................... 60 Gambar 10b.Grafik amplifikasi DNA B. cereus hasil isolasi metode dengan pelarut fenol:kloroform ..................................... 60 Gambar 10c. Grafik amplifikasi DNA B. cereus hasil isolasi metode dengan kit komersial ..................................................... 60 Gambar 10d.Kurva standar kultur murni B. cereus metode pendidihan ......... 60 vi Gambar 10e. Kurva standar kultur murni B. cereus metode dengan pelarut fenol:kloroform .......................................................................... 60 Gambar 10f. Kurva standar kultur murni B. cereus metode dengan kit komersial ............................................................................... 60 Gambar 11a. Melt curve dari DNA yang diisolasi dengan metode pendidihan 67 Gambar 11b. Melt curve dari DNA yang diisolasi dengan metode ekstraksi fenol:kloroform .......................................................................... 66 Gambar 11c. Melt curve dari DNA yang diisolasi dengan metode ekstraksi dengan kit komersial .................................................................. 66 Gambar 12a.Grafik amplifikasi isolat DNA B. subtilis .................................. 67 Gambar 12a. Melt curve isolat DNA B. subtilis ............................................. 67 vii DAFTAR LAMPIRAN Halaman Lampiran 1. Struktur organisasi Badan POM RI.......................................... 74 Lampiran 2. Prosedur pembuatan media TSB (Atlas, 2006) ....................... 75 Lampiran 3. Prosedur pembuatan media MYPA (Atlas, 2006) ................... 76 Lampiran 4. Persiapan kultur bakteri (PROM Biotech, 2008) .................... 77 Lampiran 5. Data jumlah koloni B. cereus dan konsentrasi kultur murni B. cereus ................................................................................. Lampiran 6. Isolasi DNA metode pendidihan (Hein et al., 2001 dengan modifikasi) .............................................................................. Lampiran 7. 78 79 Isolasi DNA metode ekstraksi dengan fenol:kloroform (Sambrook et al., 1989 dengan modifikasi) ........................... 80 Lampiran 8. Isolasi DNA sesuai dengan panduan kit komersial ................ 81 Lampiran 9. Penetapan Limit of Detection (LOD) Mikroba (PROM Biotech, 2008) ........................................................... 82 Lampiran 10. Uji motilitas Salmonella spp. ................................................. 83 viii I. PENDAHULUAN A. LATAR BELAKANG Peristiwa keracunan pangan sering dilaporkan di media massa dan sering pula diasumsikan sebagai penyakit ringan seperti sakit perut, mual, dan muntah. Asumsi ini menyebabkan kajian dan penelusuran terhadap penyakit asal pangan dengan gejala tersebut kurang mendapat perhatian dari masyarakat, pemeriksaan kurang seksama dari pemerintah serta pihak terkait bidang kesehatan. Hal ini terbukti dengan minimnya data dan laporan mengenai Kejadian Luar Biasa (KLB) yang diperantarai pangan, padahal pelaporan sangat penting untuk tindak lanjut investigasi keracunan pangan dan untuk itu diperlukan pendeteksian dan pengidentifikasian penyebab keracunan pangan. Penanganan produk olahan pangan yang buruk dan konsumsi pangan kurang matang biasanya menjadi penyebab utama keracunan pangan. Salah satu contoh penyakit akibat keracunan pangan ialah Salmonellosis, hal ini sering menjadi masalah kesehatan manusia yang disebabkan oleh bakteri patogen Salmonella (Gast, 2003). Salmonellosis sangat mudah ditularkan dari hewan ke manusia baik secara langsung ataupun melalui perantara seperti produk makanan yang berasal dari hewan, tumbuhan, dan lingkungan. Salmonella enterica serovar Enteritidis merupakan salah satu penyebab utama gastroenteritis. Pangan yang berpeluang tinggi menyebabkan keracunan pangan ialah pangan yang frekuensi konsumsinya tinggi dan dikonsumsi dalam jumlah banyak. Pangan yang banyak dikonsumsi oleh penduduk Indonesia ialah nasi (Deptan, 2009). Nasi selain sebagai sumber karbohidrat bagi manusia, juga menjadi sumber nutrisi bagi mikroorganisme. Produk olahan nasi yang cukup banyak diminati oleh penduduk Indonesia ialah nasi goreng. Selain itu, nasi goreng yang digunakan biasanya berasal dari nasi sisa yang dibiarkan semalam dan baru dimasak keesokan paginya. Hal ini rentan terhadap kontaminasi bakteri patogen karena dibiarkan pada zona berbahaya (suhu 5-60°C). Oleh karena itu, diperlukan pendeteksian yang tepat untuk 1 menganalisis dan menjamin keamanan pangan nasi goreng dari bahaya mikrobiologi tersebut. Bakteri patogen yang berkembang dengan baik pada substrat berkarbohidrat tinggi salah satunya ialah Bacillus cereus (Supardi dan Sukamto, 1992). Bakteri ini merupakan jenis bakteri Gram positif yang memiliki peptidoglikan tebal dan mampu menghasilkan spora tahan panas serta toksin ekstraseluler. Hal ini menyebabkan B. cereus masih mungkin berkembang walaupun makanan telah dimasak. Jumlah B. cereus yang mencapai 106 koloni per gram pangan telah mampu menyebabkan keracunan pangan (USFDA, 2001). Pendeteksian bakteri patogen dalam pangan pada umumnya dilakukan dengan metode konvensional yang berbasiskan pada reaksi biokimia. Metode konvensional memerlukan serangkaian uji, yaitu uji morfologi, uji biokimia, dan perlu konfirmasi dengan uji serologi. Penetapan serovar melalui uji serologi merupakan rangkaian pendeteksian Salmonella yang penting sebab bakteri ini sangat beragam serotipenya. Selain itu, penetapan serovar sangat membantu studi epidemiologi dan penelusuran sumber bakteri penyebab penyakit asal pangan. Salah satu metode konvensional untuk menetapkan serovar Salmonella adalah Slide Agglutination Test (SAT). Melalui uji ini data kasus epidemiologi keracunan pangan di seluruh dunia dapat diperbandingkan. Hal ini memakan waktu berhari-hari, padahal konsumsi pangan terus berlangsung dan perlu tindakan cepat untuk mendeteksi bahaya mikrobiologi dalam pangan. Oleh karena itu, dibutuhkan metode deteksi yang mampu menganalisis faktor penyebab keracunan pangan secara cepat, tepat, dan sensitif. Metode deteksi terus mengalami perkembangan untuk melengkapi ataupun mengatasi keterbatasan metode standar sebelumnya. Metode cepat yang dalam dekade ini berkembang pesat ialah metode deteksi berbasiskan DNA. Metode berbasiskan DNA memiliki spesifisitas terbaik dan sesuai untuk mendeteksi patogen dalam pangan (de Boer dan Beumer, 1999). Prinsip metode ini adalah hibridisasi antara DNA target dan fragmen oligonukleotida yang komplementer terhadap DNA target tersebut. Metode 2 berbasiskan DNA dibedakan menjadi tiga, yaitu hibridisasi asam nukleat dengan probe, amplifikasi dengan Polymerase Chain Reaction (PCR), dan subtyping molekuler. Amplifikasi DNA dengan PCR, dimana DNA spesifik dari bakteri patogen diperbanyak mencapai jutaan kopi DNA sehingga tampak sebagai pita DNA, dilanjutkan dengan deteksi elektroforesis gel agarosa. Namun, proses amplifikasi dan deteksi yang terpisah ini dianggap berisiko terhadap kontaminasi pita DNA dan kurang praktis. Oleh karena itu, dikembangkanlah real-time PCR yang melangsungkan proses amplifikasi dan deteksi sekaligus dalam sebuah instrumen. Sensitivitas uji PCR yang tinggi sangat menguntungkan penelusuran patogen penyebab keracunan pangan (misalnya B. cereus). Secara teoritis hanya dengan satu molekul DNA dari bakteri dalam pangan, pendeteksian patogen dapat dilakukan (Naravaneni dan Jamil, 2005). Di samping itu, teknik ini fleksibel sehingga berkembang secara pesat disesuaikan dengan kebutuhan analisis. Fleksibilitasnya memberikan keleluasaan bagi peneliti dalam memodifikasi tahapan persiapan sampel hingga berupa isolat DNA. Persiapan isolat DNA dan amplifikasi seringkali berbeda bagi setiap bakteri. Hal ini dilakukan untuk memperoleh optimasi amplikon sehingga deteksi B. cereus dalam pangan semakin sensitif, akurat, dan valid. Preparasi isolat DNA sebelum amplifikasi menjadi tahapan penting agar amplifikasi berjalan lancar. Isolasi DNA menjadi tahapan kritis setiap metode berbasiskan DNA. Untuk memperoleh isolat DNA dengan kuantitas dan kualitas yang baik, beragam metode isolasi DNA telah dikembangkan. Beberapa metode yang digunakan untuk preparasi isolat DNA ialah metode pendidihan, metode pelisisan dengan pelarut alkalin, dan metode ekstraksi dengan fenol:kloroform. Metode isolasi DNA dengan fenol:kloroform paling banyak digunakan untuk mengekstraksi DNA kromosomal dari sel (Sambrook et al., 1989). Kemurnian isolat DNA berpengaruh besar terhadap sensitivitas uji real-time PCR. Sensitivitas yang diperoleh dari real-time PCR lebih tinggi dibandingkan PCR standar, yang ditunjukkan dengan angka limit deteksi yang rendah. 3 Hal yang menjadi tantangan dalam deteksi cepat ialah persiapan sampel yang belum terstandarisasi. Untuk itu diperlukan pengembangan metode pada tahap persiapan isolat DNA agar metode PCR menjadi metode analisa yang lebih baik, lebih cepat serta lebih sensitif, dan selanjutnya metode tersebut dapat divalidasi. B. TUJUAN Secara umum, kegiatan magang ini bertujuan untuk memperluas wawasan, melatih sikap dan kemampuan teknis mahasiswa serta mengaplikasikan ilmu selama magang di bidang keamanan pangan, Pusat Riset Obat dan Makanan Badan POM RI. Tujuan khusus dari kegiatan magang ini, antara lain: 1. Mengidentifikasi serovar Salmonella spp dengan metode deteksi berbasiskan imunologi. 2. Mempelajari metode deteksi B. cereus berbasiskan DNA dengan realtime PCR, mencakup tahap isolasi DNA dan amplifikasi. 3. Mengevaluasi limit deteksi dan spesifisitas uji B. cereus dari sampel pangan nasi goreng. C. MANFAAT Manfaat yang dapat diperoleh dari kegiatan ini adalah: 1. Melengkapi data serovar Salmonella spp. yang tumbuh pada sampel pangan dan data tersebut dapat dipergunakan untuk penelusuran sumber penyebab kasus keracunan pangan yang mungkin disebabkan oleh genus Salmonella. 2. Memberikan pengetahuan mengenai perkembangan metode deteksi cepat dengan real-time PCR serta metode isolasi DNA yang dapat diterapkan oleh laboratorium-laboratorium uji untuk mendeteksi bakteri patogen dalam sampel pangan. 4 II. TINJAUAN PUSTAKA A. KEADAAN UMUM INSTANSI MAGANG 1. Sejarah dan Perkembangan Instansi Kemajuan teknologi telah membawa perubahan-perubahan yang cepat dan signifikan pada industri farmasi, obat asli Indonesia, makanan, kosmetika dan alat kesehatan. Dengan menggunakan teknologi modern, industri-industri tersebut kini mampu memproduksi dalam skala yang sangat besar mencakup berbagai produk dengan range yang sangat luas. Dengan dukungan kemajuan teknologi transportasi dan entry barrier yang makin tipis dalam perdagangan internasional, maka produk-produk tersebut dalam waktu yang amat singkat dapat menyebar ke berbagai negara dengan jaringan distribusi yang sangat luas dan mampu menjangkau seluruh strata masyarakat. Konsumsi masyarakat terhadap produk-produk termaksud cenderung terus meningkat, seiring dengan perubahan gaya hidup masyarakat termasuk pola konsumsinya. Sementara itu pengetahuan masyarakat masih belum memadai untuk dapat memilih dan menggunakan produk secara tepat, benar dan aman. Di lain pihak, iklan dan promosi secara gencar mendorong konsumen untuk mengkonsumsi secara berlebihan dan seringkali tidak rasional. Perubahan teknologi produksi, sistem perdagangan internasional dan gaya hidup konsumen tersebut pada realitasnya meningkatkan risiko dengan implikasi yang luas pada kesehatan dan keselamatan konsumen. Apabila terjadi produk sub standar, rusak atau terkontaminasi oleh bahan berbahaya maka risiko yang terjadi akan berskala besar dan luas serta berlangsung secara amat cepat. Untuk itu Indonesia harus memiliki Sistem Pengawasan Obat dan Makanan (SisPOM) yang efektif dan efisien yang mampu mendeteksi, mencegah dan mengawasi produk-produk termaksud untuk melindungi keamanan, keselamatan dan kesehatan konsumennya baik di dalam maupun di luar negeri sehingga dibentuklah Badan POM yang memiliki 5 jaringan nasional dan internasional serta kewenangan penegakan hukum dan memiliki kredibilitas profesional yang tinggi. 2. Lokasi dan Tata Letak Instansi Badan POM RI terletak di Jalan Percetakan Negara No. 23, Jakarta Pusat. 3. Visi dan Misi Instansi Adapun Visi dan Misi dari Badan POM RI adalah : a. Visi Badan POM RI Obat dan Makanan terjamin aman, bermanfaat dan bermutu. b. Misi Badan POM RI Melindungi masyarakat dari Obat dan Makanan yang berisiko terhadap kesehatan. 4. Struktur Organisasi Instansi Badan POM RI ditetapkan berdasarkan Keputusan Presiden Nomor 166 Tahun 2000 tentang Kedudukan, Tugas, fungsi, Kewenangan, Susunan Organisasi dan Tata Kerja Lembaga Pemerintah Non Departemen, sebagaimana telah diubah dengan Keputusan Presiden Nomor 173 tahun 2000. Pembentukan Badan POM RI ini ditindaklanjuti dengan Keputusan Kepala Badan Pengawas Obat dan Makanan Nomor 02001/SK/KBADAN POM RI, tanggal 26 Februari tahun 2001, tentang Organisasi dan Tata Kerja Badan Pengawas Obat dan Makanan setelah mendapatkan persetujuan Menteri Negeri Pendayagunaan Aparatur Negara Nomor 34/M.PAN/2/2001 tanggal 1 Februari 2001. Berikut ini adalah struktur organisasi Badan POM: a. Kepala Badan Pengawas Obat dan Makanan b. Sekretariat Utama c. Inspektorat d. Deputi Bidang Pengawasan Produk Terapetik dan Narkotika, Psikotropika, dan Zat Adiktif (NAPZA) 6 e. Deputi Bidang Pengawasan Obat Tradisional, Kosmetik dan Produk Komplemen f. Deputi Bidang Pengawasan Keamanan Pangan dan Bahan Berbahaya g. Pusat Pengujian Obat dan Makanan Nasional h. Pusat Penyidikan Obat dan Makanan i. Pusat Riset Obat dan Makanan j. Pusat Informasi Obat dan Makanan k. Unit Pelaksana Teknis Badan POM Struktur organisasi dalam bentuk skema dapat dilihat pada Lampiran 1. Pusat Riset Obat dan Makanan, bagian dari struktur organisasi Badan POM RI, merupakan tempat magang dipenuhi. Berikut ini adalah penjelasan lebih lanjut mengenai sub-instansi ini: a. Kedudukan Pusat Riset Obat dan Makanan (PROM) adalah unsur pelaksana tugas Badan POM RI yang berada di bawah dan bertanggung jawab kepada Kepala Badan POM. Dalam pelaksanaan tugas sehari-hari secara teknis dibina oleh Deputi dan secara administrasi dibina oleh Sekretariat Utama. Pusat Riset Obat dan Makanan dipimpin oleh seorang kepala pusat. b. Tugas dan Fungsi Sesuai dengan SK Kepala Badan POM RI No. 02001/KBADAN POM RI tanggal 26 Februari 2001, Pusat Riset Obat dan Makanan mempunyai tugas melaksanakan kegiatan di bidang riset toksikologi, keamanan pangan dan produk terapetik. Pusat Riset Obat dan Makanan menyelenggarakan fungsi : 1. Penyusunan rencana dan program riset Obat dan Makanan. 2. Pelaksanaan riset obat dan makanan. 3. Evaluasi dan penyusunan laporan pelaksanaan riset Obat dan Makanan. c. Susunan Organisasi Secara organisasi, Pusat Riset Obat dan Makanan terdiri dari: 7 1) Bidang Toksikologi 2) Bidang Keamanan Pangan 3) Bidang Produk Terapetik 4) Kelompok Pejabat Fungsional 5) Sub Bagian Tata Usaha d. Bidang Toksikologi Bidang Toksikologi mempunyai tugas melaksanakan penyusunan rencana dan program serta evaluasi dan penyusunan laporan pelaksanaan riset toksikologi. e. Bidang Keamanan Pangan Bidang Keamanan Pangan mempunyai tugas melaksanakan penyusunan rencana dan program serta evaluasi dan penyusunan laporan pelaksanaan riset keamanan pangan. f. Bidang Terapetik Bidang Produk terapetik mempunyai tugas melaksanakan penyusunan rencana dan program serta evaluasi dan penyusunan laporan pelaksanaan riset produk terapetik. g. Sub Bagian Tata Usaha Sub Bagian Tata Usaha mempunyai tugas memberikan pelayanan teknis dan administrasi di lingkungan Pusat Riset Obat dan Makanan. B. KERACUNAN PANGAN Pangan merupakan kebutuhan pokok manusia karena di dalamnya terdapat zat-zat gizi yang penting bagi kehidupan. Zat-zat gizi tersebut diperlukan untuk memulihkan dan memperbaiki jaringan tubuh yang rusak, mengatur proses di dalam tubuh, perkembangbiakan, dan menghasilkan energi untuk beraktivitas. Zat gizi yang dimaksud, antara lain: karbohidrat, protein, lemak, vitamin dan beberapa mineral. Bahan pangan dengan komponen tersebut juga merupakan medium yang baik untuk pertumbuhan mikroba. Pertumbuhan mikroba dalam bahan pangan dipengaruhi oleh berbagai faktor, seperti tersedianya nutrien, air, suhu, pH, oksigen, dan adanya zat penghambat. Keberadaan mikroba di dalam pangan tidak selamanya 8 menguntungkan, tetapi juga dapat mendatangkan kerugian. Misalnya jika kehadiran mikroba tersebut mengubah bau, rasa, dan warna yang tidak dikehendaki; menurunkan berat atau volume; menurunkan nilai gizi/nutrisi; mengubah bentuk dan susunan senyawa serta menghasilkan toksin yang membahayakan di dalam pangan (Supardi dan Sukamto, 1999). Pangan yang memiliki kandungan mikroba tertentu dapat menimbulkan penyakit bila dikonsumsi. Menurut penyebabnya, penyakit yang ditimbulkan oleh makanan dapat digolongkan dalam dua kelompok besar, yaitu keracunan dan infeksi mikroba. Keracunan dapat terjadi karena tertelannya suatu racun baik organik atau anorganik yang mungkin terdapat secara alamiah pada bahan pangan, serta tertelannya toksin yang merupakan hasil metabolisme sel-sel mikroba tertentu. Gejala keracunan karena toksin tersebut disebut intoksikasi. Sedangkan tertelannya atau masuknya mikroba ke dalam tubuh, kemudian menembus sistem pertahanan tubuh dan hidup serta berkembang biak di dalam tubuh disebut infeksi (Supardi dan Sukamto, 1999). Menurut Walderhaug (2007), patogen-patogen penyebab keracunan pangan, antara lain: Salmonella spp., Staphylococcus aureus, Clostridium perfringens, Bacillus cereus, Campylobacter sp., Shigella sp., Clostridium botulinum, dan Escherichia coli. Salah satu bentuk kasus keracunan pangan yang disebabkan oleh bakteri patogen ialah keracunan pangan yang menjadi Kejadian Luar Biasa (KLB) di Amerika, Inggris, Kanada, dan Norwegia. Bakteri penyebab KLB tersebut adalah Bacillus cereus yang terdapat pada nasi putih dan nasi goreng (Supardi dan Sukamto, 1999). Agar KLB tersebut dapat dicegah di Indonesia, diperlukan metode yang mampu mendeteksi bakteri patogen tersebut pada sampel pangan secara sensitif, spesifik, akurat, dan cepat untuk menjamin keamanan pangan. C. Bacillus cereus Bacillus cereus adalah bakteri pembentuk spora yang tergolong ke dalam famili Bacillaceae. Spora B. cereus tahan terhadap panas dan radiasi. Bakteri ini bersifat aerobik sampai anaerobik fakultatif, katalase positif, dan kebanyakan Gram positif serta mempunyai enzim proteolitik (Fardiaz, 1992). 9 B. cereus juga memproduksi enterotoksin dan metabolit-metabolit lainnya. Tidak memproduksi indol, reaksi Voges-Proskauer positif, dapat menggunakan sitrat sebagai sumber karbon, mereduksi nitrat, tidak memproduksi urease dan penisilinase, dapat tumbuh secara anaerobik di dalam media cair yang mengandung 1% glukosa, memproduksi asam dari glukosa, sukrosa, maltosa dan gliserol, serta tahan terhadap lisozim (Supardi dan Sukamto, 1999). Jenis pangan yang sering ditumbuhi B. cereus terutama adalah daging, nasi, sayuran, sosis, makaroni, dan kadang-kadang ikan, susu atau es krim. Pangan penyebab keracunan umumnya mengandung sel B. cereus dalam jumlah tinggi. Analisis mikrobiologi terhadap B. cereus pada agar darah menunjukkan bahwa dari 17 KLB yang terjadi di Inggris tahun 1971-1976, kandungan B. cereus pada nasi penyebab keracunan tersebut berkisar antara 3 x 105 – 2 x 109 CFU/g dengan rata-rata 5 x 107 CFU/g (Supardi dan Sukamto, 1999). Konsumsi pangan yang mengandung lebih dari 106 B.cereus/g (USFDA, 2001) sudah dapat menyebabkan keracunan pangan, khususnya pada pangan yang dibiarkan saat preparasi tanpa dimasukkan ke dalam lemari pendingin sebelum dihidangkan. B. cereus tumbuh cepat apabila substratnya mengandung karbohidrat. Sedangkan bila substratnya tidak mengandung karbohidrat, pertumbuhannya akan sangat lambat dan tidak dapat membentuk toksin. B. cereus dapat tumbuh secara baik pada media yang mengandung 0.025 M glukosa dan mencapai maksimum setelah 4.5 jam (Supardi dan Sukamto, 1999). Produksi toksin terjadi selama pertumbuhan logaritmik, dan mencapai maksimum sampai glukosa di dalam medium habis dipecah oleh bakteri tersebut. Galur B. cereus yang bersifat patogenik digolongkan ke dalam bakteri penyebab intoksikasi dan dapat dibedakan atas dua grup berdasarkan sifat patogeniknya, yaitu galur penyebab diare dan galur penyebab muntah. Galur penyebab diare yang memproduksi enterotoksin dapat tumbuh pada berbagai pangan dan mempunyai waktu inkubasi sejak tertelan sampai timbulnya gejala intoksikasi berkisar antara 8-16 jam. Galur yang memproduksi toksin 10 emetik mempunyai masa inkubasi lebih pendek, sekitar 1-5 jam (Supardi dan Sukamto, 1999). Galur B. cereus yang beragam tersebut dapat dibedakan berdasarkan gen spesifik yang dimiliki masing-masing galur. Berdasarkan beberapa penelitian terdapat beberapa gen spesifik yang terdapat pada B. cereus, antara lain: gen penghasil cereulide (ces), sejenis toksin emetik (Fricker et al., 2007), phosphotidyl inositol (PI-1) (Myers dan Sakelaris, 2004), gyrase (gyrB) (Myers dan Sakelaris, 2004), enterotoksin non-hemolitik (Nhe) (Hansen dan Hendriksen, 2001), sitotoksin (cytK) (Lund et al., 2000), hemolysin (hblA) (Mantynen dan Lindstrom, 1998), dan enterotoksin T (BceT) (Mantynen dan Lindstrom, 1998). Target gen spesifik akan menentukan primer yang digunakan dalam uji amplifikasi dengan PCR, target gen yang banyak digunakan ialah ces karena toksin ini spesifik dihasilkan B. cereus emetik (Fricker et al., 2007). Berbeda dengan sekuens gen 16S rRNA karena identik dengan beberapa bakteri lain, termasuk B. anthracis. Keidentikan tersebut terlihat dari struktur primer 16S rRNA Bacillus anthracis, Bacillus cereus, Bacillus mycoides, dan Bacillus thuringiensis setelah ditetapkan dengan metode sekuensing dideoksi transkripsi terbalik, hasilnya menunjukkan semua galur memiliki sekuens serupa (tingkat kemiripannya lebih dari 99%) (Ash et al., 1991). Penggunaan gen spesifik bersamaan dengan dye fluoresence pada realtime PCR memberikan limit deteksi (LOD) B.cereus sebesar 101-103 cfu/g sampel pangan tanpa tahap pre-enrinchment. Dengan pengkayaan, limit deteksi dapat mencapai 100 cfu/g. Apabila metode isolasi DNA dengan pendidihan saja, limit deteksi menjadi kurang sensitif (Fricker et al., 2007). D. Salmonella spp. Salmonella merupakan bakteri Gram negatif berbentuk batang yang tidak membentuk spora, fakultatif anaerobik, mampu tumbuh pada pH mendekati 4.0-9.0 dengan pH optimum 7.0, dan termasuk dalam famili Enterobacteriaceae. Bakteri ini berukuran 0.7-1.5 x 2-5 µm, motil dengan flagela peritrikus, kecuali Salmonella Pullorum dan Salmonella Gallinarum 11 yang tidak motil karena tidak mempunyai flagela. Bakteri ini tumbuh optimum pada suhu 35-37°C, dapat mengkatabolisme bermacam karbohidrat menjadi asam dan gas, menggunakan sitrat sebagai sumber tunggal karbon, memproduksi H2S dan dapat mendekarboksilasi lysine menjadi kadaverin dan ornithin menjadi putrescin. Salmonella dapat tumbuh pada kadar garam maksimal 8% (Rusyanto, 2005). Nama Salmonella pertama kali diberikan oleh Lignieres pada tahun 1990 sebagai penghormatan kepada seorang ahli bakteriologi Amerika Serikat D.E. Salmon yang telah berhasil mengkarakterisasikan bakteri Bacillus penyebab kolera pada babi dan memasukkannya dalam famili Enterobacteriaceae. Taksonomi Salmonella pertama kali diperkenalkan oleh White (1929) dan dimodifikasi oleh Kauffman (1934). Tahun 1966, Kauffman mulai mengklasifikasikan 51 grup somatik Salmonella menjadi 4 sub genera dengan menggunakan karakteristik biokimia. Sub genus kelima lalu ditambahkan setelah ditemukannya beberapa karakteristik yang berbeda. Adapun karakteristik biokimia masing masing sub genus dapat dilihat pada Tabel 1. Tabel 1. Pembagian subgenus Salmonella berdasarkan karakteristik biokimia* Klasifikasi Subgenus 1 Subgenus 2 Subgenus 3 Subgenus 4 Subgenus 5 Karakteristik Mampu menggunakan dulcitol, banyak berhubungan atau ditemukan pada manusia atau hewan vertebrata berdarah panas. Isolasi frekuensinya sangat tinggi Mampu menggunakan dulcitol dan malonat Mampu menggunakan malonat dan O-nitriphenyl-ß-Dgalactopyranoside (ONPG) Mampu tumbuh dalam KCN Tidak dapat memetabolisme dulcitol, malonat, mampu mengkatabolisme ONPG dan tumbuh dalam KCN *Sumber: D’Aoust (2000) Genus Salmonella terdiri dari dua spesies, yaitu S. enterica. dan S. bongori. Spesies S. enterica masih dapat dikelompokkan lagi menjadi enam subspesies, antara lain: S. enterica subsp. enterica, S. enterica subsp. salamae, S. enterica subsp. arizonae, S. enterica subsp. diarizonae, S. enterica subsp. houtenae, and S. enterica subsp. indica berdasarkan antigen (O) somatik dan antigen (H) flagelar melalui serotyping. Distribusi spesies beserta jumlah serovar dari masing-masing subspesies disajikan pada Tabel 2. 12 Spesies dan subspesies dibedakan berdasarkan uji biokimia, serologi, dan genetik (Mazumdar, 2008). Tabel 2. Distribusi serovar dalam genus Salmonella pada tahun 2007* Spesies Salmonella enterica Subspesies Enterica (I) Salamae (II) Arizonae (IIIa) Diarizonae (IIIb) Houtenae (IV) Indica (V) Salmonella bongori (VI) Total Jumlah serovar 1531 505 99 336 73 13 22 2579 *Sumber: World Health Organization (WHO), 2009 Spesies terbanyak dari genus Salmonella adalah Salmonella enterica dan subspesies terbanyak ialah subspesies enterica yang berjumlah 1531 serovar. Angka romawi pada masing-masing subspesies, kecuali angka VI pada spesies Salmonella bongori, digunakan untuk mempersingkat penamaan serovar. Pada tingkat genus diferensiasi Salmonella ke dalam spesies didasarkan pada perbedaan reaksi bakteri terhadap antigen. Hingga tahun 2000 lebih dari 2500 serovar Salmonella telah teridentifikasi (D’Aoust, 2000). Skema yang disepakati secara internasional untuk menetapkan serovar Salmonella hingga saat ini ialah skema Kauffmann-White. Seluruh serotipe Salmonella ditetapkan berdasarkan antigennya (Mazumdar, 2008). Antigen O merupakan antigen karbohidrat bagian dari lipopolisakarida. Gram negatif, salah satunya Salmonella, memiliki antigen O yang unik. Dan antigen H tersusun dari subunit protein, disebut flagelin, yang membentuk karakteristik flagela. Salmonella merupakan bakteri enterik yang unik karena bakteri ini dapat mengekspresikan dua antigen flagela yang berbeda. Kedua antigen flagela dikenal dengan antigen H fase 1 (monofasik) dan fase 2 (bifasik). Isolat dengan flagela monofasik hanya menunjukkan satu tipe flagelin, sedangkan Salmonella bifasik memiliki fase 1 dan 2 pada antigen H. Penentuan tipe bifasik dengan cara menginokulasi Salmonella pada media inversi, yaitu media semi solid yang diberikan antisera positif dari fase 1. Salmonella bifasik akan tetap menunjukkan pergerakan yang ditandai dengan 13 meluasnya koloni ke tepi agar, sedangkan Salmonella monofasik tidak motil sebab antibodi yang terdapat pada sera telah berikatan dengan antigen flagela pada bakteri tersebut. Salmonella merupakan bakteri patogen penyebab Salmonellosis. Gejala klinis salmonellosis pada manusia terbagi menjadi demam tifoid dan non tifoid. Demam tifoid atau disebut dengan demam enterik disebabkan infeksi oleh galur tifoid atau paratyphi sehingga disebut salmonellosis tifoid. Demam enterik dapat ditularkan atau disebarkan oleh individu yang terinfeksi serta makanan atau minuman yang terkontaminasi. Demam non tifoid disebut juga gastroenteritis merupakan infeksi terbatas dan terlokalisasi pada epitel intestinal. Gejala salmonellosis non tifoid hampir mirip dengan salmonellosis tifoid, yaitu: mual, kram abdominal, diare berair dan mungkin berdarah, demam dengan durasi kurang dari 48 jam, serta muntah dapat terjadi 8-72 jam setelah terinfeksi. Center for Disease Control and Prevention (CDC, 2008) Amerika Serikat mengestimasi setiap tahunnya di Amerika Serikat jumlah kasus penyakit Salmonellosis non tifoid dari bahan pangan (foodborne disease) mencapai 1.4 juta kasus, 15608 harus dirawat dan 553 meninggal (30.6% dari seluruh kasus kematian yang disebabkan oleh patogen asal pangan). E. METODE DETEKSI KONVENSIONAL Analisis pangan terhadap kemungkinan adanya bakteri patogen atau bakteri pembusuk merupakan standar yang diharuskan untuk mengetahui kualitas pangan dan menjamin keamanan pangan (de Boer dan Beumer, 1999). Analisis tersebut biasanya menggunakan metode konvensional yang telah distandarkan sehingga setiap negara di dunia memiliki standar yang sama dalam menilai kualitas pangan. Metode deteksi konvensional yang dimaksud ialah metode pengkulturan bakteri pada media spesifik dan menghitung sel bakteri yang hidup dalam pangan. Prinsipnya ialah mikroorganisme bermultiplikasi pada media pertumbuhan sehingga dapat dideteksi. Metode ini sensitif, dapat memberi 14 hasil uji baik kuantitatif ataupun kualitatif, dan menjadi standar bagi uji mikrobiologi terhadap produk pangan secara internasional. Metode konvensional meliputi persiapan media kultur (pengkayaan, pengkayaan selektif, dan penumbuhan pada agar selektif), penghitungan koloni, pengkarakterisasian dengan uji biokimia, yang dapat dilanjutkan dengan penetapan serotipe (serovar) dengan uji serologi. Uji biokimia saat ini dipermudah dengan adanya API test. Identifikasi dengan API, menurut USFDA dalam BAM (2007), tidak dapat menggantikan uji serologi. API test hanya untuk mengidentifikasi perkiraan spesies tertentu, misalnya Salmonella spp. Oleh karena itu, penetapan serovar Salmonella tidak dapat ditentukan dengan uji biokimia API. Bakteri dari genus yang sama, setelah diuji biokimiawi memiliki sifat biokimia yang sama, tidak menjamin bahwa bakteri-bakteri tersebut berasal dari serovar yang sama. Bakteri-bakteri tersebut bisa saja berasal dari subspesies yang sama, tetapi serotipenya berbeda. Hal ini disebabkan dinding sel mikroorganisme mengandung protein dan lipopolisakarida. Setiap bakteri memiliki kemungkinan stuktur molekul protein atau lipopolisakarida yang berbeda. Antigen merupakan bagian dari struktur tersebut, oleh karena itu antigen setiap bakteri mungkin pula berbeda-beda. Pada genus Salmonella pembedaan antar subspesies dapat lebih lanjut dilakukan dengan melihat antigen O, antigen Vi, dan antigen H spesies tersebut. Konfirmasi patogen dengan uji imunologi dapat dilakukan dengan Slide Agglutination Test (SAT) yang diterapkan pada uji serologi. Uji serologi (serotyping) merupakan tahapan dalam metode konvensional untuk mengkonfirmasi koloni tipikal yang telah diperoleh dari uji morfologi dan uji biokimia. Juga mengidentifikasi organisme pada tingkat subspesies dan menjadi salah satu alat penting bagi klasifikasi taksonomi serta pengawasan kasus keracunan pangan atau KLB akibat kontaminasi patogen tertentu, misalnya Salmonella spp. Walaupun uji biokimia juga mengkonfirmasi koloni tipikal, uji tersebut tidak dapat mensubstitusi uji serologi. Sebab kedua uji mengidentifikasi dua sifat yang berbeda, sifat biokimia dan serotipe. 15 Serotipe galur Salmonella dilakukan dengan mengidentifikasi antigen permukaan (LPS, antigen O) terlebih dahulu, kemudian antigen flagela (protein, antigen H). Sebagian besar galur Salmonella menunjukkan 2 fase antigen H. Setelah keseluruhan serotyping dilaksanakan, barulah serotipe dapat ditetapkan berdasarkan skema Kauffmann-White, Popoff dan Le Minor, (WHO, 2009) memanfaatkan kombinasi antigen O dan antigen H spesifik. Prinsip uji ini adalah aglutinasi antigen pada patogen-antibodi dalam antisera. Aglutinasi yang diharapkan terjadi (uji positif) pada SAT terbentuk karena antigen dan antisera berikatan. Proses aglutinasi memerlukan proporsi antisera dan suspensi bakteri yang tepat atau ekuivalen, agar dapat terlihat jelas dengan mata telanjang. Penetesan antisera dikendalikan dengan pipet tetes (lihat tanda panah pada Gambar 1a) yang melengkung dan menyempit di bagian ujungnya untuk memperkecil volume antisera yang diteteskan. (a) (b) Gambar 1. (a) Antisera; (b) Visualisasi aglutinasi [sebelah kiri pada slide] Kelebihan atau kekurangan salah satu di antara kedua komponen tersebut tidak akan menghasilkan ikatan yang kompak sehingga aglutinasi tidak terlihat jelas pada gelas objek. Visualisasi dari aglutinasi ditunjukkan oleh tanda panah pada Gambar 1b. Kekurangan dari metode ini ialah memerlukan pengalaman yang baik dari peneliti untuk melihat aglutinasi, menghabiskan antisera cukup banyak, memerlukan waktu pengujian serologi saja minimal tiga hari. Prosedur yang panjang dan diperlukannya waktu inkubasi di setiap tahapan menyebabkan waktu deteksi dengan metode konvensional hingga berhari-hari. Kelemahan 16 lain dari metode ini ialah diperlukan banyak alat gelas dan tenaga peneliti (tidak otomatis dengan instrumen). Saat ini telah banyak teknik yang dikembangkan untuk mempermudah pelaksanaan metode konvensional, misalnya: gravimetric diluter, pulsifier dan stomacher yang mempermudah homogenisasi; spiral plater, dipslide, dan petrifilm untuk enumerasi dan deteksi; colony counter dan kit uji untuk konfirmasi atau identifikasi (de Boer dan Beumer, 1999). Meskipun demikian waktu pendeteksian tidak berkurang secara signifikan hingga dikembangkanlah metode deteksi cepat. F. METODE DETEKSI CEPAT (RAPID METHOD) BAKTERI PATOGEN DENGAN PCR Perkembangan terkini dalam teknologi, secara teori membuat proses deteksi dan identifikasi bakteri patogen lebih cepat, tepat, sensitif, dan spesifik dibandingkan dengan metode konvensional (Anonim, 2001). Metode deteksi dengan teknik molekuler meliputi DNA probes, Polymerase Chain Reaction (PCR), Restriction Enzyme Analysis (REA), Random Amplification of Polymorphic DNA (RAPD), Pulsed Field Gel Electrophoresis (PFGE), dan Restriction Fragment Length Polymorphism (RFLP) (Jay, 1996). Metode-metode tersebut sering disebut sebagai metode cepat (rapid methods). Metode deteksi cepat patogen dan kontaminan mikrobiologi lainnya dalam makanan merupakan hal yang sangat penting untuk menjamin keamanan pangan konsumen. Keuntungan dari metode cepat dibandingkan dengan metode konvensional adalah waktu lebih singkat (4–48 jam), tidak membutuhkan banyak tenaga, serta lebih sensitif dan akurat (Patel dan Williams, 1994; Anonim, 2001). Sedangkan metode konvensional untuk mendeteksi patogen asal pangan bergantung pada waktu yang dibutuhkan untuk pertumbuhan bakteri dalam media kultur, yang diikuti oleh isolasi, identifikasi biokimia, dan terkadang harus diikuti dengan uji serologi. Metode uji berbasiskan DNA yang telah dikembangkan secara komersial untuk mendeteksi patogen asal pangan, antara lain: probes, PCR dan bakteriofage (Patel dan Williams, 1994). Metode paling populer adalah 17 amplifikasi DNA dengan Polymerase Chain Reaction (PCR) (de Boer dan Beumer, 1999). PCR adalah metode in vitro untuk memperbanyak sekuens DNA tertentu, perbanyakan tersebut seperti halnya replikasi DNA pada sel hidup. Lazimnya metode ini (PCR standar) memerlukan tahapan isolasi DNA, amplifikasi sekuens target dengan PCR, pemisahan produk amplifikasi (amplikon) dengan elektroforesis gel agarosa, dan memperkirakan ukuran fragmen dengan DNA ladder setelah pewarnaan dengan ethidium bromida. Pada bagian akhir, verifikasi hasil PCR dilakukan dengan pembelahan spesifik amplikon oleh endonuklease restriksi atau pemindahan amplikon terpisah ke dalam membran kemudian dihibridisasi dengan probe DNA spesifik. Alternatif verifikasi lain dengan sekuensing langsung menggunakan semi-nested PCR. Perkembangan PCR lainnya yang lebih nyaman digunakan ialah real-time PCR, di mana amplifikasi PCR dan verifikasi dilakukan sekali jalan dengan probe khusus (oligonukleotida berlabel fluoresen yang spesifik terhadap gen target). Real-time PCR melakukan amplifikasi dan deteksi dalam satu tahapan sebab akumulasi produk spesifik dicatat secara kontinyu selama siklus. Hal ini tidak dapat dilakukan pada PCR standar yang masih mengandalkan elektroforesis gel agarosa untuk mengkuantitasi amplikon. Hasil dari elektroforesis gel agarosa ini memperpanjang waktu deteksi dan kurang tepat karena pengukurannya berdasarkan ukuran molekul. Padahal molekul yang berbeda mungkin saja memiliki ukuran yang sama atau hampir sama. Pendeteksian dengan gel agarosa menganggap molekul yang berbeda tersebut sebagai molekul yang sama (Dharmaraj, 2009). Pendeteksian produk real-time PCR secara kuantitatif berbeda dengan PCR standar yang mengkuantitasi amplikon berdasarkan panjang basa atau bobot molekul. Kuantitas produk real-time PCR dihitung berdasarkan threshold cycle (Ct) yaitu waktu di mana intensitas fluoresen lebih besar daripada fluoresen yang ditimbulkan oleh noise (background fluorescence). Noise dapat disebabkan oleh penempelan larutan isolat DNA beserta pereaksi-pereaksi PCR pada dinding tabung microwell. 18 Format PCR standar yang berdasarkan analisis titik akhir (fase plato) kurang kuantitatif karena hasil akhir produknya tidak didasarkan konsentrasi sekuens target dalam sampel, tetapi berdasarkan pada ukuran molekul hasil amplifikasi (amplikon). Berikut ini beberapa kekurangan deteksi amplikon jika dilakukan pada fase plato dengan elektroforesis gel agarosa: kurang tepat, sensitivitas rendah, tidak otomatis, hanya berdasarkan ukuran molekul, pewarnaan ethidium bromida kurang sensitif, dan memperpanjang waktu deteksi (Dharmaraj, 2009). Keunggulan real-time PCR adalah pendeteksian diukur tepat pada saat target amplifikasi terdeteksi pertama kali di setiap siklus (fase eksponensial), bukan di fase akhir amplifikasi (fase plato) seperti yang terjadi pada PCR standar. Keunggulan real-time PCR lainnya ialah analisis dapat dilakukan tanpa membuka tabung sehingga mengurangi risiko kontaminasi amplikon PCR atau molekul target lainnya. Menurut Edwards et al. (2004), aplikasi teknologi real-time PCR mengurangi waktu penanganan atau pengujian dan meningkatkan keakuratan kuantifikasi metode PCR. Dengan demikian, penggunaan teknik real-time PCR lebih efisien dan efektif dibandingkan PCR standar (Edwards et al., 2004). Kuantifikasi real-time PCR dapat dilakukan dengan dua teknik: (1) penggunaan dye fluoresence yang berikatan dengan DNA untai ganda, dan (2) penggunaan probe oligonukleotida DNA modifikasi yang mengeluarkan fluoresen ketika hibridisasi dengan DNA komplementer. Data yang dihasilkan dapat dianalisis dengan perangkat lunak komputer yang terhubung dengan thermal cycler untuk menghitung jumlah kopi DNA atau threshold cycle (Ct) dari patogen dalam sampel pangan tertentu. Sebelum tahap amplifikasi dengan real-time PCR, diperlukan tahap persiapan, kemudian setelah tahap amplifikasi diperlukan evaluasi produk PCR. Tahap persiapan dan evaluasi tersebut dijelaskan lebih lanjut di bawah ini: 1. Tahap Pra-Amplifikasi Real-Time PCR Tahap pra-amplifikasi PCR standar dan real-time PCR tidak berbeda, meliputi persiapan sampel dan isolasi DNA untuk memperoleh 19 isolat DNA sebagai DNA target amplifikasi. Prosedur isolasi DNA merupakan tahapan yang paling banyak dimodifikasi sebelum amplifikasi dengan PCR dimulai. Terdapat enam tahap dalam mengisolasi DNA, yaitu preparasi sampel, pelisisan sel, proteksi dan stabilisasi DNA, pemisahan DNA dari debris sel, presipitasi DNA, dan pemekatan DNA. Prosedur yang umum digunakan untuk mengisolasi DNA kromosomal sebagai DNA target untuk uji amplifikasi dikembangkan oleh Sambrook et al. (1989). Prosedur isolasi DNA juga dikembangkan oleh Chapaval et al. (2008). Tahapan isolasi DNA prosedur-prosedur tersebut dapat diperbandingkan berdasarkan enam tahap dalam mengisolasi DNA. Enam tahap mengisolasi DNA berdasarkan Sambrook et al., (1989), sebagai berikut: a. Preparasi sampel Sampel pangan yang telah diinokulasi secara artifisial dan dihomogenisasi, kemudian diencerkan. b. Pelisisisan sel Pelisisan sel dilakukan dengan 10% sodium dodecylsulphate (SDS) dan 0.2 mg/ml proteinase K. Selanjutnya divorteks dan diinkubasi selama 2 jam pada suhu 65°C. c. Proteksi dan stabilisasi DNA Suspensi dari tahap persiapan sampel ditambahkan 500 µl buffer (100 mM NaCl, 500 mM Tris (pH 8.0) untuk menjaga kestabilan DNA ketika pelisisan sel. d. Pemisahan DNA dari debris sel dan protein DNA dipisahkan melalui ekstraksi dengan larutan fenol:kloroform (1:1 v/v, pH 8.0). Ekstraksi tersebut diulang hingga tiga kali dan setiap kali pengulangan fase aqueous dipindahkan ke tabung bersih. e. Presipitasi DNA Fase aqueous ditambah etanol 95% (mengandung 0.3 M sodium asetat) sebanyak 2.5 kali volume fase aqueous, diaduk, kemudian 20 diinkubasi semalam pada suhu -20°C untuk mengendapkan DNA kromosomal. f. Pemekatan DNA DNA di-recovery dengan sentrifugasi dan pelet DNA dicuci dua kali dengan etanol 70%. Enam tahapan mengisolasi DNA berdasarkan metode yang dikembangkan oleh Chapaval et al. (2008) adalah sebagai berikut: a. Preparasi sampel Sebanyak 2.5 ml kultur yang telah diinkubasi semalam dalam Brain Heart Infusion Broth (BHIB) disentrifus dengan kecepatan 33000 g selama 30 detik, kemudian supernatan dibuang dan pelet diresuspensi. b. Pelisisisan sel Pelet diresuspensi dalam 700 µl buffer ekstraksi (1.4 M NaCl; 100 mM Tris-HCl [pH 8.0]; 200 mM EDTA pH 8.0; 40% PVP (polyvinylpyrrolidone); 2% CTAB (cetyltrimethylammonium bromide), 20 mg/ml Proteinase K; 0.2% β-Mercaptoethanol). Tabung diinkubasi pada suhu 65°C selama 30 menit dengan pengocokan setiap 10 menit. c. Proteksi dan stabilisasi DNA Suspensi dari tahap persiapan sampel diberi buffer 1.4 M NaCl; 100 mM Tris-HCl pH 8.0 untuk menjaga kestabilan DNA ketika pelisisan sel. Buffer tersebut diberikan bersamaan dengan larutan pelisis sel. d. Pemisahan DNA dari debris sel dan protein Sebanyak 650µl kloroform:isoamil alkohol (24:1) ditambahkan dan larutan disentrifus pada kecepatan 33000 g selama 7 menit. Fase aqueous di bagian atas dipindahkan ke dalam sebuah tabung 1.5 ml dan 200 µl buffer ekstrasi tanpa proteinase K ditambahkan. Larutan diaduk perlahan dan 650 µl kloroform:isoamil alkohol (24:1) ditambahkan. Tabung kembali disentrifus dan ekstraksi 21 kloroform:isoamil alkohol (24:1) dilakukan dua kali menggunakan 650 µl kloroform:isoamil alkohol. e. Presipitasi DNA DNA dipresipitasi dengan penambahan sejumlah volume yang sama dengan isopropanol pada suhu ruang. f. Pemekatan DNA Isopropanol dihilangkan dan pelet dicuci dengan 70 µl etanol 70%. Pelet DNA dikeringudarakan dan diresuspensi dalam 40 µl buffer Tris-EDTA (TE) (10 mM Tris-HCl pH 8.0; 1 mM EDTA pH 8.0 dan 10 µg/ml RNAse). Prinsip dasar isolasi DNA adalah serangkaian proses untuk memisahkan DNA dari komponen-komponen sel lainnya. Hasil ekstraksi merupakan tahapan penting untuk langkah berikutnya. Oleh sebab itu, tahapan ini harus dilakukan dengan baik dan bebas kontaminasi. Isolasi yang banyak digunakan ialah ekstraksi dengan fenol:kloroform, seperti halnya kedua metode yang telah dijelaskan di atas. Teknik ekstraksi fenol:kloroform menghasilkan kualitas yang lebih baik dibandingkan dengan metode pendidihan (Fricker et al., 2007). Reagen lain yang penting dalam isolasi DNA ialah Phosphate Buffer Saline (PBS) yang berfungsi melarutkan matriks pangan sehingga berada dalam kondisi fisiologis, sekaligus mengendapkan inhibitor-inhibitor yang tidak diperlukan seperti kalsium. Buffer TE digunakan untuk menjaga tekanan osmotik dan Cetyltrimethyl Ammonium Bromida (CTAB), Sodium Dodecyl Sulphate (SDS), Ethylene Diamine Tetraacetic Acid (EDTA) berfungsi untuk melisis matriks pangan dan merusak lipid pada membran sel sehingga DNA lebih mudah diekstraksi (Anonim, 2008). Proteinase K digunakan untuk merusak protein dan enzim RNAse berfungsi untuk menghilangkan RNA sehingga dalam suspensi tertinggal hanya DNA. 22 2. Pembacaan Produk Hasil Real-Time PCR Amplifikasi yang dijalankan dalam thermal cycler ditampilkan dalam bentuk grafik pada layar komputer dengan software yang aplikabel terhadap thermal cycler, contohnya adalah software IQ-5 (Bio-Rad). Grafik-grafik yang diperoleh berupa grafik amplifikasi, kurva standar, dan kurva peleburan (melt curve). Grafik tersebut digunakan untuk mengevaluasi kinerja amplifikasi real-time PCR. Grafik amplifikasi Grafik amplifikasi terbentuk semenjak proses amplifikasi dimulai. Grafik ini berfungsi untuk menentukan apakah dalam thermal cycler terjadi amplifikasi atau tidak. Grafik amplifikasi dapat dilihat pada Gambar 2 di bawah ini. PCR Base Line Subtracted RFU a. DNA teramplifikasi Base line threshold Perpotongan siklus dan Bt Cycle DNA tidak teramplifikasi Ct Gambar 2. Grafik sigmoidal proses amplifikasi dengan real-time PCR (Edwards et al., 2004) Amplifikasi ditetapkan berdasarkan intensitas fluoresen, semakin banyak produk amplifikasi yang dihasilkan, semakin besar akumulasi fluoresen yang terbaca. Peningkatan fluoresen tersebut ditandai dengan terbentuknya gradik sigmoidal, seperti yang tergambar pada Gambar 2 (garis hijau). Grafik sigmoid akan berpotongan dengan base line threshold yang telah ditentukan secara otomatis oleh program. Titik perpotongan antara grafik sigmoid dan base line threshold jika direfleksikan terhadap sumbu x (Cycle) adalah threshold cycle (Ct) bagi sampel yang diamplifikasi. Garis 23 biru pada Gambar 2 akan terbentuk jika tidak terjadi amplifikasi pada thermal cycler. Nilai Ct adalah siklus di atas noise dimana akumulasi produk (senilai 2n, n ialah jumlah pengulangan siklus amplifikasi) terbaca pertama kali pada fase eksponensial. Fase eksponensial berakhir menjadi fase plato saat pereaksi dalam campuran reaksi PCR sudah habis. Nilai Ct digunakan dalam kurva standar sebagai fungsi terhadap log konsentrasi bakteri. Kurva standar Kultur murni bakteri yang telah diketahui konsentrasinya (CFU/ml) diencerkan hingga diperoleh konsentrasi bakteri terendah (1 CFU/ml). Seluruh tingkat pengenceran yang berturut-turut, misalnya mengandung 100-106 CFU/ml diisolasi DNAnya dan diamplifikasi. Amplifikasi beberapa tingkat pengenceran suatu kultur murni secara otomatis oleh software IQ-5 akan digambarkan dalam bentuk kurva standar, selain grafik amplifikasi. Kurva standar amplifikasi disajikan dalam Gambar 3. Kurva Standar Threshold cycle (Ct) b. Log konsentrasi bakteri (CFU/ml) Gambar 3. Kurva standar amplifikasi kultur murni dari enam tingkat pengenceran dengan real-time PCR (Edwards et al., 2004) Enam titik pada kurva standar menunjukkan terdapat enam tingkat pengenceran yang masing-masing memiliki nilai Ct. Kurva 24 standar merupakan hubungan antara log konsentrasi bakteri dan threshold cycle (Ct). Kurva ini digunakan dalam penetapan limit deteksi konsentrasi bakteri yang belum diketahui pada suatu sampel pangan. Penetapan limit deteksi tersebut memanfaatkan persamaan linear yang dibentuk kurva standar. Nilai Ct yang diketahui setelah isolat DNA diamplifikasi merupakan nilai y dan log konsentrasi merupakan nilai x dalam persamaan linear tersebut sehingga konsentrasi bakteri dalam suatu sampel pangan dapat diketahui. Melt curve Kurva peleburan atau melt curve merupakan kurva hubungan antara suhu (T) dan turunan dari unit fluoresen (-d(RFU)/dT) digunakan untuk memonitor perubahan dalam suhu peleburan, yaitu suhu di mana 50% amplikon DNA untai ganda terpisah menjadi dua untai tunggal. Melt curve diperoleh berdasarkan siklus yang ditambahkan setelah siklus amplifikasi. Target amplifikasi dalam siklus tersebut adalah produk amplifikasi (amplikon). Melt curve disajikan pada Gambar 4. -d(RFU)/dT c. Suhu (°C) Gambar 4. Melt curve yang memiliki sebuah puncak Tm Puncak kurva pada melt curve merupakan nilai Tm amplikon, jika nilainya dibaca pada sumbu absis. Amplikon yang berasal dari DNA target yang sama akan memiliki nilai Tm yang sama, 25 ditunjukkan dengan puncak pada titik suhu yang sama. Melt curve pada Gambar 4 memiliki nilai Tm yang sama, berarti produk berasal dari DNA yang sama yaitu DNA target dan produknya spesifik. Selain itu melt curve mampu menunjukkan ada atau tidaknya produk non spesifik. Produk non spesifik akan membentuk puncak pada suhu yang berbeda dengan amplikon (produk spesifik) atau melt curve dapat pula memiliki lebih dari satu puncak suhu. 26 III. METODOLOGI PENELITIAN A. TEMPAT DAN WAKTU MAGANG Kegiatan magang dilakukan di laboratorium Mikrobiologi Pusat Riset Obat dan Makanan, Badan POM RI, Jakarta. Waktu penelitian dilaksanakan dari bulan Februari sampai dengan Juli 2009. B. LINGKUP KEGIATAN PENELITIAN Kegiatan yang dilakukan mencakup dua kegiatan, yaitu (1) melakukan identifikasi serovar Salmonella spp. berbasiskan imunologi dan (2) melakukan deteksi B. cereus dengan metode berbasiskan DNA. Identifikasi serovar dengan cara uji aglutinasi pada gelas objek dilakukan terhadap sepuluh isolat Salmonella spp. yang merupakan koleksi Departemen Ilmu dan Teknologi Pangan (IPB) dan telah diuji secara biokimiawi. Sepuluh isolat tersebut diuji serologi untuk mengetahui antigen O yang dimiliki oleh masing-masing isolat sebab antigen O merupakan dasar pengelompokan serogrup Salmonella spp. Selain itu, dilakukan uji motilitas untuk mengetahui masing-masing isolat bersifat motil atau tidak. Antigen O merupakan antigen yang diuji pertama kali pada uji serologi, sedangkan motilitas diuji sebagai prasyarat uji antigen H. Diagram alir kegiatan deteksi Salmonella spp. dapat dilihat pada Gambar 5. Isolat Salmonella spp. Pre-enrichment dalam Trypticase Soy Broth (24 jam) Ditumbuhkan dalam Nutrient Agar (24 jam) Uji Serologi (Antigen O) Uji Motilitas dengan agar semi solid Gambar 5. Tahapan kerja pada identifikasi serovar Salmonella spp. 27 Kegiatan yang dilakukan untuk mendeteksi DNA B. cereus dengan realtime PCR meliputi isolasi DNA kultur murni bakteri B. cereus dan isolasi DNA dari sampel pangan nasi goreng, amplifikasi dan evaluasi kinerja realtime PCR berupa limit deteksi amplikon. Isolasi DNA dilakukan dengan tiga metode, yaitu metode pendidihan, metode ekstraksi dengan fenol:kloroform, dan metode ekstraksi dengan kit komersial. Isolat DNA B. cereus, dari kultur murni dan nasi goreng, diamplifikasi dengan real-time PCR. Amplifikasi DNA kultur murni B. cereus digunakan untuk membentuk kurva standar, sedangkan amplifikasi DNA B. cereus dalam sampel pangan untuk menetapkan limit deteksi B. cereus dalam nasi goreng. Tahapan kerja isolasi DNA dari kultur murni B. cereus dan dari sampel pangan disajikan pada Gambar 6 dan 7. Kultur murni (KM) B. cereus Pengenceran (10-1-10-8) Ditumbuhkan dalam MYPA Metode pendidihan Isolasi DNA Metode ekstraksi dengan fenol:kloroform Metode ekstraksi dengan kit komersial Amplifikasi Real-time PCR Gambar 6. Tahapan kerja pada uji amplifikasi DNA kultur murni B. cereus 28 Sampel pangan nasi goreng Inokulasi artifisial (KM 10-2) Sampel pangan tanpa inokulasi Pengenceran (10-1-10-6) Pengenceran (10-1) Isolasi DNA Metode pendidihan Metode ekstraksi dengan fenol:kloroform Amplifikasi Real-time PCR Gambar 7. Tahapan kerja pada uji amplifikasi DNA B. cereus dalam nasi goreng C. BAHAN DAN ALAT 1. Identifikasi Serovar Salmonella spp. a. Kultur bakteri Galur bakteri yang digunakan dalam uji serologi ialah Salmonella spp. yang diisolasi dari daging ayam yang berasal dari pasar lokal. Sepuluh isolat yang disimpan dalam Brain Heart Infusion Broth (BHIB) merupakan koleksi Departemen Ilmu dan Teknologi Pangan, Institut Pertanian Bogor dan telah ditetapkan sebagai Salmonella spp. berdasarkan uji biokimia. b. Bahan-bahan Bahan-bahan yang digunakan adalah Nutrien Agar (Merck) dan Antisera O (National Salmonella Centre – Statens Seruminstitut). c. Alat-alat Alat-alat yang digunakan adalah gelas objek (slide), cawan petri, pipet mikro, jarum ose, dan Laminar Air Flow (Vision). 29 2. Metode Deteksi B. cereus Berbasiskan DNA a. Kultur bakteri Galur referensi yang digunakan merupakan jenis bakteri Gram positif, Bacillus cereus ATCC 11778 dan Bacillus subtilis ATCC 6633 NCTC 10400. Bakteri-bakteri tersebut ditumbuhkan di Trypticase Soy Broth (TSB) (Merck) pada suhu 37°C. Sel bakteri dihitung dengan perhitungan cawan sebar (USFDA, 2001) pada media agar spesifik Mannitol Yolk Polymixin Agar (MYPA) (Oxoid) dan diinkubasi pada suhu 30°C selama 48-72 jam. Sampel pangan yang dianalisa adalah pangan yang banyak mengandung karbohidrat yaitu nasi goreng. Sampel diambil dari tempat penjualan makanan di sekitar kampus IPB, Bogor. b. Bahan-bahan Bahan-bahan yang digunakan adalah Ilustra Bacteria Genomic Prep Mini Spin Kit (GE Healthcare) yang terdiri dari: buffer lisis, buffer elusi, dan buffer pencuci. Selain itu juga digunakan Lisozim (USB), Proteinase K (USB), RNAse A (USB), CTAB (Merck), PBS (Merck), EDTA (Merck), Tris-HCl solution (USB), Triton-X solution (USB), SDS solution (USB), Isopropanol (Merck), Amonium asetat (Merck), Fenol (Merck), Kloroform (Merck), Asam asetat glasial (Merck), NaCl (Merck), Etanol absolut (Merck), MiliQ, RNAse free water (USB), IQ SYBR Green (Bio-Rad), Primer forward dan reverse BceT (Bio-Rad), isolat DNA yang diekstrak dari kultur murni B. cereus (pengenceran 10-1-10-8), isolat DNA dari sampel pangan yang tidak diinokulasi (pengenceran 10-1), isolat DNA yang telah diinokulasi secara artifisial (pengenceran 10-1-10-6). c. Alat-alat Alat-alat yang digunakan adalah Stomacher (Seward), Refrigerator Microsentrifuge (Hettich), Water Bath, Laminar Air Flow (Vision), Hot Plate (Thermolyne), Touch Mixer (Thermolyne), 30 Analytical Balance (Shimadzu), Autoklaf (Hirayama), Automatic Colony Counter (Acolyte), Lampu UV, kamera digital, tabung mikrosentrifus 1.5 ml, tabung konikal 15 ml (Iwaki), inkubator, bunsen, pipet mikro (Eppendorf), pipet volumemetrik, bulb, gelas piala, gelas ukur, labu Erlenmeyer, botol semprot, cawan petri, jarum ose, tabung reaksi, rak tabung reaksi, sudip, magnetic stirrer, gelas pengaduk, Real-time PCR (Bio-Rad), micro well, dan adhesive sealer. D. METODE PENELITIAN 1. Identifikasi Serovar Salmonella spp. (WHO, 2009) Konfirmasi Salmonella spp. dengan uji imunologi dilakukan untuk mendeteksi antigen dengan O Slide Agglutinasion Test (SAT) dan motilitas dengan agar semi solid. Mula-mula, sepuluh isolat Salmonella spp. dalam BHIB diinokulasi dan ditumbuhkan pada media NA, kemudian diinkubasi selama 24 jam pada suhu 35°C. Uji serologi antigen O dimulai dengan satu tetes antisera O polivalen diteteskan pada gelas objek, kemudian 1 ose Salmonella spp. diambil dari NA dan dicampur dengan jarum ose selama 1 menit. Gelas objek tersebut digoyang perlahan selama 1-2 menit. Uji positif ditunjukkan dengan adanya aglutinasi. Uji positif berarti antibodi pada sera spesifik berikatan dengan antigen yang dimiliki bakteri. Aglutinasi tidak akan terjadi jika bakteri tidak memiliki antigen yang dapat berikatan dengan antibodi. Uji motilitas dilakukan pada hari yang sama dengan pengujian antigen O. Satu ose Salmonella spp. diambil dari NA, kemudian diinokulasi satu titik pada bagian tengah agar semi solid dan diinkubasi semalam pada suhu 37°C. Isolat yang menunjukkan pergerakan (sekitar 40 mm dari titik inokulasi atau lebih dari 50% jari-jari cawan agar) ke tepi agar semi solid dikonfirmasi bersifat motil. 31 2. Metode Deteksi B. cereus Berbasiskan DNA (Alarcon et al., 2006; Fricker et al., 2007) a. Tahap persiapan 1) Persiapan media Media yang perlu dipersiapkan untuk penyegaran kultur bakteri adalah media Trypticase Soy Broth. Media selektif untuk isolasi bakteri adalah Mannitol Egg Yolk Polymyxin Agar (media selektif B.cereus), Egg Yolk Emulsion serta Polymyxin B (untuk menghambat pertumbuhan bakteri Gram negatif). Media selektif digunakan untuk isolasi dan penghitungan kultur bakteri patogen, baik kultur murni maupun kultur dari sampel pangan. Prosedur pembuatan media dapat dilihat pada Lampiran 2 dan 3. 2) Persiapan kultur bakteri Bacillus cereus disegarkan pada media TSB setiap 2 minggu sekali dan diinkubasi pada suhu 37ºC selama 24 jam, lalu disimpan pada suhu 16°C. Untuk pengecekan kemurnian kultur, kultur B. cereus dalam TSB diinokulasi sebanyak 1 lup ke permukaan agar Mannitol Egg Yolk Polymyxin Agar (MYPA) padat dalam cawan petri steril. Koloni tipikal B. cereus pada MYPA dengan suplementasi polymyxin B akan berwarna pink cerah dan dikelilingi zona presipitasi. Selain bakteri patogen B. cereus, disiapkan juga B. subtilis yang digunakan untuk menguji spesifisitas primer BceT. Primer BceT merupakan DNA dari gen penyandi enterotoksin T pada DNA kromosomal B. cereus. Isolat DNA bakteri tersebut diamplifikasi kemudian melt curve yang terbentuk dianalisis untuk mengetahui apakah primer BceT dapat menempel pada DNA kromosomal selain DNA gen penyandi enterotoksin B. cereus yang berarti primer kurang spesifik. Bakteri ini pun disegarkan setiap 2 minggu sekali dalam TSB dan diinkubasi 32 pada suhu 37ºC selama 24 jam, lalu disimpan pada suhu 16°C. Prosedur persiapan kultur bakteri dapat dilihat pada Lampiran 4. B. cereus dan B.subtilis disegarkan dan diinkubasi pada suhu 37ºC selama 24 jam sehari sebelum DNA diisolasi. Kultur murni B. cereus yang hendak diisolasi diencerkan terlebih dahulu (10-1-10-8), sedangkan kultur murni B. subtilis diambil 1 ml dan langsung diisolasi. Pengenceran serial kultur murni B. cereus dilakukan untuk diisolasi DNA-nya sebanyak 1 ml dari masing-masing pengenceran dan untuk ditumbuhkan dalam MYPA sebanyak 0.1 ml. Penumbuhan kultur bakteri dalam MYPA dilakukan dengan teknik surface plate untuk perhitungan konsentrasi bakteri yang terdapat pada kultur murni (CFU/ml). Jumlah koloni pada beberapa tingkat pengenceran dan konsentrasi bakteri kultur murni B. cereus dapat dilihat pada Lampiran 5. Konsentrasi tersebut digunakan sebagai perhitungan log konsentrasi bakteri dalam kurva standar amplifikasi. 3) Persiapan sampel pangan Sampel pangan yaitu nasi goreng dibagi menjadi dua, masing-masing sejumlah 25 gram dan salah satunya diinokulasi B. cereus dengan konsentrasi 8.8 x 105 CFU/g (konsentrasi diperoleh dari perhitungan koloni dalam MYPA). Bagian sampel pangan yang tidak diinokulasi disebut sampel pangan (SP), sedangkan bagian yang diinokulasi secara artifisial dengan kultur murni B. cereus disebut sampel pangan spike (SPS). Baik SP (25 gram) maupun SPS (25 gram) dimasukkan ke dalam plastik steril yang masing-masing berisi 225 ml NaCl 0.85% dan dihomogenisasi dengan stomacher (30 detik, 230 rpm), lalu dituang ke dalam botol sampel. Setelah itu, SP diambil 1 ml dan diisolasi DNA-nya, sedangkan SPS diencerkan hingga tingkat pengenceran 10-6 kemudian dari seluruh tingkat pengenceran 33 diambil 1 ml untuk diisolasi DNA-nya dan diambil 0.1 ml untuk diiinokulasikan pada MYPA dan dihitung koloni B. cereus yang tumbuh. Pengamatan dan penghitungan koloni dilakukan setelah MYPA diinkubasi (37ºC) selama 2 hari. Perhitungan koloni tipikal B. cereus mengikuti hitungan cawan (USFDA, 2001). Koloni tipikal B. cereus pada MYPA adalah koloni pink cerah dan dikelilingi zona presipitasi. Cawan yang digunakan untuk perhitungan ialah cawan dengan koloni tipikal berkisar antara 15-150 koloni. Perhitungan jumlah koloni per ml dilakukan dengan Standard Plate Count (BAM edisi ke-8) dengan rumus sebagai berikut: Keterangan: N = jumlah koloni per ml atau per g produk ∑C = jumlah semua koloni yang dihitung n1 = jumlah cawan pada pengenceran pertama n2 = jumlah cawan pada pengenceran kedua d = pengenceran pertama yang dihitung b. Tahap isolasi DNA Sebanyak 1 ml dari masing-masing pengenceran diisolasi DNAnya dengan tiga metode, yaitu (1) Metode pendidihan, (2) Metode ekstraksi dengan fenol:kloroform, dan (3) Metode ekstraksi dengan kit komersial. Metode pendidihan dan metode ekstraksi dengan fenol:kloroform digunakan untuk perbandingan hasil amplifikasi dengan kit komersial dalam uji sensitivitas dan spesifisitas. Berikut ini uraian prosedur kerja dalam ketiga metode tersebut. 1) Metode pendidihan (Hein et al., 2001 dengan modifikasi) Kultur murni atau sampel pangan yang telah dihomogenisasi dan dilakukan pengenceran (10-1-10-8) diambil masing-masing 1 ml, lalu disentrifus. Supernatan dibuang dan pelet dicuci dengan 500 µl buffer TE 1X sebanyak dua kali, disentrifus, kemudian pelet disuspensikan dengan buffer TE 1X 34 dan lisozim, diinkubasi pada suhu ruang (25°C) selama 15 menit, ditambahkan proteinase K, setelah itu diinkubasi pada suhu 55°C selama 1 jam, kemudian dididihkan selama 15 menit, didinginkan dalam freezer (4°C) selama 30 menit, dan supernatan digunakan sebagai sumber DNA target. Prosedur kerjanya dapat dilihat pada Lampiran 6. Modifikasi yang dilakukan terhadap metode pendidihan (Hein et al., 2001) adalah waktu sentrifugasi dari 5700 g selama 15 menit menjadi 14000 g selama 10 menit untuk menjamin seluruh bakteri telah menjadi pelet, kemudian setelah pendidihan 100°C selama 15 menit, suspensi DNA tidak disentrifus, melainkan segera didinginkan dalam freezer (4°C) selama 30 menit untuk merenaturasi DNA yang telah terdenaturasi saat dididihkan. 2) Metode ekstraksi dengan fenol:kloroform (Sambrook et al., 1989 dengan modifikasi) Kultur murni atau sampel pangan yang telah dihomogenisasi dan dilakukan pengenceran (10-1-10-8) diambil masing-masing 1 ml, lalu disentrifus dan pelet diresuspensi dengan 500 µl buffer TE 1X sebanyak 2 kali. Setelah disentrifus, pelet diresuspensi dengan 500 µl buffer TE 1X dan 100 µl lisozim, diinkubasi pada suhu 4°C selama 5 menit. Kemudian ditambahkan 25 µl SDS 10%, 50 µl NaCl 5M, dan 100 µl proteinase K (20 mg/ml), divortex dan diinkubasi pada suhu 55°C selama 2 jam. Selanjutnya ditambahkan 500 µl fenol:kloroform (1:1 v/v), divortex dan diinkubasi -20°C selama 30 menit, lalu disentrifus pada suhu 4°C, 12000 rpm selama 10 menit. Lapisan air bagian atas diambil dan dipindahkan ke tabung eppendorf baru, ditambahkan 500 µl fenol:kloroform (1:1 v/v), divortex dan diinkubasi -20°C selama 30 menit, lalu disentrifus pada suhu 4°C, 12000 rpm selama 10 menit. Lapisan 35 air bagian atas diambil dan dipindahkan ke tabung eppendorf baru, ditambahkan 500 µl kloroform, disentrifus pada suhu 4°C, 12000 rpm selama 10 menit. Lalu lapisan air bagian atas dipindahkan ke tabung eppendorf baru, ditambahkan 0.3 kali volume lapisan air ammonium asetat 10M pH 7.4 dan isopropanol dingin 1 kali volume fase aqueous, diinkubasikan -20°C selama semalam, kemudian disentrifus 14000 rpm selama 30 menit. Selanjutnya pelet ditambah dengan 500 µl etanol 70% dan disentrifus 12000 rpm selama 5 menit. Supernatan dibuang dan pelet dikeringudarakan, kemudian dilarutkan dalam 50 µl buffer TE 1X. Prosedur kerja metode ekstraksi dengan fenol:kloroform ini dapat dilihat pada Lampiran 7. Modifikasi yang dilakukan terhadap metode pendidihan (Hein et al., 2001) adalah buffer lisis yang digunakan pada metode Sambrook et al. (1989) pada 1 ml kultur bakteri diganti dengan buffer TE untuk menjaga tekanan osmosis bakteri, campuran buffer lisis pada Sambrook et al. (1989) yaitu 100 mM NaCl, 500 mM Tris (pH 8.0), 10% sodium dodecylsulphate, dan 0.2 mg/ml proteinase K diganti dengan campuran buffer yang diberikan bertahap, terdiri atas 500 µl buffer TE 1X dan 100 µl lisozim untuk merusak dinding sel bakteri, kemudian ke dalam campuran buffer dan kultur bakteri ditambahkan 25 µl SDS 10%, 50 µl NaCl 5M, dan 100 µl proteinase K (20 mg/ml) untuk menhilangkan protein yang dapat mengganggu amplifikasi. Modifikasi juga dilakukan pada komposisi pelarut fenol:kloroform (1:1 v/v) sebagai ganti dari pelarut fenol:kloroform:isoamil alkohol (25:24:1 v/v/v). Kloroform biasanya ditambahkan dengan isoamil alkohol dengan perbandingan 24:1 untuk mengurangi busa yang terbentuk ketika ekstraksi. Campuran senyawa ini sering disebut CIAA (Chloroform Isoamyl Alcohol). Pelarut ini dimodifikasi karena keterbatasan pelarut isoamil alkohol. Fase aqueous dari 36 ekstraksi dengan fenol:kloroform pada metode Sambrook et al. (1989) ditambahkan 0.3 M Natrium Asetat pH 4.8, namun pada metode yang dimodifikasi garam yang ditambahkan adalah 10 M Ammonium Asetat pH 7.4 disesuaikan dengan garam yang tersedia. Kedua garam tersebut berfungsi sama dan dapat saling menggantikan. Selain itu, etanol absolut pada metode Sambrook et al. (1989) diganti dengan isopropanol karena isopropanol lebih cepat mempresipitasikan DNA, lalu waktu dan kecepatan sentrifus diganti dari 15 menit menjadi 30 menit untuk memperoleh pelet DNA karena waktu 15 menit tidak cukup untuk mengendapkan DNA di dasar tabung. Pelet yang dikeringvakumkan diganti dengan cara dikeringudarakan karena keterbatasan instrumen. 3) Metode ekstraksi sesuai panduan kit komersial Sebanyak 1 ml kultur murni B. cereus dari delapan tingkat pengenceran masing-masing dimasukkan ke dalam tabung mikrosentrifus dan disentrifus dengan kecepatan 14000 rpm selama 3 menit. Supernatan yang terdapat dalam tabung dibuang tanpa merusak pelet, lalu ditambahkan 40 µL buffer lisozim dan segera divorteks selama 10 detik sampai sel bakteri benar-benar tersuspensi, kemudian suspensi tersebut ditambahkan 20 µL lisozim, divorteks dan diinkubasi selama 5 menit pada suhu kamar 25ºC, lalu disentrifus 5000 rpm selama 5 detik untuk mengumpulkan sampel di dasar tabung. Inkubasi dilanjutkan pada suhu kamar (25ºC) selama 5 menit. Kemudian 20 µL Proteinase K ditambahkan ke dalam sampel, lalu divorteks. Setelah itu, ditambahkan 10 µL buffer lisis, divorteks dan disentrifus 5000 rpm selama 5 detik. Suspensi diinkubasi pada suhu 55oC selama 7 menit, divorteks selama 10 detik dan disentrifus 5000 rpm selama 5 detik. Proses inkubasi dilanjutkan pada suhu 55oC selama 8 menit, kemudian disentrifus 5000 rpm 37 selama 5 detik. Lalu ditambahkan 5 µL RNAse-A, divorteks dan disentrifus 5000 rpm selama 5 detik. Sampel kembali diinkubasi pada suhu kamar (25ºC) selama 15 menit. Setelah itu, ditambahkan 500 µl buffer lisis, divorteks selama 10 detik dan inkubasi pada suhu kamar (25ºC) selama 5 menit. Setelah itu, cairan dipindahkan ke dalam kolom mini yang diletakkan di dalam collection tube dan disentrifus pada 15000 rpm selama 1 menit, lalu cairan pada collection tube dibuang dan kolom diletakkan pada collection tube. Sebanyak 500 µL buffer lisis ditambahkan ke dalam kolom mini dan disentrifus 15000 rpm selama 1 menit, kemudian cairan pada collection tube dibuang kembali dan kolom diletakkan kembali ke dalam collection tube. Setelah itu, ke dalam kolom mini ditambahkan 500 µL wash buffer dan disentrifus dengan kecepatan 14000 rpm selama 3 menit, kemudian collection tube dan kolom mini dipindahkan ke dalam tabung mikrosentrifus baru. Sebanyak 200 µL buffer elusi (70oC) ditambahkan ke dalam matriks fiber dalam kolom mini, lalu diinkubasi pada suhu kamar (25ºC) selama 1 menit dan disentrifus pada 14000 rpm selama 1 menit. Cairan pada tabung mikrosentrifus digunakan sebagai isolat DNA. Prosedur kerja metode isolasi DNA dengan kit komersial dapat dilihat pada Lampiran 8. c. Tahap amplifikasi (Alarcon et al., 2006 dengan modifikasi) Sebanyak 2.0 µl isolat DNA, yang diperoleh dari 8 tingkat pengenceran untuk kultur murni, 1 tingkat pengenceran SP, dan 6 tingkat pengenceran SPS, digunakan sebagai templat untuk amplifikasi PCR. Isolat DNA (2.0 µl) dicampur dengan 9.5 µl RNAse free water, 12.5 µl SYBR Green supermix, 0.5 µl masingmasing forward dan reverse primer BceT. Volume total larutan adalah 25 µl. 38 Larutan tersebut diamplifikasi dengan kondisi, sebagai berikut: predenaturasi 95°C selama 10 menit, denaturasi pada suhu 95°C selama 10 detik dan penempelan pada suhu 55°C selama 30 detik (40 siklus) dengan thermal cycler iCycler IQ System, dilanjutkan dengan 80 siklus (suhu 55°C selama 10 detik) untuk menghasilkan melt curve yang digunakan dalam spesifisitas uji. Kondisi amplifikasi menurut Alarcon et al. (2006) yaitu denaturasi pada suhu 95°C selama 15 detik dan penempelan pada suhu 60°C selama 1 menit dengan thermal cycler GeneAmp 5700 Sequence Detection System dimodifikasi menjadi kondisi seperti yang dijelaskan di atas untuk menyesuaikan dengan jenis primer dan thermal cycler yang digunakan sehingga amplifikasi dapat berlangsung. Primer (forward dan reverse) BceT dianalisis dengan Basic Local Alignment Search Tool (BLAST, NCBI) untuk mengetahui spesifitasnya terhadap gen target. Urutan nukleotida sepasang primer tersebut adalah 5’-GAA TTC CTA AAC TTG CAC CAT CTC G-3’ untuk forward primer yang terletak pada urutan basa nukleotida 2208-2232 pada DNA kromosomal B. cereus dan 5’-CTG CGT AAT CGT GAA TGT AGT CAA T-3’ reverse primer yang terletak pada urutan basa nukleotida 1587-1611 pada DNA kromosomal B. cereus. Produk amplifikasi dari sepasang primer ini adalah 646 pasang basa. d. Evaluasi kinerja real-time PCR Amplifikasi real-time PCR dilakukan untuk menetapkan limit deteksi kultur murni B. cereus dan limit deteksi B. cereus pada nasi goreng. Selain itu, real-time PCR dapat digunakan untuk menghasilkan melt curve. Kurva tersebut dipakai dalam spesifisitas uji. Kinerja real-time PCR dievaluasi dengan cara menetapkan limit deteksi, sensitivitas dan spesifisitas uji. 39 1) Limit deteksi Isolat DNA kultur murni dengan beberapa tingkat pengenceran diamplifikasi dan diperoleh nilai Ct dari masingmasing pengenceran serta kurva standar. Pengenceran tertinggi yang masih dapat teramplifikasi merupakan limit deteksi kultur murni B. cereus dalam penelitian ini. Kurva standar memberikan persamaan linear yang digunakan untuk mengukur limit deteksi B. cereus dalam sampel pangan. Persamaan linear kurva standar digunakan untuk menghitung konsentrasi bakteri yang belum diketahui dalam sampel pangan. Konsentrasi B. cereus pada sampel pangan nasi goreng dapat dihitung dengan memasukkan nilai Ct hasil amplifikasi sebagai nilai y pada persamaan linear, kemudian nilai yang diperoleh diantilogkan. Limit deteksi dengan real-time PCR memiliki satuan CFU/ml atau CFU/g. Oleh karena itu, setiap isolasi DNA dilakukan, pencawanan pada media selektif dari beberapa pengenceran juga dilakukan untuk mengetahui jumlah koloni pada kultur murni atau pun koloni pada sampel pangan. Prosedur penetapan limit deteksi bakteri patogen dengan PCR dapat dilihat pada Lampiran 9. 2) Spesifisitas uji Primer BceT diuji spesifisitasnya dengan cara membandingkan melting temperature (Tm) dari puncak melt curve yang terbentuk antara amplikon yang diperoleh dari isolat DNA B. cereus dan amplikon yang diperoleh dari isolat DNA B. subtilis. Hasil amplifikasi (amplikon) yang spesifik ditunjukkan dengan nilai Tm yang sama pada puncak melt curve. Tm merupakan suhu peleburan di mana 50% DNA untai ganda telah terdenaturasi menjadi DNA untai tunggal. Secara teoritis, Tm amplikon dari isolat DNA B. cereus dan B. subtilis berbeda karena DNA target merupakan DNA spesifik dari B. cereus. 40 IV. HASIL DAN PEMBAHASAN A. IDENTIFIKASI SEROVAR Salmonella spp. Isolat Salmonella yang digunakan telah dikonfirmasi dengan API 20E dan teridentifikasi sepuluh isolat tersebut sebagai Salmonella spp. Melalui uji serologi Salmonella spp. dapat lebih dispesifikkan menjadi subspesies yang berbeda berdasarkan antigen-antigen yang dimiliki masing-masing isolat. Pengujian serologi penting karena struktur molekul protein atau polisakarida setiap bakteri berbeda dan juga struktur antigennya. Bagi genus Salmonella, uji serologi merupakan standar metode deteksi konvensional. Hasil uji serologi terhadap antigen O dan motilitas Salmonella spp. dapat dilihat pada Tabel 3. Tabel 3. Data uji serologi antigen O dan motilitas No. Spesimen Uji Serologi Uji Motilitas(*) 1. Gb OMF Motil 2. PB5 O:8 Motil 3. PGB3 O:1,4,12 Motil 4. PGB5 O:12 Motil 5. PKS1 O:8 Tidak motil 6. PKS2 O:5 Motil 7. PL21 O:5 Tidak motil 8. PL22 O:6,7,14 Tidak motil 9. PM3 O:8 Tidak motil 10. RGB1 O:1,4,12 Motil Keterangan: (*) Motil jika koloni yang seragam meluas ke tepi agar semi solid (>50% jari-jari cawan) Uji serologi antigen O diikuti dengan uji motilitas karena uji motilitas merupakan bagian dari uji serologi. Uji motilitas harus dilakukan terlebih dahulu sebelum antigen H (flagela) diuji untuk memastikan bahwa isolat Salmonella yang akan diuji antigen H-nya memiliki flagela (motil). Uji 41 serologi yang lengkap, seharusnya meliputi uji serologi terhadap antigen (O) somatik, antigen Vi, dan antigen (H) flagelar. Namun, serotyping yang dilaksanakan di Pusat Riset Obat dan Makanan dibatasi pada tahap uji antigen O, disesuaikan dengan antisera yang tersedia. Uji antigen Vi tidak dilakukan sebab antisera tidak tersedia, sehingga penentuan apakah bakteri tersebut lebih patogen (jika uji positif) atau tidak belum dapat terlaksana, sedangkan uji antigen H tidak dilaksanakan karena antisera tidak lengkap. Padahal dalam pengujian aglutinasi antigen-antisera diperlukan seluruh jenis antisera individual. Kelengkapan tersebut penting karena kerja dilakukan bertahap, apabila terdapat antigen-antisera yang tidak menunjukkan aglutinasi, kemudian dapat dilanjutkan dengan antisera lainnya hingga diperoleh aglutinasi. Jika ada tahapan uji yang dilewati maka kespesifikan antigen pada patogen tidak dapat dipastikan. Uji antigen O dilakukan pada Salmonella karena bakteri ini termasuk Gram negatif. Bakteri Gram negatif memiliki membran luar yang memiliki lipopolisakarida, di mana antigen O merupakan bagian dari lipopolisakarida tersebut. Oleh karena itu, antisera mudah bereaksi dengan antigen karena letaknya di permukaan membran luar sel bakteri. Uji serologi pada spesimen Gb hanya dapat dilakukan sampai pada antigen O serogrup OMF (O Multi-phase F sera), karena antisera O individual bagi grup F tidak tersedia. Antisera individual berarti sera tersebut hanya mengandung satu faktor tunggal antigen, berbeda dengan antisera polivalen yang memiliki beberapa faktor tunggal antigen. Serogrup F jarang teridentifikasi sebagai penyebab penyakit pada manusia atau pun sapi. Sedangkan serogrup yang diidentifikasi banyak menyebabkan penyakit pada anak sapi, susu sapi, dan manusia berasal dari Salmonella serogrup B, C, D, dan E (Jayarao dan Henning, 2001). Spesimen PB5, PKS1, dan PM3 teridentifikasi memiliki antigen O:8, spesimen PKS2 dan PL2 memiliki antigen O:5, dan spesimen PGB5 memiliki antigen O:12. Terdapat pula spesimen yang teridentifikasi memiliki antigen O lebih dari satu faktor tunggal, yaitu spesimen PGB3 dan RGB1 dengan serovar O:1,4,12 dan spesimen PL22 dengan serovar O:6,7,14. 42 Hasil serotyping terhadap kesepuluh isolat menujukkan Salmonella dengan serovar O:12 paling banyak ditemukan pada isolat Salmonella spp. dalam daging ayam, yakni dari spesimen PGB3, PGB5, dan RGB1. Serovar O:12 terdapat pada serogrup B, anggota serogrup B yang dilaporkan menjadi sumber penyakit di Indonesia, yaitu Salmonella enterica subspesies enterica serovar Typhimurium dan S. Paratyphi B. Penamaan S. Typhimurium berdasarkan hasil uji serologi menjadi I 4,5,12:i:1,2, artinya galur S. Typhimurium merupakan spesies Salmonella enterica subspesies enterica yang memiliki antigen O:4, O:5, dan O:12, antigen H fase 1 (i) dan antigen H fase 2 (1 dan 2). Anggota genus Salmonella mempunyai struktur antigen yang tidak stabil dan dapat mengalami perubahan sewaktu-waktu dan bakteri ini pada suatu saat dapat membentuk variasi secara tiba-tiba (Poernomo, 2004). Galur Salmonella yang awalnya teridentifikasi memiliki antigen O, bisa saja ketika diuji ulang mengalami autoglutinasi (rough strain of Salmonella), galur ini ialah galur yang telah kehilangan sifat antigen O-nya. Oleh karena itu pada setiap awal tahapan uji serologi perlu dilakukan suspensi bakteri dengan NaCl 0.85%, jika suspensi menggumpal maka bakteri tersebut telah mengalami autoglutinasi dan perlu disubkulturkan pada Agar darah atau Agar MuellerHinton untuk me-recovery kondisi smooth (mengandung struktur antigen O). Seluruh isolat Salmonella spp. saat pengujian autoglutinasi menunjukkan reaksi negatif, maka uji serologi dilanjutkan dengan menggunakan antisera polivalen O. Jika aglutinasi tidak terjadi dengan antisera Polivalen O berarti bakteri bukan Salmonella dan serotyping tidak perlu dilakukan. Antibodi pada sera spesifik akan menggumpal dengan bakteri yang memiliki antigen sesuai (CDC, 2009). Setelah uji antigen O dilaksanakan, sepuluh isolat harus ditumbuhkan dalam medium agar semi solid, Swarm Agar, untuk melihat motilitasnya. Jika Salmonella motil, berarti bakteri tersebut berflagela dan memiliki antigen H yang harus diuji lebih lanjut. Swarm Agar yang diprasyaratkan oleh WHOCDC (2009), tersusun atas TSB, KNO3, dan agar. Karena media ini tidak tersedia di laboratorium, dibuatlah media agar semi solid dari Nutrien Agar, 43 Nutrien Broth, Beef extract, dan KNO3 untuk uji motilitas. Modifikasi tersebut tetap memberi komposisi yang sama dengan medium uji motilitas (M103, BAM) dan motilitas dapat diuji. Perpindahan tipikal dari Salmonella ialah yang menunjukkan pergerakan sejauh 40 mm dari titik/tempat yang telah diinokulasi. Pergerakan bakteri dapat dilihat dari koloni yang menyebar dari inokulasi satu titik di tengah agar menjadi melebar ke tepi agar. Bakteri yang dianggap layak untuk dilakukan uji serologi antigen H ialah bakteri (koloni seragam) yang bergerak ke tepi melebihi setengah dari jari-jari agar semi solid. Jika terdapat pergerakan menuju tepi agar, berarti bakteri tersebut berflagela dan secara teoritis memiliki antigen H. Apabila tidak terdapat pergerakan, padahal uji morfologi dan biokimia mengidentifikasi secara umum bahwa bakteri uji ialah Salmonella, inkubasi perlu dilanjutkan selama 24 jam (35°C). Jika tetap tidak ada pergerakan bakteri pada medium semi solid, bakteri dalam medium tersebut diinkubasi kembali pada suhu 25°C selama 5 hari. Hasil pengamatan terhadap kesepuluh isolat yang diinokulasi pada agar semi solid dan diinkubasi selama satu hari, isolat yang menunjukkan motilitas tipikal ialah spesimen Spesimen Gb, PB5, PGB5, PKS2, RGB1, dan PGB3. Visualisasi uji motilitas isolat Salmonella spp. dapat dilihat pada Lampiran 10. Spesimen PKS1, PL21, PL22, dan PM3 walaupun tidak motil, masih dapat diasumsikan sebagai Salmonella spp. karena tidak semua Salmonella motil. Ada beberapa spesies Salmonella yang tidak mempunyai antigen (H) flagela (tidak motil), misalnya S. Pullorum dan D. Gallinarum (Gast, 2003). Menurut Gast (2003), lebih dari 2400 subspesies S. enterica bersifat motil. Oleh karena itu, uji serologi tidak cukup hanya pada tahap uji antigen O, tetapi harus dilengkapi dengan uji antigen H fase 1 dan 2 sehingga identifikasi serovar lengkap. Kelebihan dilakukannya uji serotipe ialah identifikasi Salmonella spp. lebih lengkap, penulisan serovar yang jelas dan diterima oleh sebagian besar negara di dunia, penyediaan data bagi kasus epidemik Salmonella dan dapat diperbandingkan dengan subspesies Salmonella yang juga pernah menyebabkan epidemik di negara-negara lain. Keuntungan diketahuinya 44 serovar melalui uji serologi ini ialah kemudahan dalam penelusuran bakteri Salmonella yang menjadi sumber penyakit asal pangan atau kasus epidemik di Indonesia dan kemudahan memperbandingkan serovar Salmonella spp. dengan negara lain. Menurut WHO dalam Global Salmonella Surveillance (2009), serotyping dapat menjadi metode terbaik dalam penetapan fenotip bakteri sebab metode ini memiliki kemampuan membedakan subspesies secara teliti dan memberikan informasi serotipe yang penting bagi kasus-kasus epidemik. Kemudahan penerapan dan pelaksanaan metode deteksi ini menjadikan SAT dapat diaplikasikan oleh pihak yang hendak melakukan uji serologi. Kendala utama bagi terlaksananya uji ini secara luas ialah biaya yang tinggi dalam penyediaan antisera. Metode lain, seperti sistem molekuler yang saat ini berkembang pesat tidak dapat menggantikan uji serotipe yang bekerja dengan prinsip aglutinasi antisera dan antigen. B. DETEKSI B. cereus BERBASISKAN DNA 1. Tahap Persiapan Sampel Tahap pengkulturan sangat penting dilakukan untuk menyediakan kecukupan jumlah bakteri B. cereus awal yang hendak diinokulasi. Pengkulturan dilakukan dalam medium cair Trypticase Soy Broth (TSB), yaitu medium yang berfungsi membiakkan bakteri atau memperkaya bakteri karena memiliki nutrisi yang menunjang pertumbuhan bakteri. Kultur murni bakteri B. cereus digunakan untuk menentukan limit deteksi kultur murni dan menghasilkan kurva standar. Pengkulturan bakteri dilakukan pada medium TSB dan medium BHIB. Setelah dicawankan ternyata pertumbuhan bakteri lebih baik pada medium BHIB. Oleh karena itu, sebaiknya untuk pengkulturan bakteri atau pun pengkayaan sebelum isolasi DNA menggunakan medium BHIB. Medium BHIB menyediakan lebih banyak nutrisi yang mendukung pertumbuhan bakteri dibandingkan dengan TSB. Media TSB termasuk media non-selektif sehingga semua bakteri dapat tumbuh. Apabila kerja tidak aseptik, bakteri selain B. cereus 45 dapat tumbuh. Oleh karena itu, untuk menjamin kemurnian biakan kultur murni B. cereus, uji morfologi untuk melihat keseragaman dan koloni tipikal B. cereus dilakukan dalam MYPA beberapa hari sebelum isolasi DNA hendak dilaksanakan. Apabila kultur murni yang dicawankan pada MYPA hanya membentuk koloni tipikal pink cerah dengan zona presipitasi di sekelilingnya, seperti pada Gambar 8, berarti kultur murni masih seragam dan tidak terkontaminasi bakteri lain sehingga dapat dilakukan isolasi DNA. Gambar 8. Koloni tipikal B. cereus pada MYPA + Polymyxin B Media MYPA merupakan media selektif B. cereus. MYPA mengandung manitol yang tidak dapat difermentasi oleh B. cereus, namun oleh beberapa bakteri lain (misalnya B.subtilis dan S. aureus) dapat difermentasi. Penambahan kuning telur dapat memperjelas koloni tipikal B. cereus sebab kuning telur memiliki lesitin dan B. cereus menghasilkan enzim fosfatidilkolin hidrolase, fosfolipase C yang menghidrolisis lesitin. Dengan begitu, koloni tipikal B. cereus akan mendegradasi lesitin yang terlihat sebagai zona presipitasi di sekeliling koloni (Schraft dan Griffiths, 1995). Polymyxin B juga ditambahkan pada MYPA untuk menghambat pertumbuhan bakteri Gram negatif. 46 Konsentrasi bakteri biakan kultur murni (KM), dihitung dari pencawanan MYPA 8.8 x 108 CFU/ml dan pengenceran KM 10-2 digunakan 0.1 ml untuk menginokulasi nasi goreng secara artifisial. Dengan asumsi, sampel pangan spike berkonsentrasi kurang lebih 106 CFU/ml pada sampel pangan. Asumsi tersebut diperkuat dengan hasil hitungan cawan yang diperoleh dari beberapa tingkat pengenceran SPS, yaitu sebesar 4.5 x 106 CFU/g seperti yang terlampir pada Lampiran 5. Konsentrasi tersebut merupakan konsentrasi patogen B. cereus yang dapat menyebabkan keracunan pangan. Pada konsentrasi tersebut metode deteksi sudah harus mampu mendeteksi keberadaan patogen tersebut. Pengenceran serial 1:10 dari kultur murni B. cereus dan sampel nasi goreng yang diinokulasi secara artifisial bertujuan untuk menetapkan limit deteksi dari B. cereus yang diamplifikasi dengan real-time PCR. Secara teoritis, amplifikasi PCR memungkinkan deteksi hanya dengan 1 CFU/ml atau 1 CFU/g sampel pangan. Oleh karena itu, SPS diencerkan hingga enam tingkat pengenceran, dengan asumsi pada tingkat pengenceran keenam diperoleh konsentrasi bakteri 101-100 CFU/g. Tujuan inokulasi ±106 B. cereus CFU/g secara artifisial pada sampel nasi goreng adalah untuk menjamin spesifisitas amplifikasi, di mana produk amplifikasi yang diharapkan terbentuk murni dari penempelan primer spesifik dan DNA target yang komplementer. Tujuan inokulasi tersebut juga dapat menggantikan waktu inkubasi khusus pada persiapan sampel sebab umumnya nasi goreng yang diinokulasi bakteri secara artifisial diberi tambahan waktu inkubasi untuk memperkaya jumlah sel bakteri patogen sehingga peluang DNA terdeteksi semakin besar. Pada penelitian PROM terdahulu (2008), sampel pangan diinkubasi pada suhu 30oC selama 4 jam untuk memperbanyak jumlah bakteri uji tersebut. Namun, pada penelitian ini nasi goreng tidak diinkubasi terlebih dahulu sesudah inokulasi kultur murni B. cereus ke dalam nasi goreng. Hal ini dilakukan karena adanya asumsi pemeriksaan risiko kontaminasi bakteri patogen merupakan tindakan segera sesaat setelah pangan selesai diolah, sehingga waktu deteksi pun lebih cepat. Inokulasi nasi goreng secara 47 artifisial dengan kultur murni yang telah diinkubasi semalam dalam TSB telah cukup untuk menjamin diperolehnya DNA target bagi proses amplifikasi. Menurut Sambrook et al. (1989), pertumbuhan bakteri mencapai fase log akhir setelah diinkubasi semalam. Fase log akhir merupakan saat yang optimal untuk memanen bakteri, di mana kuantitas DNA maksimal. 2. Tahap Isolasi DNA a. Diferensiasi metode isolasi DNA 1) Metode pendidihan Metode pendidihan menggunakan enzim untuk melisis sel bakteri dan mengisolasi DNA, juga buffer TE 1X untuk menjaga tekanan osmotik DNA. Enzim yang digunakan adalah lisozim dan proteinase K. Lisozim ditambahkan karena enzim ini mampu mencerna membran luar sel bakteri. Aktivitas enzim lisozim efektif pada suhu ruang (25°C). Proteinase K merupakan enzim proteolitik yang digunakan untuk menghilangkan protein. Protein dapat menghambat proses amplifikasi DNA. Suhu optimum aktivitas enzim proteinase K ialah 55-60°C selama 1-2 jam. Semakin lama inkubasi setelah penambahan proteinase K, daya recovery DNA semakin baik (Sambrook et al., 1989). Oleh karena itu, waktu inkubasi yang digunakan adalah selama 2 jam pada suhu 55°C. Pendidihan cukup dilakukan sekali karena denaturasi berulang dikhawatirkan malah merusak struktur DNA atau pun DNA menjadi sulit renaturasi kembali saat didinginkan. Menurut Sambrook et al. (1989) semakin panjang waktu pendidihan, denaturasi DNA akan menjadi irreversibel. 2) Metode ekstraksi dengan fenol:kloroform Metode ekstraksi dengan fenol:kloroform mampu menghasilkan isolat DNA yang lebih murni karena sel bakteri dilisis oleh enzim dan deterjen, kemudian DNA diekstraksi 48 berulang. Enzim yang digunakan pada metode ini sama dengan pada metode pendidihan, yaitu lisozim dan proteinase K. Perbedaannya adalah pada metode ini digunakan deterjen SDS untuk membantu pelisisan sel. SDS merupakan salah satu deterjen yang berfungsi melisis membran sel dengan membuat lubang kecil sehingga DNA plasmid lepas, tetapi tetap mempertahankan keutuhan DNA kromosomal berbobot molekul tinggi. Deterjen tersebut mengemulsi lipid juga protein pada sel dan mengganggu interaksi polar yang mempersatukan membran sel, kemudian membentuk kompleks dengan lipid dan protein. Hal ini menyebabkan lipid dan protein tersebut berpresipitasi dalam larutan. SDS ditambahkan bersamaan dengan proteinase K, lalu diinkubasi. Penambahan enzim proteolitik (dalam penelitian ini proteinase K) dilakukan sebelum DNA diekstraksi dengan pelarut fenol:kloroform. Basis sebagian besar dari purifikasi asam nukleat adalah menghilangkan protein yang sering terbawa ketika mengekstrak larutan aqueous asam nukleat (Sambrook et al., 1989). Penggunaan beberapa senyawa pelisis diperlukan karena B. cereus merupakan bakteri Gram positif yang memiliki peptidoglikan tebal. Apabila pelisisan tidak sempurna, DNA tidak akan terekstraksi dan proses amplifikasi tidak dapat terjadi. 3) Metode dengan kit komersial Metode isolasi DNA yang telah diterapkan di Pusat Riset Obat dan Makanan, Badan POM RI (2008) menggunakan kit komersial. Kit komersial menggunakan PBS pada tahap awal isolasi DNA. PBS digunakan untuk melarutkan matriks pangan, menjaga keseimbangan kondisi fisiologis, dan mengendapkan inhibitor-inhibitor yang tidak diperlukan seperti kalsium (Espy et al., 2006). Buffer lisozim yang ditambahkan terdiri atas NaCl, Tris-HCl pH 8.0, EDTA, Triton X-100. Penambahan EDTA dan Triton X-100 berfungsi untuk membantu pelisisan sel terjadi, 49 sedangkan NaCl berperan sebagai sumber ion Na+ untuk menghambat muatan negatif menempel pada fosfat di DNA yang menyebabkan molekul DNA saling tolak menolak dan Tris-HCl berfungsi untuk mempertahankan pH, pH 8.0 diperlukan oleh lisozim agar dapat bekerja dengan baik. Enzim RNAse A juga digunakan untuk menghilangkan RNA, karena enzim ini mampu mencerna RNA dan bekerja sangat cepat di suhu ruang. Kolom mini dan larutan yang telah disediakan oleh menufaktur digunakan, setelah pelisisan matriks pangan dan membran luar sel bakteri serta penghilangan protein dan RNA yang dapat menjadi inhibitor proses amplifikasi. Kolom mini merupakan kolom silika yang berguna untuk menahan DNA, namun permeabel bagi molekul-molekul lainnya. Larutan dari kit komersial, terdiri atas buffer lisis, buffer pencuci, dan buffer elusi. Buffer lisis digunakan untuk melisis sel bakteri, buffer pencuci digunakan untuk mencuci benang DNA, dan buffer elusi digunakan untuk mengelusi DNA dan membawa DNA melewati membran pada kolom mini. Buffer lisis umumnya adalah larutan garam dan deterjen (Dollard, 1994). Proses amplifikasi B. cereus yang diinokulasi pada sampel pangan hanya menggunakan metode pendidihan dan metode dengan kit komersial dengan alasan yang akan dijelaskan pada pembahasan penetapan limit deteksi. b. Perbandingan metode isolasi DNA a. Preparasi sampel Metode pendidihan, metode ekstraksi dengan fenol:kloroform, dan metode ekstraksi dengan kit komersial berasal dari sampel yang sama sehingga tidak ada faktor yang dapat pembeda hasil isolasi hingga tahap ini. 50 Sentrifugasi pada awal tahap isolasi DNA berfungsi untuk mendapatkan pelet dari sampel pangan yang mengandung bakteri, sedangkan lemak/minyak pangan berada pada supernatan dan dibuang. b. Pelisisisan sel Pelisisan sel pada metode pendidihan hanya menggunakan SDS dan lisozim, sedangkan pada metode pendidihan juga ditambahkan SDS dan waktu inkubasi 55C lebih lama 1 jam dibandingkan metode pendidihan. Hal ini berpengaruh terhadap kemurnian isolat DNA yang dihasilkan. Isolat DNA yang dilisis dengan lisozim, proteinase K, dan SDS lebih mempermudah DNA terekstraksi karena sel dilisiskan lebih sempurna dan protein sebagai inhibitor PCR dicerna lebih baik (waktu inkubasi lebih panjang) oleh proteinase K. Kit komersial juga menggunakan lisozim, proteinase K dan SDS dalam pelisisan sel. Namun waktu inkubasi untuk enzim bekerja sangat singkat sehingga pelisisan atau pencernaan oleh enzim maupun SDS tidak optimal. c. Proteksi dan stabilisasi DNA Proteksi dan stabilisasi DNA dilakukan oleh buffer dan kation tertentu untuk mempertahankan pH dan stabilitas DNA. Metode pendidihan tidak menambahkan NaCl dalam buffernya karena menginginkan DNA tetap larut dalam air. Ion Na+ berfungsi menetralisir muatan negatif fosfat di DNA dengan cara membentuk ikatan ionik dengan muatan negatif tersebut, dan memperkenankan molekul-molekul DNA bersatu (Dollard, 1994). Jika muatan negatif tersebut tidak terikat dengan ion Na+, DNA akan saling tolak menolak. Metode ekstraksi dengan fenol:kloroform dan kit komersial menambahkan NaCl pada buffer enzim sehingga stabilitas DNA lebih terjaga dan mempermudah presipitasi DNA. 51 d. Pemisahan DNA dari debris sel dan protein Metode pendidihan memisahkan DNA dengan cara sentrifugasi, DNA kromosomal akan larut pada supernatan, sedangkan debris sel mengendap akibat pendidihan yang disusul segera dengan pendinginan. Pendidihan menyebabkan DNA terdenaturasi dan menginaktivasi enzim nuklease atau DNase yang mencerna asam nukleat dan menyebabkan penurunan kualitas maupun kuantitas DNA selama penyimpanan. Pendinginan segera setelah pendidihan menyebabkan DNA kromosomal terenaturasi kembali dan larut air, sedangkan DNA plasmid sulit didenaturasi karena secara topologi intertwined (Sambrook et al., 1989) sehingga mengendap bersama debris sel serta protein yang terdenaturasi. Metode ekstraksi dengan fenol:kloroform memisahkan DNA dari lipid, polisakarida, dan protein melalui 3 fase yang dibentuk, yaitu fase aqueous dimana DNA larut, interfase dimana protein larut, dan fase organik dimana lipid atau polisakarida larut. DNA termasuk molekul polar sehingga larut pada air (fase aqueous) dan fase ini yang diambil saat ekstraksi DNA dengan pelarut fenol:kloroform. Pemisahan komponen-komponen tersebut ke dalam tiga fase memberikan isolat DNA yang lebih murni. Ekstraksi fenol:kloroform dilakukan dua kali hingga lapisan keruh interfase tidak tebal. Lapisan keruh interfase yang tebal dapat terlihat pada Gambar 9a, sedangkan hasil yang diharapkan adalah lapisan interfase yang sangat tipis (Gambar 9b). Lapisan keruh menandakan protein masih banyak terdapat pada interfase. Protein tersebut dapat menjadi inhibitor PCR jika menempel pada isolat DNA. Pada penelitian ini ekstraksi dengan fenol:kloroform dilanjutkan dengan penambahan kloroform untuk menghilangkan sisa fenol dan protein yang terbawa pada fase aqueous. 52 atas tengah bawah (a) (b) Gambar 9. Fase aqueous (atas), Interfase (tengah), dan Fase organik (bawah) dalam isolasi fenol:kloroform, (4a) Lapisan interfase tebal dan (4b) Lapisan interfase sangat tipis Fenol yang digunakan harus memiliki pH lebih dari 7.6 agar DNA larut pada fase aqueous (bagian atas). Tris-HCl pH 8.0 digunakan untuk meningkatkan pH fenol. Setelah pH tercapai, fenol seharusnya ditambah dengan hydroxyquinolin sebanyak 0.05% total volume fenol sebagai antioksidan dan 0.1M Tris-Cl pH 8.0 sebanyak 0.1 total volume fenol sebagai buffer bagi fenol. Namun karena keterbatasan senyawa kimia hidroxyquinolin, senyawa ini tidak ditambahkan ke dalam fenol. Untuk mencegah oksidasi, fenol disimpan pada botol gelap di suhu -20°C. Fenol tetap bekerja sebagaimana mestinya pada saat ekstraksi fenol:kloroform tanpa hydroxyquinolin, tetapi tentu saja mengurangi efektivitas ekstraksi karena hydroxyquinoline (0.1 %) selain merupakan antioksidan, juga inhibitor RNAase dan kelator ion logam yang lemah. Dalam ekstraksi dengan fenol:kloroform, senyawa ini akan mempermudah pembedaan fase organik dan fase aqueous karena memberi warna kuning pada fase organik. Campuran fenol:kloroform akan membentuk fase cair-cair. Bagian atas disebut fase aqueous, sedangkan bagian bawah disebut fase organik. DNA akan larut pada fase aqueous, asalkan pH fenol:kloroform lebih dari 7.6 (Sambrook et al., 1989). Oleh 53 sebab itu, saat ekstraksi DNA dilakukan bagian yang diambil ialah fase aqueous. Fase aqueous yang diambil hanya sekitar 90% karena dikhawatirkan protein pada interfase ikut terambil saat pemipetan. Ekstraksi fenol:kloroform dilakukan dua kali karena protein pada interfase sudah tidak tampak pada ekstraksi kedua, setelah itu dilanjutkan dengan pemberian kloroform untuk menghilangkan sisa fenol. Kloroform juga berperan dalam mendenaturasi protein dan membantu pemisahan fase aqueous dan fase organik (Birren et al., 1997). Pemberian fenol:kloroform memerlukan inkubasi pada suhu -20°C selama 30 menit. Suhu tersebut merupakan suhu yang dapat menjaga kestabilan DNA, mempresipitasi DNA, dan menginaktivasi enzim yang mungkin ikut terekstraksi. Pemberian kloroform tidak memerlukan inkubasi sebab sekedar mencuci fase aqueous dan menghilangkan fenol. Pencucian dengan kloroform dilanjutkan dengan sentrifugasi 12000 rpm selama 10 menit pada suhu 4°C untuk menjaga DNA tidak terdegradasi. Setelah sentrifus akan terbentuk 2 fase cair-cair, yang diambil adalah fase aqueous di mana DNA larut, sedangkan fase organik berisi kloroform dan komponen terlarut (lipid) dan interfase berisi fenol dan komponen terlarut (protein) dibuang. Metode dengan kit komersial memisahkan DNA dari debris sel dengan cara filtrasi oleh membran silika pada kolom yang telah disediakan kit tersebut. Sebagian besar debris berukuran lebih besar daripada DNA. Kemurnian DNA seharusnya lebih baik dibandingkan isolat DNA dari metode pendidihan dan ekstraksi fenol:kloroform karena membran tersebut hanya mengikat DNA berdasarkan kekuatan ionik yang dimiliki DNA, namun permeabel terhadap komponen lain selain DNA. Ikatan ionik tersebut dapat terbentuk jika pH buffer 8.0 dan garam dengan konsentrasi yang tepat, misal 5 mM KPO4 dilarutkan 54 dalam etanol 80%. Jika kondisi tersebut tidak terpenuhi, DNA tidak akan teradsorpsi pada membran atau kolom silika. e. Presipitasi DNA Penambahan garam dan alkohol yang tepat berperan penting dalam presipitasi DNA setelah ekstraksi fenol:kloroform. Garam yang ditambahkan adalah 10 M Ammonium Asetat pH 7.4 sebanyak 0.3 volume hasil ekstraksi DNA. Selain garam 10 M Ammonium Asetat pH 7.4 dapat juga digunakan garam 3 M Natrium Asetat pH 4.8. Kedua garam tersebut memiliki fungsi dan peran yang sama dalam tahap isolasi DNA. Pemberian garam kation monovalen (Na+ atau NH3+) ini berfungsi untuk menurunkan pH sehingga DNA mengalami renaturasi. Garam 3 M Natrium Asetat pH 4.8 disarankan untuk mempresipitasi sebab DNA mudah di-recovery dari larutan cair yang berkation monovalen (Birren et al., 1997). Penambahan alkohol dimaksudkan untuk me-recovery DNA sebab DNA tidak larut dalam alkohol. Alkohol kurang polar dibandingkan dengan air, sehingga ikatan ionik antara Na+ dan fosfat semakin kuat karena berkurangnya hidrasi oleh air. Akibatnya DNA berpresipitasi. Alkohol dingin diperlukan saat hendak mengisolasi DNA yang kurang dari 100 pasang basa atau DNA yang diperoleh dari hasil pengenceran berkali-kali (Birren et al., 1997). Presipitasi DNA oleh alkohol dapat dilakukan dengan etanol absolut atau isopropanol. Kedua alkohol tersebut memiliki sifat yang berbeda, yaitu: (1) Etanol lebih volatil dibandingkan dengan dikeringudarakan tanpa isopropanol vakum, (2) sehingga pelet dapat Etanol lebih efektif dibandingkan isopropanol dalam melarutkan garam sehingga garam tidak tertinggal pada pelet DNA, (3) Etanol membutuhkan 2.5 kali volume total suspensi DNA untuk dapat mempresipitasi DNA, sedangkan isopropanol cukup dengan 1 kali volume total, dan (4) Akibat kepolarannya, etanol lebih lama mengendapkan 55 DNA dibandingkan dengan isopropanol. Beberapa protokol menyarankan etanol atau isopropanol diberikan dalam kondisi dingin (4°C) untuk mempertahankan integritas DNA. Menurut Sambrook et al. (1989), suhu yang dingin (4°C) atau suhu ruang (25°C) alkohol tidak berpengaruh signifikan terhadap hasil presipitasi DNA. Sesudah pemberian garam berkation monovalen dan etanol/isopropanol, fase aqueous tersebut diinkubasi pada suhu -20°C semalam sehingga muncul flokulan putih. Flokulan tersebut terdiri dari DNA kromosomal dan ammonium, jika ekstraksi berjalan sempurna. Apabila ekstraksi tidak murni, kemungkinan kontaminan yang terdapat pada flokulan putih tersebut adalah RNA berbobot molekul tinggi, SDS, protein, dan kompleks membran. Flokulan berupa benang putih DNA akan mudah teramati jika konsentrasi DNA yang terekstrak tinggi. Tahap sentrifugasi perlu dilakukan setelah inkubasi (-20°C) semalam untuk melekatkan flokulan pada dasar tabung eppendorf. Bagian yang dibuang setelah sentrifugasi ini adalah supernatan dan pelet dijaga tetap berada pada dasar tabung eppendorf. DNA kromosomal akan membentuk kompleks tak larut dan lebih sulit terenaturasi dibandingkan dengan DNA plasmid karena target isolat DNA ialah DNA kromosomal, yang diambil ialah bagian peletnya, bukan supernatan yang mengandung DNA plasmid. Metode pendidihan memerlukan DNA larut pada supernatan sehingga tidak ada tahap presipitasi, sedangkan metode ekstraksi dengan kit komersial mempresipitasi DNA ketika tahap adsorpsi DNA dilakukan, yaitu dengan etanol 80%. Etanol juga merupakan senyawa kurang polar dibandingkan dengan air. Prinsip presipitasi etanol sama dengan isopropanol. 56 f. Pemekatan DNA Pelet DNA sangat sulit diperoleh dan recovery sulit terjadi, yang berarti konsentrasi DNA rendah. Menurut Birren et al. (1997), untuk DNA dengan konsentrasi rendah, peningkatan daya recovery dapat dilakukan dengan memperpanjang waktu sentrifugasi, bukan dengan memperlama waktu inkubasi. Oleh karena itu, sentrifugasi yang biasanya dilakukan 10 menit diperpanjang menjadi 30 menit. Pelet hasil sentrifugasi diberi etanol 70% untuk mencuci benang DNA. Etanol 70-80 % akan melarutkan garam NH3+ yang kemungkinan masih terdapat pada isolat DNA, tanpa melarutkan asam nukleat. Setelah pencucian dengan etanol 70%, pelet dikeringkan. Pengeringan tersebut dapat dilakukan dengan alat vakum jika kuantitas pelet besar. Namun, kuantitas pelet yang diperoleh dari isolasi DNA ini sangat rendah (pelet tidak tampak jelas) sehingga pelet dikeringudarakan. Pelet yang sudah kering dilarutkan dalam buffer TE 1X untuk menjaga tekanan osmotik DNA. Metode isolasi yang banyak digunakan untuk mengisolasi DNA adalah metode ekstraksi dengan fenol:kloroform. Fenol mampu mendenaturasikan protein secara efisien dan menghasilkan isolat DNA yang cukup murni. 3. Tahap Amplifikasi Amplifikasi real-time PCR menggunakan campuran pereaksi dan isolat DNA dengan total volume 25 µl, yaitu volume minimal bagi thermal cycler yang digunakan dalam penelitian. Komposisi atau konsentrasi dye fluorescence, buffer PCR, Mg2+, deoksinukleotida, DNA polimerase, dan primer sangat berpengaruh terhadap efektivitas amplifikasi. Campuran buffer PCR, Mg2+, deoksinukleotida, dan DNA polimerase telah tersedia 57 secara komersial yakni IQ SYBR Green Supermix dan digunakan dalam proses amplifikasi ini. SYBR Green digunakan sebagai dye fluorescence sebab lebih mudah untuk mendeteksi produk PCR (amplikon) dan ekonomis, dibandingkan penggunaan probe, dan juga lebih sensitif dibandingkan pewarnaan dengan ethidium bromida pada elektroforesis gel agarosa. SYBR Green bekerja dengan cara mengikat DNA untai ganda dan mengeksitasikan sinar. Sinar berupa fluoresen akan meningkat sebanding dengan akumulasi produk PCR. Sayangnya, SYBR Green mengikat juga DNA untai ganda lain (bukan produk spesifik) dalam reaksi, misalnya primer dimer. Komposisi campuran reaksi mengandung RNAse free water yang berfungsi untuk melarutkan senyawa lainnya dalam larutan untuk reaksi PCR. SYBR Green berperan sebagai pewarna fluoresen, DNA polimerase berperan sebagai enzim yang memperpanjang primer secara komplementer terhadap DNA target, dan deoksinukleotida berfungsi untuk menyediakan basa nukleotida bagi proses polimerisasi oleh DNA polimerase. Primer berfungsi untuk mengawali polimerisasi oleh DNA polymerase dan menjadi penentu spesifisitas gen yang dianalisis. Isolat DNA atau DNA templat berfungsi sebagai DNA target yang akan diamplifikasi. Campuran reaksi beserta DNA target dipipet pada microwell dan ditutup oleh microseal adhesive agar cairan tidak menguap keluar dari well saat amplifikasi berjalan. Apabila cairan dibiarkan menguap akan mengurangi DNA target yang teramplifikasi, dengan kata lain mengurangi konsentrasi bakteri yang terdeteksi. Amplikasi dilaksanakan pada suhu 95°C selama 10 menit sebagai tahap predenaturasi untuk menjamin bahwa semua DNA untai tunggal telah menjadi DNA untai tunggal; (40 siklus) 95°C selama 10 detik tahap denaturasi dan 55°C selama 45 detik tahap penempelan (annealing). Suhu tersebut merupakan suhu yang dikembangkan Fricker et al. (2007) untuk mendeteksi B. cereus pada sampel pangan pasta dan nasi. Waktu annealing diturunkan dari 60 detik menjadi 45 detik berdasarkan trial and error. Dalam waktu 45 detik DNA target sudah teramplifikasi dan 58 terdeteksi. Tahap perpanjangan (extension) tidak perlu lagi dilakukan dengan real-time PCR karena yang diperlukan ialah data awal terjadinya amplifikasi di setiap siklus (fase eksponensial), bukan akumulasi produk di fase akhir (fase plato) seperti halnya PCR standar. Hal ini mempersingkat waktu amplifikasi. Proses amplifikasi (40 siklus) dilanjutkan dengan 80 siklus untuk memperoleh melt curve dan mengetahui melting temperature (Tm) produk PCR. Amplifikasi dari bakteri yang sama dengan primer yang sama akan memiliki melting temperature (suhu saat DNA untai ganda menjadi DNA untai tunggal) yang sama (Tm). Nilai Tm dapat digunakan untuk menetapkan spesifisitas uji dan hanya dapat dibaca pada melt curve. Melt curve ialah grafik perbandingan suhu dan turunan RFU terhadap suhu sehingga dapat diketahui spesifisitas bakteri. Untuk memperoleh melt curve, amplifikasi dilanjutkan dengan menambahkan 80 siklus dengan suhu 55°C (sesuai suhu annealing) selama 10 detik. 4. Evaluasi Kinerja Real-Time PCR Pemaparan mengenai limit deteksi untuk menilai sensitivitas uji PCR dan spesifisitas uji akan dijelaskan di bawah ini. a. Limit deteksi Grafik amplifikasi dan kurva standar hasil amplifikasi dari isolat DNA yang diisolasi dengan metode pendidihan, metode ekstraksi dengan fenol:kloroform, dan metode ekstraksi dengan kit komersial disajikan pada Gambar 10. Grafik amplifikasi berbentuk sigmoid terdiri atas beragam warna yang masing-masing menandakan pengenceran isolat DNA yang teramplifikasi. Grafik 10a, menggambarkan 8 tingkat pengenceran teramplifikasi, Grafik 10b menggambarkan 7 tingkat pengenceran teramplifikasi, Grafik 10c hanya 3 tingkat pengenceran teramplifikasi. Kurva standar tersebut menunjukkan bahwa konsentrasi bakteri berbanding terbalik dengan nilai Ct. Semakin tinggi konsentrasi bakteri maka nilai Ct akan semakin rendah karena jumlah DNA target lebih banyak sehingga 59 primer lebih cepat menempel pada DNA target. Oleh karena itu, nilai Ct pada kultur murni B. cereus sebelum pengenceran paling rendah Threshold cycle (Ct) PCR Base Line Subtracted RFU dan semakin tinggi pengenceran, semakin tinggi pula nilai Ct. Log konsentrasi bakteri (CFU/ml) Cycle (d) Threshold cycle (Ct) PCR Base Line Subtracted RFU (a) Log konsentrasi bakteri (CFU/ml) Cycle (e) Threshold cycle (Ct) PCR Base Line Subtracted RFU (b) Log konsentrasi bakteri (CFU/ml) Cycle (c) (f) Gambar 10. (a-c) Grafik amplifikasi DNA B. cereus, (a) Hasil isolasi metode pendidihan; (b) Hasil isolasi metode fenol:kloroform; (c) Hasil isolasi dengan kit komersial. (d-f) Kurva standar kultur murni B. cereus, (d) Metode pendidihan; (e) Metode ekstraksi dengan fenol:kloroform; (f) Metode dengan kit komersial 60 Kurva standar kultur murni B. cereus yang DNA-nya diisolasi dengan kit komersial hanya menunjukkan tiga tingkat pengenceran yang DNA-nya teramplifikasi. Hal ini disebabkan tahap persiapan isolat DNA tidak sebaik isolasi DNA dengan metode pendidihan dan ekstraksi dengan fenol:kloroform. Hal tersebut dapat disebabkan oleh sedikitnya waktu saat pelisisan sel dengan enzim dan deterjen. Masing-masing kurva standar di atas memiliki nilai R2, slope, y-int serta efisiensi kurva standar yang ditabulasikan pada Tabel 4. Nilai R2 dijadikan tolak ukur kurva standar yang paling baik dalam penelitian ini. Nilai optimum R2 dan E suatu kurva standar, berturutturut, adalah 1.000 dan 90-110%. Kurva standar yang memiliki nilai optimum diasumsikan sebagai kurva standar yang terbaik (Alarcon et al., 2006). Kurva standar yang memberi nilai R2 mendekati nilai optimum adalah kurva standar dari metode pendidihan dengan nilai R2 sebesar 0.944. Melalui persamaan linear yang diperoleh dari kurva standar juga dapat ditentukan efisiensi amplifikasi. Nilai efisiensi merupakan perbandingan antara reagen dan produk yang terbentuk dan menunjukkan persentase efisiensi PCR dalam proses amplifikasi. Tabel 4. Data kurva standar dari metode isolasi pendidihan, ekstraksi dengan fenol:kloroform, dan ekstraksi dengan kit komersial DNA Metode Metode Metode Nilai ekstraksi dengan ekstraksi dengan pendidihan fenol:kloroform kit komersial R2 0.944 0.817 0.894 Slope -0.321 -0.087 -0.639 y-int 31.208 29.805 37.172 1.3 x 105 2.8 x 1013 3.6 x 103 Efisiensi (%) Seluruh nilai efisiensi amplifikasi ini melebihi kisaran optimal efisiensi amplifikasi. Hal ini disebabkan oleh produk non spesifik yang terbentuk. Produk non spesifik disebabkan karena amplifikasi 61 yang terjadi bukan antara primer BceT dan DNA target, misalnya amplifikasi antara primer dan primer, amplifikasi antara primer dan RNA sehingga terjadilah over efficiency di mana reagen dianggap sangat efisien menghasilkan banyak produk. Karena seluruh nilai efisiensi terlampau tinggi, kurva standar yang terbaik untuk penetapan limit deteksi hanya ditentukan berdasarkan nilai R2 yang mendekati optimum. Mengacu pada data kurva standar (Tabel 4) yang diperoleh, isolasi DNA dengan metode pendidihan dijadikan sebagai kurva standar dalam penelitian ini. Oleh karena itu, untuk penetapan limit deteksi B. cereus dalam sampel pangan selanjutnya akan menggunakan persamaan linear dari kurva standar metode pendidihan. Sensitivitas uji merupakan limit deteksi terendah yang mampu dicapai oleh pendeteksian real-time PCR. Data penentuan limit deteksi kultur murni B. cereus dan B. cereus pada sampel pangan nasi goreng dapat dilihat pada Tabel 5. Tabel 5. Limit deteksi kultur murni B. cereus dan B. cereus dalam nasi goreng Sampel KM SPS SP Pengenceran Sampel 1.0 x 10-1 1.0 x 10-2 1.0 x 10-3 1.0 x 10-4 1.0 x 10-5 1.0 x 10-6 1.0 x 10-7 1.0 x 10-8 1.0 x 10-6 1.0 x 10-1 Keterangan: Threshold Cycle (Ct) Metode Metode Pendidihan Fenol: Kloroform 29.45 28.99 29.92 29.31 30.36 29.36 31.05 29.40 31.58 29.42 31.16 29.44 31.94 29.68 31.79 ‐ 31.68 31.78 31.78 31.56 CFU/ml Metode Metode Fenol: pendidihan Kloroform 7 8.8 x 10 8.8 x 106 8.8 x 105 8.8 x 104 8.8 x 103 8.8 x 102 8.8 x 101 8.8 1.6 1.3 8.8 x 107 8.8 x 106 8.8 x 105 8.8 x 104 8.8 x 103 8.8 x 102 8.8 x 101 8.8 1.3 2.0 KM = Kultur murni B. cereus SPS = Nasi goreng yang telah diinokulasi secara artifisial dengan kultur murni (10-1) SP = Nasi goreng yang tidak diinokulasi secara artifisial Kultur murni B. cereus masih terdeteksi hingga pengenceran tertinggi (10-8) dengan metode pendidihan sehingga limit deteksinya adalah 8.8 CFU/ml, sedangkan DNA B. cereus yang diisolasi dengan metode 62 ekstraksi fenol:kloroform limit deteksi adalah 88 CFU/ml. Metode ekstraksi dengan fenol:kloroform tidak dapat mengisolasi DNA pada pengenceran tertinggi (10-8) sehingga tidak terjadi amplifikasi. Isolat DNA pada pengenceran 10-8 tidak terisolasi dengan baik akibat homogenisasi yang kurang pada tahap pengenceran juga tahap isolasi DNA. Metode pendidihan dan metode ekstraksi dengan fenol:kloroform mampu mengamplifikasi DNA B. cereus dari SPS hingga pengenceran tertinggi (10-6). Limit deteksi B. cereus pada sampel pangan yang diinokulasi secara artifisial (SPS) dari metode pendidihan adalah 1.6 CFU/ml, sedangkan dari metode ekstraksi dengan fenol:kloroform adalah 1.3 CFU/ml. Sampel pangan yang tidak diinokulasi turut diisolasi dan isolat DNA diamplifikasi untuk menguji kesensitifan metode ini. Metode deteksi berbasiskan DNA berupa amplifikasi dengan real-time PCR mampu mendeteksi B. cereus pada sampel pangan tanpa inokulasi kultur murni B. cereus. Limit deteksi B. cereus pada sampel nasi goreng (SP) dengan metode pendidihan ialah 1.3 CFU/ml, sedangkan ekstraksi dengan metode fenol:kloroform adalah 2.0 CFU/ml. Angka limit deteksi metode pendidihan lebih rendah daripada metode fenol:kloroform karena senyawa inhibitor pada metode pendidihan lebih banyak. Isolasi DNA dengan metode pendidihan hanya menggunakan lisozim dan proteinase K untuk menghancurkan dinding sel dan menghilangkan protein, sedangkan isolasi DNA dengan ekstraksi fenol:kloroform selain menggunakan lisozim, SDS dan proteinase K, juga masih dilanjutkan dengan ekstraksi fenol:kloroform sehingga inhibitor PCR seperti lipid, protein, polisakarida terpisah dari DNA yang hendak diisolasi. Hal tersebut menyebabkan DNA yang diisolasi oleh ekstraksi fenol:kloroform konsentrasinya lebih tinggi dan teramplikasi lebih cepat, Ct amplikon hasil isolasi dengan ekstraksi fenol:kloroform lebih rendah dibandingkan dengan metode pendidihan. Real-time PCR masih memerlukan optimasi pada beberapa komponen reaksi dan suhu annealing untuk memperoleh hasil optimum. Komponen reaksi yang masih perlu dioptimasi, antara lain konsentrasi magnesium dan konsentrasi primer. 63 Magnesium memungkinkan enzim polimerase berfungsi secara optimal sebab menstimulasi aktivitas enzim tersebut. Dalam penelitian ini, magnesium tidak ditambahkan pada campuran komponen reaksi PCR, dengan asumsi Mg2+ yang terdapat pada SYBR telah optimum untuk proses amplifikasi. Konsentrasi magnesium yang terlalu tinggi akan menghasilkan akumulasi produk non spesifik, tetapi konsentrasi terlalu rendah tidak akan memberikan hasil amplikon. Menurut Anderson (2006), Mg2+ penting bahkan perlu ditambahkan ketika DNA target disimpan dalan buffer TE, karena EDTA akan mengkelat Mg2+ sehingga reaksi akan kekurangan Mg2+. Konsentrasi primer juga perlu dioptimasi karena konsentrasi yang terlalu tinggi akan meningkatkan kemungkinan terbentuknya produk non spesifik, sedangkan konsentrasi terlalu rendah proses amplifikasi tidak akan berkangsung. Hal lain yang masih memerlukan optimasi ialah suhu annealing. Suhu annealing yang terlalu tinggi menyebabkan primer tidak akan menempel, sedangkan suhu annealing yang terlalu rendah menyebabkan primer akan menempel di mana saja dan terbentuklah produk non spesifik. Hal ini yang diduga menyebabkan nilai efisiensi sangat besar pada amplifikasi DNA B. cereus di atas. b. Spesifisitas uji Spesifisitas uji dipengaruhi oleh pasangan primer yang digunakan, mencakup susunan nukleotida, panjang nukleotida, melting temperature (Tm) primer, annealling tempereture, dan konsentrasi primer. Primer yang spesifik terhadap B. cereus dianalisis dengan program BLAST. Primer spesifik yang digunakan adalah BceT1_F 5’-GAA TTC CTA AAC TTG CAC CAT CTC G-3’ untuk forward primer dan BceT2_R 5’-CTG CGT AAT CGT GAA TGT AGT CAA T-3’ untuk reverse primer. Primer tersebut komplementer terhadap DNA target gen penyandi enterotoksin B. cereus. Panjang basa nukleotida keduanya ialah 25 basa nukleotida. 64 Semakin panjang oligonukleotida, semakin spesifik primer tersebut. Ukuran amplikon yang dihasilkan adalah 646 pasang basa. Forward primer memiliki Tm 62.9 dan reverse primer memiliki Tm 61.3 sehingga selisih suhu peleburan keduanya adalah 1.6°C. Selisih Tm sepasang primer sebaiknya tidak lebih dari 3°C. Selisih Tm pasangan primer semakin kecil, maka pasangan primer tersebut semakin baik digunakan dalam amplifikasi sebab Tm akan berpengaruh pada suhu penempelan antara primer dan DNA target. Suhu penempelan (annealing) biasanya 5°C di bawah Tm primer. Penempelan akan berjalan serempak apabila Tm keduanya sama. Penentuan suhu Tm amplikon berguna untuk mengukur spesifisitas uji amplifikasi dari isolat DNA B. cereus. Nilai Tm amplikon ditentukan berdasarkan melt curve yang diperoleh dari metode pendidihan dan metode ekstraksi dengan fenol:kloroform, dan metode dengan kit komersial tersaji pada Gambar 11. Berdasarkan melt curve, puncak kurva amplikon yang terbentuk dari amplifikasi B. cereus dan primer BceT berada pada suhu 81.5°C sehingga suhu tersebut merupakan Tm amplikon. Puncak melt curve yang dibentuk dari DNA hasil isolasi dengan metode pendidihan tidak terpusat pada Tm yang sama karena spesifisitas amplikon terganggu oleh adanya inhibitor PCR akibat isolat DNA kurang murni. Metode dengan kit komersial juga menghasilkan puncak kurva pada suhu yang berbeda-beda yang berarti terdapat produk non spesifik pada hasil amplifikasi. Spesifisitas terbaik dicapai ketika hanya satu puncak yang terbentuk pada sebuah melt curve dan melt curve dari beberapa pengenceran suatu sampel memperlihatkan puncak pada suhu yang sama. Melt curve dari ketiga metode ada yang memiliki dua puncak juga karena adanya larutan di dinding microwell yang teramplifikasi dan amplikon dari amplifikasi tersebut yang terdeteksi pertama kali, kemudian ketika larutan tersebut habis karena menguap barulah larutan pada dasar microwell, yang seharusnya menjadi target deteksi, mengalami proses amplifikasi dan terbentuklah puncak kedua pada melt curve. Apabila 65 terdapat dua puncak, kemungkinan terbesar puncak yang menunjukkan Tm -d(RFU)/dT amplikon sesungguhnya adalah puncak kedua. Suhu (a)(°C) -d(RFU)/dT (a) Suhu (b)(°C) -d(RFU)/dT (b) Suhu (c) (°C) (c) Gambar 11. Melt curve dari DNA yang diisolasi dengan (a) metode pendidihan, (b) metode ekstraksi dengan fenol:kloroform, dan (c) metode ekstraksi dengan kit komersial 66 Kurva amplikon dari isolat DNA hasil isolasi dengan metode pendidihan dan ekstraksi fenol:kloroform semuanya memiliki puncak dengan suhu 81.5 °C. Berdasarkan melt curve tersebut, nilai Tm produk PCR (amplikon) ditetapkan sebesar 81.5°C. Spesifisitas amplikon yang diperoleh dari metode isolasi DNA dengan pelarut fenol:kloroform paling baik karena puncaknya lebih terpusat pada Tm 81.5°C. Setiap warna kurva pada melt curve menunjukkan isolat DNA berasal dari pengenceran yang berbeda, misalkan pada Gambar 11c terdapat tiga warna kurva yang membentuk puncak maka terdapat tiga pengenceran isolat berbeda yang menghasilkan produk PCR. Agar melt curve hanya memiliki satu puncak, isolat DNA harus murni, larutan yang akan diamplifikasi harus homogen, dan diusahakan larutan tersebut tidak menempel di sekitar dinding atas microwell, selain di dinding dasar microwell. Spesifistitas diuji pula dengan menggunakan kontrol negatif B. subtilis. Primer spesifik B. cereus ini (BceT) komplementer terhadap DNA gen penyandi enterotoksin B. cereus sehingga tidak dapat mengamplifikasi DNA selain DNA dari gen penyandi enterotoksin B. cereus. Jika primer spesifik hanya terhadap B. cereus maka amplifikasi tidak dapat terjadi pada kontrol negatif. Grafik amplifikasi dan melt curve dari B. subtilis yang diamplifikasi dengan primer BceT disajikan pada -d(RFU)/dT PCR Base Line Subtracted RFU Gambar 12. Cycle Suhu (°C) (a) (b) Gambar 12. (a) Grafik amplifikasi dan (b) melt curve isolat DNA B. subtilis 67 Pada penelitian ini, primer BceT ternyata mampu meningkatkan fluoresen yang berarti proses amplifikasi terjadi. Amplifikasi yang terjadi ditunjukkan dengan terbentuknya kurva sigmoidal pada grafik amplifikasi (Gambar 9a) dan puncak melt curve (Gambar 9b) menunjukkan Tm 82°C berbeda dengan Tm amplikon dari amplifikasi isolat DNA B. cereus. Nilai Tm amplikon yang hanya berselisih sedikit ini masih perlu dilakukan amplifikasi ulang untuk menjamin bahwa Tm B. subtilis tersebut berasal dari amplikon DNA B. subtilis, bukan dari produk non spesifik yang terbentuk saat amplifikasi karena kurang optimalnya konsentrasi primer atau suhu annealing. Konsentrasi primer yang terlalu tinggi akan menyebabkan primer menempel di sembarang sekuens DNA dan terbentuklah produk yang dalam melt curve ini memiliki nilai Tm bukan pada DNA target. Oleh karena itu, perlu dilakukan optimasi konsentrasi primer, baik reverse maupun forward primer. Apabila produk non spesifik terbentuk, suhu annealing perlu dinaikkan (1-2°C) secara bertahap. 68 VI. KESIMPULAN DAN SARAN A. KESIMPULAN Antigen yang dimiliki patogen dapat digunakan untuk mengklasifikasikan patogen tersebut ke dalam tingkatan yang lebih rendah dari spesies, penetapan serovar berdasarkan antigen ini banyak diterapkan pada penamaan Salmonella. Sepuluh isolat yang telah dikonfirmasi merupakan spesies yang sama yaitu Salmonella spp. ternyata terdiri dari 6 serovar yang berbeda berdasarkan antigen O yang dimilikinya, yaitu Salmonella spp. dengan antigen O:5, O:8, O:11 (OMF hanya memiliki faktor tunggal 0:11), O:12, O:1,4,12 dan O:6,7,14. Uji amplifikasi dengan real-time PCR merupakan salah satu metode deteksi patogen berbasiskan DNA yang dapat diterapkan pada sampel pangan yang telah diinokulasi. Isolasi DNA dapat langsung dilakukan tanpa penambahan waktu inkubasi setelah kultur murni B.cereus diinokulasi. Isolasi DNA menggunakan metode ekstraksi dengan fenol:kloroform direkomendasikan untuk pengujian yang memerlukan DNA murni, seperti halnya amplifikasi dengan PCR. Metode deteksi berbasiskan DNA dengan real-time PCR memiliki sensitivitas tinggi, limit deteksi terendah yang dicapai pada sampel pangan nasi goreng yang diinokulasi B. cereus adalah 1.3 CFU/ml. Sampel pangan nasi goreng yang tidak diinokulasi ternyata juga mengandung B. cereus dan patogen tersebut dapat dideteksi secara langsung oleh metode deteksi dengan real-time PCR. Sampel pangan yang mengandung 1.3 CFU/ml B. cereus dapat terdeteksi dengan real-time PCR. B. SARAN 1. Uji serologi sebaiknya dilakukan secara lengkap sehingga penetapan serovar dan penamaan subspesies dapat dilakukan. 2. Optimasi suhu annealing, konsentrasi primer, dan konsentrasi Mg2+ perlu dilakukan untuk memperoleh hasil optimal dalam proses amplifikasi PCR. 69 DAFTAR PUSTAKA Alarcon, B., B. Vicedo, and R. Aznar. 2006. PCR-based procedures for detection and quantification of Staphylococcus aureus and their application in food. Journal of Applied Microbiology 100 (2006), p.352-364. Anderson, J.M. 2006. Design and Applications in Molecular Biology Research: PCR and Primer Design. http://www.google.co.id/ppt [10 Maret 2009] Anonim. 2001. Rapid Methods for Detecting Foodborne Patogens. Bacteriological Analytical Manual Online, FDA. Ash, C., J. A. E. Farrow, M. Dorsch, E. Stackebrandt, and M. D. Collins. 1991. Comparative analysis of Bacillus anthracis, Bacillus cereus, and related species on the basis of reverse transcriptase sequencing of 16S rRNA. Int. J.Syst. Bacteriol. 41:343–346. Atlas, R.M. 2006. Handbook Of Microbiological Media for the Examination of Food. 2nd Ed. CRC Press, Boca Raton. [Badan POM] Badan Pengawas Obat dan Makanan. 2008. Kajian dan Penelusuran Mikroba Patogen Penyebab Keracunan Pada Pangan. Laporan Bidang Keamanan Pangan Pusat Riset Obat dan Makanan. Jakarta. Birren, B., E.D. Green, S. Klapholz, R.M. Myers, and J. roskams. 1997. Genome Analysis. A Laboratory Manual Vol.1/Analyzing DNA. Cold Spring Harbor Laboratory Press, USA. Boom, R., C.J.A. Sol, M.M.M. Salimans, C. L. Jansen, P. M. E. Wertheim-Van Dillen, and J. Noordaa. 1990. Rapid and simple method for purification of nucleic acids. J. Clin. Microbiol. 28:495-503. [CDC] Center for Disease Control and Prevention. 2008. Biosafety in Microbiological and Biomedical Laboratories (BMBL) - 4th edition, US Government Printing Office, Washington. http://www.cdc.gov/od/ohs/biosfty.htm [21 April 2009]. Chapaval, L., D.H. Moon, J.E. Gomes, F.R. Duarte, and S.M. Tsai. 2008. An alternative method for Staphylococcus aureus DNA isolation. Arq. Bras. Med. Vet. Zootec., v.60, n.2, p.299-306. Cohen, N.D., L.J. Martin, R.B. Simpson, D.E. Wallis, and H.L. Neibergs. 1996 Comparison of polymerase chain reaction and microbiological culture for detection of salmonellae in equine feces and environmental samples. American Journal of Veterinary Research 57, 780–786. 70 de Boer, E. and R.R. Beumer. 1999. Methodology for detection and typing of foodborne microorganisms. International Journal of Food Microbiology 50 (1999) 119–130. [Deptan] Departemen Pertanian. 2009. Situasi konsumsi. http://bkp.deptan.go.id/seputar bkp/web konsumsi [16 juli 2009]. Dharmaraj, S. 2009. RT-PCR: The Basics. http://www.ambion.com/techlib [27 Februari 2009]. Dollard, K. 1994. DNA isolation from union. http://www.accessexcellence.org/AE/AEC/AEF/1994/dollard_onionDNA.ph p [21 Juli 2009]. D’Aoust, J.Y. 2000. Salmonella. Di dalam: The Micobiological Safety and Quality of Food, Vol.III. Lind B.M., Baird-Parker T.C., dan Gould G.W., editor. Aspen Publisher, Maryland. Edwards, K., J. Logan, and N. Sanders. 2004. Real-time PCRAn Essential Guide. Horizon Bioscience, London. Espy, M. J., J. R. Uhl, L. M. Sloan, S. P. Buckwalter, M. F. Jones, E. A. Vetter, J. D. C. Yao, N. L. Wengenack, J. E. Rosenblatt, F. R. Cockerill III, and T. F. Smith. 2006. Real-time PCR in Clinical Microbiology: Applications for Routine Laboratory Testing. Clinical Microbiology Reviews, Vol. 19 No. 1, p. 165–256. Fardiaz, S. 1992. Mikrobiologi Pangan 1. Gramedia Pustaka Utama, Jakarta Fricker, M., U. Messelha¨ußer, U. Busch, S. Scherer, and M. Ehling-Schulz. 2007. Diagnostic real-time PCR assays for the detection of emetic Bacillus cereus strains in foods and recent food-borne outbreaks. Applied And Environmental Microbiology, Vol. 73, No. 6. Mar. 2007, P. 1892–1898. Gast, R.K. 2003. Salmonella Infections. Di dalam: Diseases of Poultry. Saif, Y.M., Barness H.J., Fadly, A.M., Glisson, F.R., McDougald, L.R., dan Swayne, D., editor. Blackwell Publishing, Iowa. Hansen, B. M and N. B. Hendriksen. 2001. Detection of Enterotoxic Bacillus cereus and Bacillus thuringiensis Strains by PCR Analysis. Applied and Environmental Microbiology, Vol. 67, No. 1, p. 185–189. Hein, I., A. Lehner, P. Rieck, K. Klein, E. Brandl, and M. Wagner. 2001. Comparison of Different Approaches To Quantify Staphylococcus aureus Cells by Real-time Quantitative PCR and Application of This Technique for Examination of Cheese. Applied and Environmental Microbiology, Vol. 67, No. 7, p. 3122–3126. 71 Jay, J. M. 1996. Staphylococcal Gastroenteritis. In Modern Food Microbiology. 5th. 429-450. Jayarao, B. M. and D. R. Henning. 2001. Prevalence of foodborne pathogens in bulk tank milk. J. Dairy Sci. 84:2157–2162. Lund, T., De M. L. Buyser, and P. E. Granum, 2000. A new cytotoxin from Bacillus cereus that may cause necrotic enteritis. Molecular Microbiology, 38: 254-261. Mäntynen, V. and K. Lindström. 1998. A rapid PCR-based DNA test for enterotoxic Bacillus cereus. Appl Environ Microbiol, Vol. 64, No. 5, p. 1634-1639. Mazumdar, S.D. 2008. Surface plasmon resonance (SPR) biosensor for rapid detection of Salmonella and Salmonella infections. Fachbereich Pharmazie, der Philipps-Universität Marburg, Marburg. Medici, D. D., L. Croci, E. Delibato, S. D. Pasquale, E. Filetici, and L. Toti. 1995. Evaluation of DNA Extraction Methods for Use in Combination with SYBR Green I Real-time PCR To Detect Salmonella enterica Serotype Enteritidis in Poultry. Applied and Environmental Microbiology, Vol. 69, No. 6, p. 3456–3461. Myers, C. J. and S. Sakelaris. 2004. Oligonucleotide primers for phosphotidyl inositol in Bacillus cereus. United States Patent 6713620. http://FreePatentsOnline.com [20 Februari 2009]. Naravaneni, R and K. Jamil. 2005. Rapid detection of foodborne pathogen by using molecular techniques. J. of Med Microbiol. 51-54. Patel, P. D., and D. W. Williams. 1994. Evaluation of commercial kits and instruments for the detection of foodborne bacterial patogens and toxins. In P. D. Patel (ed.), Rapid analysis techniques in food microbiology. Chapman & Hall, Glasgow, Scotland. hal 61-97. Poernomo, J.S. 2004. Variasi tipe antigen Salmonella pullorumyang ditemukan di Indonesia dan penyebaran serotipe Salmonella pada ternak. Wartazoa Vol. 14 No. 4, p. 143-159. Rusyanto, W. 2005. Prevalensi Serovar dan Galur Resisten Antibiotik Salmonella pada Rantai Produksi Udang Tambak. Tesis. IPB Press, Bogor. Sambrook, J., E.F. Fritsch, and T. Maniatis. 1989. Molecular Cloning. A Laboratory Manual. Cold Spring Harbor Laboratory Press, New York. 72 Schraft, H. and M. W. Griffiths. 1995. Specific oligonucleotide primers for detection of lecithinase-positive Bacillus spp. by PCR. Applied and Environmental Microbiology, Vol. 61, No. 1, p. 98–102. Supardi, I. dan Sukamto. 1999. Mikrobiologi dalam Pengolahan dan Keamanan Pangan. Alumni, Bandung. [USFDA] U.S Food and Drug Administration. 2001. Bacillus cereus. Bacteriological Analytic Manual January 2001, Chapter 14. FDA, United State. [USFDA] U.S Food and Drug Administration. 2007. Salmonella. Bacteriological Analytic Manual December 2007 Edition. FDA, United State. [WHO] World Health Organization. 2009. Global Salm-Surv: A Global Salmonella Surveillance and Laboratory Support Project of the World Health Organization. WHO National Salmonella and Shigella Center, Thailand. 73 Lampiran 1. Struktur organisasi Badan POM RI KEPALA BADAN PENGAWAS 1. 2. Inspektorat 3. 4. Pusat Pengujian Obat dan Makanan Nasional Deputi I Bidang Pengawasan Produk Terapetik dan Narkotika, Psikotropika dan Zat Adiktif 1. Dit. Penilaian Obat dan Produk Biologi 2. Dit. Pengawasan Distribusi Produk Terapetik & PKRT 3. Dit.Standardisasi Produk Terapetik & PKRT 4. Dit. Pengawasan Produksi Produk Terapetik & PKRT 5. Dit. Pengawasan Narkotika, Psikotropika dan Zat Adiktif (NAPZA) Sekretariat Utama Biro Perencanaan dan Keuangan Biro Kerjasama Luar Negeri Biro Hukum dan Humas Biro Umum Pusat Penyidikan Obat dan Makanan Pusat Riset Obat dan Makanan Pusat Informasi Obat dan Makanan Deputi II Bidang Pengawasan Obat Tradisional, Kosmetik, dan Produk Komplemen Deputi III Bidang Pengawasan Keamanan Pangan dan Bahan Berbahaya 1. Dit. Penilaian Obat Tradisional, Suplemen Makanan dan Kosmetik 2. Dit. Standardisasi Obat Tradisional, Kosmetik dan Produk Komplemen 3. Dit. Inspeksi dan Sertifikasi Obat Tradisional, Kosmetik, dan Produk Komplemen 4. Dit. Obat Asli Indonesia 1. Dit. Penilaian Keamanan Pangan 2. Dit. Standardisasi Produk Pangan 3. Dit. Inspeksi dan Sertifikasi Produk Pangan 4. Dit. Surveilan dan Penyuluhan Keamanan Pangan 5. Dit. Pengawasan Produk dan Bahan Berbahaya UNIT PELAKSANA TEKNIS BPOM 74 Lampiran 2. Prosedur pembuatan media (Atlas, 2006) TSB (Triptone Soy Broth) 75 Lampiran 3. Prosedur pembuatan media (Atlas, 2006) MYPA (Mannitol Egg Yolk Polymyxin Agar) (BAM M95) 76 Lampiran 4. Persiapan kultur bakteri (PROM Biotech, 2008) a. Bakteri uji b. Bakteri kontrol 77 Lampiran 5. Data jumlah koloni dan konsentrasi B. cereus Kultur murni B. cereus (KM) pada medium MYPA Cawan 10-6 10-7 10-8 CFU/ml 1 88 7 1 8.8 x 108 2 87 5 0 78 Lampiran 6. Isolasi DNA metode pendidihan (Hein et al., 2001 dengan modifikasi) 79 Lampiran 7. Isolasi DNA metode ekstraksi dengan fenol:kloroform (Sambrook et al., 1989 dengan modifikasi) 80 Lampiran 8. Isolasi DNA sesuai dengan panduan kit komersial 81 Lampiran 9. Penetapan Limit of Detection (LOD) Mikroba (PROM Biotech, 2008) 82 Lampiran 10. Uji motilitas Salmonella spp. Uji motilitas Visualisasi Motilitas Positif Motil Negatif Tidak motil 83