24 BAB 4 HASIL PERCOBAAN DAN PEMBAHASAN Pada
advertisement
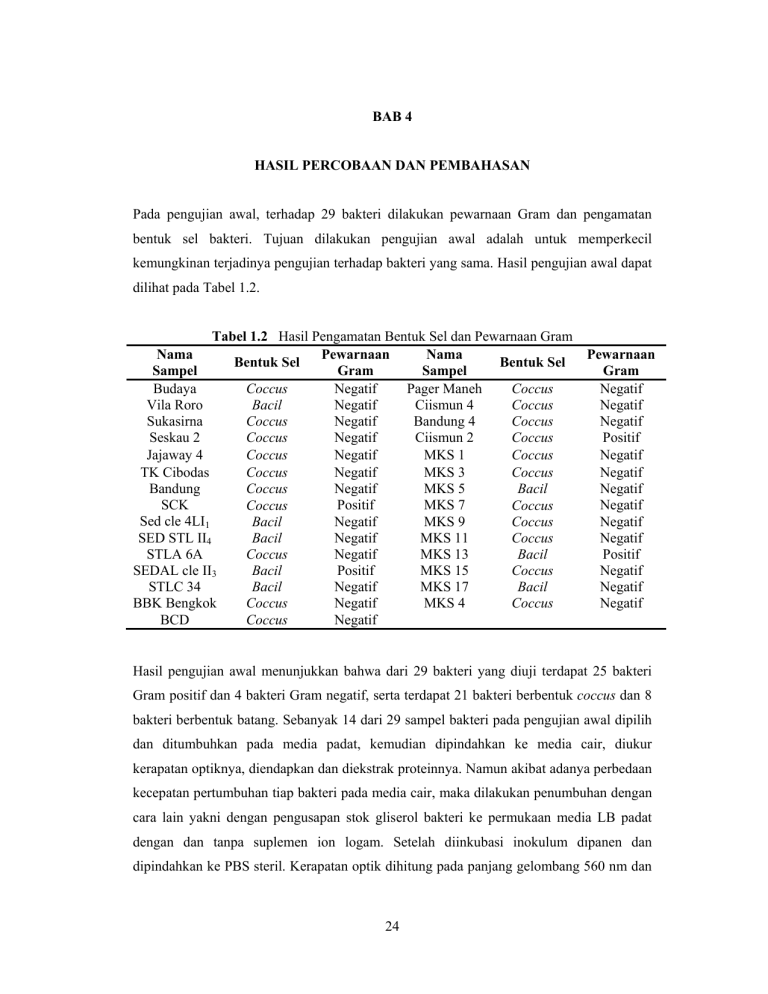
BAB 4 HASIL PERCOBAAN DAN PEMBAHASAN Pada pengujian awal, terhadap 29 bakteri dilakukan pewarnaan Gram dan pengamatan bentuk sel bakteri. Tujuan dilakukan pengujian awal adalah untuk memperkecil kemungkinan terjadinya pengujian terhadap bakteri yang sama. Hasil pengujian awal dapat dilihat pada Tabel 1.2. Tabel 1.2 Hasil Pengamatan Bentuk Sel dan Pewarnaan Gram Pewarnaan Nama Nama Bentuk Sel Bentuk Sel Gram Sampel Sampel Budaya Negatif Pager Maneh Coccus Coccus Vila Roro Negatif Ciismun 4 Bacil Coccus Sukasirna Negatif Bandung 4 Coccus Coccus Seskau 2 Negatif Ciismun 2 Coccus Coccus Jajaway 4 Negatif MKS 1 Coccus Coccus TK Cibodas Negatif MKS 3 Coccus Coccus Bandung Coccus Negatif MKS 5 Bacil SCK Positif MKS 7 Coccus Coccus Sed cle 4LI1 Negatif MKS 9 Bacil Coccus Negatif MKS 11 SED STL II4 Bacil Coccus STLA 6A Negatif MKS 13 Coccus Bacil SEDAL cle II3 Positif MKS 15 Bacil Coccus STLC 34 Negatif MKS 17 Bacil Bacil BBK Bengkok Negatif MKS 4 Coccus Coccus BCD Negatif Coccus Pewarnaan Gram Negatif Negatif Negatif Positif Negatif Negatif Negatif Negatif Negatif Negatif Positif Negatif Negatif Negatif Hasil pengujian awal menunjukkan bahwa dari 29 bakteri yang diuji terdapat 25 bakteri Gram positif dan 4 bakteri Gram negatif, serta terdapat 21 bakteri berbentuk coccus dan 8 bakteri berbentuk batang. Sebanyak 14 dari 29 sampel bakteri pada pengujian awal dipilih dan ditumbuhkan pada media padat, kemudian dipindahkan ke media cair, diukur kerapatan optiknya, diendapkan dan diekstrak proteinnya. Namun akibat adanya perbedaan kecepatan pertumbuhan tiap bakteri pada media cair, maka dilakukan penumbuhan dengan cara lain yakni dengan pengusapan stok gliserol bakteri ke permukaan media LB padat dengan dan tanpa suplemen ion logam. Setelah diinkubasi inokulum dipanen dan dipindahkan ke PBS steril. Kerapatan optik dihitung pada panjang gelombang 560 nm dan 24 25 hasil yang diharapkan adalah 0,4 sampai 0,6. Data pertumbuhan bakteri dapat dilihat pada Tabel 1.3. Tabel 1.3 Pertumbuhan Bakteri dan Kerapatan Optik Kerapatan Media Padat Media Cair Nama Sampel Optik Bakteri NL L NA NL L PBS NL L 0,54 0,42 Sukasirna 0,55 0,54 Seskau 2 0,58 0,44 TK Cibodas 0,54 0,58 Bandung SCK 0,46 0,52 0,48 SEDAL cle II3 BBK Bengkok 0,48 0,58 0,54 0,55 BCD Ciismun 4 0,51 0,54 Ciismun 2 0,48 0,54 MKS 1 0,42 0,41 0,51 0,49 MKS 11 0,53 MKS 13 Keterangan : Tanda ( ) menandakan media dimana bakteri ditumbuhkan. NL = media padat/cair LB tanpa ion logam, L = media padat/cair dengan logam, NA = pertumbuhan pada media padat NA, sedangkan PBS = pemindahan inokulum ke dapar PBS. Pada penelitian ini diperoleh 13 bakteri yang berhasil ditumbuhkan dan menghasilkan nilai kerapatan optik antara 0,4 hingga 0,6. Rentang kerapatan optik antara 0,4 hingga 0,6 menjadi penyetaraan kasar terhadap jumlah bakteri yang terkandung sebelum dilakukan ekstraksi protein total dan perhitungan aktivitas SOD total. Setelah ekstrak protein total didapatkan melalui sonikasi, sampel protein dipekatkan hingga setengah volume awal dan diuji aktivitasnya dengan metode spektrofotometri. Nilai absorbansi antara sampel protein dan pembanding serta persen inhibisi reduksi sampel dapat dilihat pada Tabel 1.4. Semakin tinggi kandungan SOD total dalam ekstrak protein total maka nilai absorbansinya akan semakin kecil karena terjadi pencegahan pembentukan warna biru dari NBT oleh SOD total yang berada dalam ekstrak protein total secara bermakna. Semakin kecilnya nilai persen inhibisi reduksi menunjukkan bahwa terdapat perbedaan yang besar antara ekstrak protein yang diuji dan pembanding, yang berarti terdapat aktivitas SOD yang cukup bermakna dibandingkan dengan NBT pada 26 pembanding. Dari hasil perhitungan persen inhibisi reduksi, terdapat 6 sampel protein yang memiliki aktivitas SOD tinggi yakni sampel SEDAL cle II3, Seskau 2, TK Cibodas, MKS 11, MKS 13 dan Ciismun 4. Tabel 1.4 Persen Inhibisi Reduksi A sampel A pembanding % Inhibisi reduksi Nama Sampel Protein NL L NL L NL L Sukasirna Seskau 2 TK Cibodas 0,22 0,12 0,10 0,28 0,13 0,15 0,34 0,33 0,27 0,32 0,31 0,28 65,16 34,66* 35,75* 86,70 41,36 52,31 Bandung 0,17 0,13 0,33 0,25 52,44 50,22 SCK 0,16 0,17 0,34 0,32 47,84 53,04 SEDAL cle II3 0,08 - 0,30 - 25,15* - BBK Bengkok 0,73 0,69 0,77 0,77 95,30 89,48 BCD 0,12 0,13 0,28 0,27 42,74 48,23 Ciismun 4 0,17 0,12 0,27 0,31 65,11 38,67* Ciismun 2 0,13 0,11 0,30 0,27 43,59 40,28 MKS 1 0,26 0,29 0,32 0,30 81,78 95,46 MKS 11 0,12 0,21 0,32 0,30 36,31* 70,76 MKS 13 0,11 - 0,28 - 37,50* - Keterangan : Tanda NL dan L menunjukkan asal penumbuhan bakteri, dengan atau tanpa logam. Tanda (*) menunjukkan nilai – nilai persen inhibisi reduksi terendah. Tanda (-) menunjukkan pengukuran tidak dilakukan karena ekstrak protein gagal diperoleh. Selain metode pengujian aktivitas SOD total dengan perhitungan persen inhibisi reduksi, dilakukan pengujian lain yakni zimografi. Zimografi yang dilakukan adalah zimografi nondenaturing sehingga ukuran protein tidak dapat diketahui. Dari hasil zimografi nondenaturing didapatkan pita yang jelas terlihat pada ekstrak protein SEDAL cle II3, Seskau 2, TK Cibodas, dan MKS 13, sedangkan pada ekstrak protein Ciismun 4 terlihat dua pita samar pada bagian atas gel dan tidak terlihat pita pada ekstrak protein MKS 11. Hasil dapat dilihat pada Gambar 1.6. Perbedaan metode zimografi dengan metode spektrofotometri adalah pada jumlah NBT yang digunakan sebagai pembanding serta pada proses visualisasi. Pada metode spektrofotometri, jumlah NBT yang digunakan sebagai pembanding sama dengan jumlah sampel protein total, selain itu aktivitas juga diketahui dengan perhitungan absorbansi. 27 Pada metode zimografi, penambahan NBT dilakukan untuk mewarnai latar belakang sehingga bercak transparan yang menunjukkan aktivitas SOD terlihat. 13 4 6 26 27 18 Gambar 1.6 Hasil Zimografi Sampel Protein Total. Keterangan : 13 = SEDAL cle II3, 4 = Seskau 2, 6 = TK Cibodas, 26 = MKS 11, 27 = MKS 13, 18 = Ciismun 4. Pita transparan ditandai dengan lingkaran. Kelemahan metode zimografi adalah karena perbandingan antara NBT dan sampel protein total cukup besar, aktivitas SOD pada ekstrak protein total kurang dapat terlihat secara jelas, lain halnya dengan metode spektrofotometri yang proses identifikasi aktivitasnya dilakukan dengan bantuan spektrofotometer yang lebih sensitif. Enam bakteri dengan aktivitas SOD total tertinggi dari pengujian aktivitas secara spektrofotometri dan zimografi diisolasi DNA kromosomnya menggunakan kit pereaksi Wizard. Elektroforesis dari hasil isolasi DNA dapat dilihat pada Gambar 1.7. 13 4 6 26 27 18 Gambar 1.7 Elektroforesis Hasil Isolasi DNA. 13 = SEDAL cle II3, 4 = Seskau 2, 6 = TK Cibodas, 26 = MKS 11, 27 = MKS 13, 18 = Ciismun 4. DNA yang didapatkan dari proses isolasi DNA digunakan sebagai cetakan pada reaksi PCR gen 16S rDNA. Pita produk PCR 16S rDNA terletak pada marka 1 kilo basa (kb) dan 2 kb, kemudian melalui perhitungan manual dengan menggunakan regresi logaritmik antara jarak sumur ke marker dan jarak sumur ke pita produk PCR didapatkan hasil ukuran produk PCR adalah sebesar 1516 pasangan basa (pb). Hal ini sesuai dengan ukuran gen 16S rDNA yang 28 memiliki variasi rentang antara 1400 hingga 1600 pb. Ukuran gen 16S rDNA tiap bakteri berbeda – beda, namun perbedaan tersebut sangat kecil, yakni hanya sekitar beberapa pasang basa saja sehingga penggunaan gel agarosa dengan konsentrasi 1% saja tidak cukup sensitif dalam mendeteksi perbedaan tersebut. Hasil PCR 16S rDNA dapat dilihat pada Gambar 1.8. Penentuan besar gen 16S rDNA dan identifikasi spesies dari gen 16S rDNA dilakukan dengan penentuan urutan nukleotida. Gambar 1.8 Elektroforesis Produk PCR 16S rDNA. Tanda 1 kb dan 2 kb menunjukkan marka 1 kilo basa dan 2 kilo basa. Tanda (-) menunjukkan kontrol negatif. 13 = SEDAL cle II3, 4 = Seskau 2, 6 = TK Cibodas, 26 = MKS 11, 27 = MKS 13, 18 = Ciismun 4. Setelah PCR 16S rDNA berhasil dilakukan, dilakukan penentuan urutan nukleotida dari gen 16S rDNA tersebut. Kelemahan dari alat penentu urutan nukleotida adalah kemampuan pembacaan nukleotida per satu reaksi dengan satu primer adalah hanya sepanjang 700 hingga 800 pb. Untuk mengatasi masalah tersebut, maka proses PCR dalam penentuan urutan nukleotida dilakukan dua arah dengan menggunakan kedua primer BactF1 dan UniB1. Dilakukan beberapa cara dalam memeriksa kualitas hasil penentuan urutan nukleotida, yakni dengan penentuan daerah tumpang tindih, penentuan rantai sense/antisense, dan pemeriksaan beberapa parameter pada program BLAST. Cara pertama, yakni penentuan daerah tumpang tindih hasil dari penentuan urutan nukleotida menggunakan kedua primer dilakukan dengan program Sequencher. Ukuran gen 16S rDNA juga dapat diketahui 29 dengan cara ini. Salah satu hasil analisa penentuan urutan nukleotida dengan cara ini dapat dilihat pada Gambar 1.9. Pada Gambar 1.9 dapat dilihat bahwa pada hasil penentuan urutan nukleotida gen 16S rDNA sampel MKS 13 terdapat daerah tumpang tindih pada nukleotida bernomor 547 hingga 946, dan panjang gen 16S rDNA utuh adalah 1466 pb. Selain itu sampel yang menghasilkan daerah tumpang tindih pada analisa ini adalah sampel Seskau 2 dan TK Cibodas pada nukleotida 539 hingga 919 dan 536 hingga 910, dengan panjang gen 16S rDNA utuh adalah 1454 dan 1448 pb. Hasil sekuensing produk PCR 16S rDNA dari sampel – sampel lain tidak memberikan daerah tumpang tindih, karena kualitas hasil penentuan nukleotida yang kurang baik pada bagian akhir urutan nukleotida. Gambar 1.9 Hasil Penentuan Daerah Tumpang Tindih dengan Program Sequencher. Daerah tumpang tindih ditandai dengan lingkaran. Cara kedua dalam menganalisis hasil penentuan urutan nukleotida yakni penentuan polaritas rantai dari hasil kedua primer, yang diketahui dengan menggunakan program BLAST dari NCBI. Hasil penentuan urutan nukleotida menggunakan primer forward menghasilkan rantai plus/plus yang berarti rantai yang dihasilkan adalah rantai sense, sedangkan hasil penentuan urutan nukleotida menggunakan primer reverse menghasilkan rantai plus/minus yang berarti rantai yang dihasilkan adalah rantai antisense dari rantai DNA. Hasil penentuan polaritas rantai dapat dilihat pada Tabel 1.5. Cara ketiga dalam menganalisis hasil penentuan urutan nukleotida adalah pemeriksaan parameter BLAST. Parameter BLAST yang diperiksa adalah query coverage dan maximum identity. Melalui parameter query coverage dapat diketahui berapa persen dari 30 total panjang urutan nukleotida sampel yang cukup baik untuk disejajarkan dengan urutan nukleotida yang dimiliki oleh bank data NCBI, sedangkan dengan parameter maximum identity dapat diketahui persen kesamaan antara urutan nukleotida sampel yang disejajarkan dengan urutan nukleotida bank data. Tabel 1.6 menunjukkan hasil analisis melalui parameter BLAST. Tabel 1.5 Penentuan Polaritas Rantai Sense/Antisense Nama Sampel Polaritas Rantai Hasil Polaritas Rantai Hasil Gen 16S rDNA Primer BactF1 Primer UniB1 SEDAL cle II3 plus/plus plus/minus Seskau 2 plus/plus plus/minus TK Cibodas plus/plus plus/minus MKS 11 plus/plus plus/minus MKS 13 plus/plus plus/minus Ciismun 4 plus/plus plus/minus Tabel 1.6 Analisis Parameter BLAST Query Maximum Identitas pada Bank Coverage (%) Identity (%) Data NCBI SEDAL cle II3F 91 92 Bacillus subtilis SEDAL cle II3R 97 93 Bacillus subtilis Seskau 2F 97 98 Enterobacter cloacae Seskau 2R 92 97 Enterobacter cloacae TK CibodasF 98 99 Shigella boydii TK CibodasR 98 99 Shigella boydii MKS 11F 91 78 Escherichia coli MKS 11R 89 93 Escherichia coli MKS 13F 97 99 Bacillus subtilis MKS 13R 97 97 Bacillus subtilis Ciismun 4 F 90 93 Escherichia coli Ciismun 4R 93 87 Escherichia coli Keterangan : Tanda F atau R di sebelah nomor sampel menandakan pengujian dilakukan dari hasil pengurutan nukleotida menggunakan primer forward BactF1 atau primer reverse UniB1 Nama Sampel Selanjutnya dilakukan pencocokan data morfologi antara data pengujian dan data dari pustaka. Data yang dicocokkan adalah data pewarnaan Gram, data pengamatan bentuk sel bakteri, dan data bentuk koloni bakteri. Data bentuk koloni bakteri dapat dilihat pada Tabel 1.7. Pewarnaan Gram yang sebelumnya dilakukan dalam pengujian awal menunjukkan hasil yang sama dengan data dari pustaka. Namun terdapat perbedaan dari pengamatan bentuk sel bakteri, dinyatakan dalam data pustaka bahwa keseluruhan bakteri memiliki bentuk batang namun pada pengamatan hanya sampel SEDAL cle II3 dan MKS 13 yang 31 teridentifikasi sebagai B. subtilis yang berbentuk batang. Untuk mengkonfirmasi kebenaran bentuk sel dan data morfologi maupun data fisiologi lainnya, disarankan untuk dilakukan identifikasi spesies dengan metode biokimia. Nama Sampel SEDAL cle II3 Seskau 2 Tabel 1.7 Perbandingan Gambar Koloni Hasil Pengujian dan Gambar Koloni dari Pustaka Gambar Koloni Hasil Gambar Koloni dari Identitas Pengujian Pustaka Bacillus subtilis Enterobacter Koloni bulat kuning cloacae transparan TK Cibodas Shigella boydii MKS 11 Escherichia coli MKS 13 Bacillus subtilis Ciismun 4 Escherichia coli