Studi Asosiasi Lintas Genom: pendekatan molekuler baru untuk
advertisement

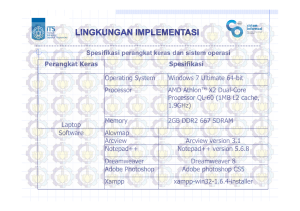
26 Studi Asosiasi Lintas Genom: pendekatan molekuler baru untuk mempersingkat waktu pemuliaan tanaman Pemuliaan tanaman identik dengan pekerjaan yang rumit dan membutuhkan waktu lama apabila dilakukan dengan cara konvensional. Studi Asosiasi Lintas Genom (GWAS) menjadikan proses pemuliaan tanaman lebih terarah, efektif dan efisien dengan mempelajari hubungan keterkaitan antara karakter yang tampak (fenotipe) dan gen-gen di dalamnya (genotipe). Fenotipe yang diinginkan oleh pemulia dapat disasar dengan akurat setelah diketahui basa nukleotida penyusun gen yang terlibat dalam ekspresi karakter tersebut. Pada awalnya, Studi Asosiasi Lintas Genom atau dikenal dengan sebutan Genome-Wide Association Study (GWAS) digunakan untuk mempelajari genom manusia yang terkait dengan gen-gen fungsional penyebab penyakit pada manusia [1]. Para peneliti berusaha untuk mencari penyebab genetis beberapa penyakit kronis seperti kanker, asma, serangan jantung, parkinson, sklerosis, dan diabetes [2-6]. Keberhasilan studi GWAS pertama kali dilaporkan pada tahun 2005, ketika kelompok peneliti menemukan dua SNP (Single Nucleotide Polymorphism) yang berbeda dibandingkan dengan kontrol pada pasien yang mengalami penuaan dini [7]. Hingga tahun 2017, lebih dari 3000 pasien telah diuji melalui pendekatan ini sehingga lebih dari 1800 penyakit berhasil diketahui penyebab dan mekanisme infeksinya [1,8]. Saat ini, Studi Asosiasi Lintas Genom sudah banyak juga digunakan untuk mempelajari penyebab genetik penyakit pada tanaman maupun karakter-karakter fenotipik lainnya yang berguna untuk program pemuliaan tanaman. Ilustrasi Studi Asosiasi Lintas Genom (GWAS) sebagai uji klinis empiris untuk mengidentifikasi dan mengkarakterisasi penyebab penyakit pada manusia [9] www.iribb.org | April 2017 | 5(1), 26-29 Irfan Martiansyah & Riza Arief Putranto - Peneliti PPBBI 27 Pemuliaan tanaman semakin berkembang seiring sejak revolusi hijau yang terus melangkah maju hingga sekarang. Pemanfaatan penanda molekuler dalam pemuliaan berpotensi mempercepat dan meningkatkan presisi dari proses seleksi tanaman. Teknik ini antara lain diaplikasikan untuk menganalisis keterpautan antara gen dengan sifat penting pada populasi segregasi sehingga gen target dapat terpetakan dalam genom tanaman [10,11]. Studi Asosiasi Lintas Genom merupakan pengembangan dari teknologi pemetaan gen dengan menggunakan data genotipe dan fenotipe untuk analisisnya. Berbeda dengan studi pemetaan gen, GWAS dapat dicobakan pada beberapa aksesi plasma nutfah tanpa memerlukan populasi segregasi hasil silangan antara dua tetua. Melalui teknologi GWAS, keragaman plasma nutfah dapat diungkap lebih detil sebagai sumber genetik dalam perbaikan varietas [8,12]. Umumnya, Studi Asosiasi Lintas Genom dilakukan pada populasi tetap spesies tanaman tertentu yang tersebar dalam beberapa fenotipe karakter dan menghasilkan peta populasi spesifik untuk karakter tersebut [13]. Ilustrasi Studi Asosiasi Lintas Genom (GWAS) untuk pemuliaan tanaman padi (http://ricewiki.big.ac.cn/index.php/Genome-Wide_Association_Studies_in_Rice) Studi Asosiasi Lintas Genom pada tanaman pertanian digunakan untuk pemetaan persilangan biparental dan pemetaan genetik multikarakter yang berkelanjutan. Lebih jauh penggunaan studi ini untuk mengungkap secara genetik morfologi tanaman, hasil produksi dan fisiologi tanaman liar maupun budidaya [14]. Hingga saat ini, GWAS telah berhasil diimplementasikan pada Oryza sativa, Zea mays, Brassica napus, Hordeum vulgare, Sorgum sp., dan Setaria sp., [14-19]. Pada padi, studi ini mendukung terciptanya varietas-varietas baru yang tahan cekaman abiotik (toleran salinitas, suhu tinggi, dan suhu rendah) [20,21] maupun biotik (resistensi terhadap nematoda akar dan ketahanan terhadap penyakit blas) [22,23] serta dapat memunculkan karakter agronomi yang diinginkan (waktu berbunga, arsitektur akar, ukuran dan bentuk bulir) [24,25]. Pada tanaman perkebunan, saat ini GWAS digunakan dalam program pemuliaan tanaman kelapa sawit untuk (1) peningkatan toleransi terhadap perubahan iklim dan (2) peningkatan www.iribb.org | April 2017 | 5(1), 26-29 Irfan Martiansyah & Riza Arief Putranto - Peneliti PPBBI 28 kandungan minyak tinggi pada mesokarp [26,27]. Selain itu studi molekuler ini berhasil diaplikasikan untuk meningkatkan karakter kuantitatif tanaman tebu dan merakit varietas tebu resisten sugarcane yellow leaf virus (SCYLV) [28,29]. Pada tanaman kakao, GWAS digunakan untuk mencari gen-gen yang terlibat dalam inkompatibilitas seksual akibat banyaknya bunga yang berguguran [30]. Namun, sejauh ini publikasi terkait penerapan GWAS belum pernah ditemukan pada pemuliaan tanaman karet. Dengan perkembangan teknologi sekuensing dan metode komputasi yang cepat, Studi Asosiasi Lintas Genom dapat menjadi salah satu pendekatan molekuler “terbaik” untuk mendeteksi variasi alami suatu tanaman dan merakit varietas-varietas baru dalam program pemuliaan tanaman masa kini. Referensi 1. Burton PR, Clayton DG, Cardon LR, Craddock N, Deloukas P, et al. (2007) Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. nature 447: 661-678. 2. Cookson MR, Bandmann O (2010) Parkinson's disease: insights from pathways. Human Molecular Genetics 19: R21-R27. 3. Garcia-Sanchez A, Isidoro-García M, Garcia-Solaesa V, Sanz C, Hernandez-Hernandez L, et al. (2015) Genome-wide association studies (GWAS) and their importance in asthma. Allergologia et immunopathologia 43: 601-608. 4. Lee J-Y, Lee B-S, Shin D-J, Woo Park K, Shin Y-A, et al. (2013) A genome-wide association study of a coronary artery disease risk variant. J Hum Genet 58: 120-126. 5. Stadler ZK, Thom P, Robson ME, Weitzel JN, Kauff ND, et al. (2010) Genome-Wide Association Studies of Cancer. Journal of Clinical Oncology 28: 4255-4267. 6. van Rheenen W, Shatunov A, Dekker AM, McLaughlin RL, Diekstra FP, et al. (2016) Genome-wide association analyses identify new risk variants and the genetic architecture of amyotrophic lateral sclerosis. Nat Genet 48: 1043-1048. 7. Klein RJ, Zeiss C, Chew EY, Tsai J-Y, Sackler RS, et al. (2005) Complement Factor H Polymorphism in Age-Related Macular Degeneration. Science (New York, NY) 308: 385-389. 8. Bush WS, Moore JH (2012) Genome-wide association studies. PLoS Comput Biol 8: e1002822. 9. Ko DC, Urban TJ (2013) Understanding human variation in infectious disease susceptibility through clinical and cellular GWAS. PLoS Pathog 9: e1003424. 10. Crossa J, Pérez P, Hickey J, Burgueño J, Ornella L, et al. (2014) Genomic prediction in CIMMYT maize and wheat breeding programs. Heredity 112: 48-60. 11. Heffner EL, Lorenz AJ, Jannink J-L, Sorrells ME (2010) Plant breeding with genomic selection: gain per unit time and cost. Crop science 50: 1681-1690. 12. Utami DW (2014) Pemanfaatan Asosiasi Lintas Genom (Studi asosiasi Genom) dalam Pemuliaan Tanaman. Jurnal Penelitian dan Pengembangan Pertanian 33: 141-148. 13. Borém A, Fritsche-Neto R (2014) Biotechnology and Plant Breeding: Applications and Approaches for Developing Improved Cultivars: Elsevier. 14. Bian Y, Holland J (2017) Enhancing genomic prediction with genome-wide association studies in multiparental maize populations. Heredity. www.iribb.org | April 2017 | 5(1), 26-29 Irfan Martiansyah & Riza Arief Putranto - Peneliti PPBBI 29 15. Visioni A, Tondelli A, Francia E, Pswarayi A, Malosetti M, et al. (2013) Genome-wide association mapping of frost tolerance in barley (Hordeum vulgare L.). BMC Genomics 14: 424. 16. Sun C, Wang B, Wang X, Hu K, Li K, et al. (2016) Genome-Wide Association Study Dissecting the Genetic Architecture Underlying the Branch Angle Trait in Rapeseed (Brassica napus L.). Scientific Reports 6. 17. Spindel J, Begum H, Akdemir D, Collard B, Redoña E, et al. (2016) Genome-wide prediction models that incorporate de novo GWAS are a powerful new tool for tropical rice improvement. Heredity. 18. Morris GP, Ramu P, Deshpande SP, Hash CT, Shah T, et al. (2013) Population genomic and genome-wide association studies of agroclimatic traits in sorghum. Proceedings of the National Academy of Sciences 110: 453-458. 19. Jia G, Huang X, Zhi H, Zhao Y, Zhao Q, et al. (2013) A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (Setaria italica). Nature genetics 45: 957-961. 20. Kumar V, Singh A, Mithra SA, Krishnamurthy S, Parida SK, et al. (2015) Genome-wide association mapping of salinity tolerance in rice (Oryza sativa). DNA Research: dsu046. 21. Lv Y, Guo Z, Li X, Ye H, Li X, et al. (2016) New insights into the genetic basis of natural chilling and cold shock tolerance in rice by genome‐wide association analysis. Plant, cell & environment 39: 556-570. 22. Dimkpa SO, Lahari Z, Shrestha R, Douglas A, Gheysen G, et al. (2015) A genome-wide association study of a global rice panel reveals resistance in Oryza sativa to root-knot nematodes. Journal of experimental botany: erv470. 23. Wang C, Yang Y, Yuan X, Xu Q, Feng Y, et al. (2014) Genome-wide association study of blast resistance in indica rice. BMC plant biology 14: 311. 24. Huang X, Wei X, Sang T, Zhao Q, Feng Q, et al. (2010) Genome-wide association studies of 14 agronomic traits in rice landraces. Nat Genet 42: 961-967. 25. Huang X, Zhao Y, Wei X, Li C, Wang A, et al. (2012) Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm. Nature genetics 44: 32-39. 26. Teh C-K, Ong A-L, Kwong Q-B, Apparow S, Chew F-T, et al. (2016) Genome-wide association study identifies three key loci for high mesocarp oil content in perennial crop oil palm. Scientific Reports 6. 27. Rival A (2017) Breeding the oil palm (Elaeis guineensis Jacq.) for climate change. OCL 24: D107. 28. Débibakas S, Rocher S, Garsmeur O, Toubi L, Roques D, et al. (2014) Prospecting sugarcane resistance to sugarcane yellow leaf virus by genome-wide association. Theoretical and Applied Genetics 127: 1719-1732. 29. Racedo J, Gutiérrez L, Perera MF, Ostengo S, Pardo EM, et al. (2016) Genome-wide association mapping of quantitative traits in a breeding population of sugarcane. BMC plant biology 16: 142. 30. da Silva MR, Clément D, Gramacho KP, Monteiro WR, Argout X, et al. (2016) Genomewide association mapping of sexual incompatibility genes in cacao (Theobroma cacao L.). Tree Genetics & Genomes 12: 1-13. www.iribb.org | April 2017 | 5(1), 26-29 Irfan Martiansyah & Riza Arief Putranto - Peneliti PPBBI